1.1 设计 基因信息学实验。
1.2 时间及地点 实验于2023年1-8月在重庆市妇幼保健院人类胚胎工程重庆市重点实验室完成。
1.3 对象
1.3.1 临床收集颗粒细胞 2023年1-2月期间在重庆市妇幼保健院进行辅助生殖治疗过程中,收集患者取卵时所获得的废弃卵丘颗粒细胞。所选患者为20-45周岁女性,低龄组患者年龄为25-35周岁,高龄组患者年龄为38-45周岁,每组收集10例患者颗粒细胞进行后续实验。
纳入标准:①收集前2个月内无任何疾病史、无激素使用史,且相关体检指标正常;②不孕原因为输卵管疾病或男性因素,无卵巢功能不全、多囊卵巢综合征、子宫内膜异位症等疾病;③无生殖道畸形、家族遗传病、血栓疾病史、染色体异常疾病;④无慢性感染性疾病、自身免疫性疾病、肝肾功能不全、结核史及肿瘤史。
排除标准:患有卵巢早衰及各类生殖内分泌疾病的女性。
所有受试者均已签署《科研捐赠知情同意书》,自愿捐赠辅助生殖治疗过程中取卵周期废弃卵泡液内的颗粒细胞并同意用于此课题研究。实验方案经重庆市妇幼保健院伦理委员会审查批准,审批号为2018-RGI-03。
1.3.2 主要试剂 M2培养液(货号M716)购自Sigma公司;RNA-seq试剂盒(货号N712-01)购自诺唯赞公司;DNA纯化磁珠(货号N411)购自诺唯赞公司;引物由上海生工生物工程技术服务有限公司合成;RNA提取试剂盒(货号KIT0214)购自Thermo Fisher公司;反转录试剂盒(货号RR036A)购自Takara公司。
1.4 实验方法
1.4.1 颗粒细胞获取 所有实验对象在黄体中期时注射促性腺激素释放激素(GnRH)拮抗剂,通过超声监测卵泡生长情况,当有3个及以上卵泡平均直径大于1.8 cm时,注射8 000-10 000 U人体绒膜促性腺激素,36 h后对患者进行局部麻醉,在阴道超声引导下采用17号双腔吸引针进行取卵。首先收集卵丘卵母细胞复合体用于体外受精,剩余卵泡液则用于颗粒细胞的收集。将卵泡液250×g离心10 min后,在沉淀中加入100 μL透明质酸酶消化颗粒细胞块,随后加入Focill-Paque,通过密度梯度离心法分离卵泡液中的颗粒细胞及血细胞,吸取颗粒细胞层并将其转移至1.5 mL EP管中,PBS清洗后加入Trizol保存于
-80 ℃冰箱中待用。
1.4.2 总RNA提取 使用RNA提取试剂盒Arcturus PicoPure RNA isolation Kit,具体操作如下:取出冻存的颗粒细胞;先加入350 μL Lysis Buffer,用移液枪轻轻吹匀;再加入350 μL体积分数为70%乙醇,再次用移液枪轻轻吹匀;将混合后样品转移至吸附柱中,12 000 r/min离心20 s,弃废液;在吸附柱中加入80 μL DNA酶,避光消化15 min;加入350 μL Wash Buffer 1,12 000 r/min离心20 s,弃废液;随后加入500 μL Wash Buffer 2,12 000 r/min 离心20 s,弃废液,再加入500 μL Wash Buffer 2,12 000 r/min 离心20 s,弃废液;13 000 r/min空离2 min,更换收集管;在吸附柱中加入20 μL DEPC水洗脱。
1.4.3 总RNA反转录 使用反转录试剂盒 PrimeScript RT Master Mix,具体操作如下:准备2×RT Master Mix,即2.0 μL 的10×RT Buffer,0.8 μL的25×dNTP Mix (100 mol/L),2.0 μL的10×RT Random Primers,1.0 μL的MultiScribe Reverse Transcriptase,4.2 μL的Nuclease-free H2O;将2×RT Master Mix与洗脱得到的RNA混合均匀,反应条件:25 ℃ 10 min,37 ℃ 2 h,85 ℃ 5 min,经反转录得到cDNA。
1.4.4 Real-time PCR实验 基因表达模式检测均采用SYBR real-time PCR,反应体系如下:1 μL模板cDNA,10 μL 2×SYBR ExTaq Mix,引物F/R (10 μmol/L)各0.5 μL (2×1 μL),8 μL dH2O。在CFX96 Real-Time System上按以下条件进行PCR 反应,95 ℃ 30 s,95 ℃ 15 s,60 ℃ 1 min (检测信号),40个循环,95 ℃ 15 s,60 ℃ 1 min (检测信号),95 ℃ 15 s。每组设置3个复孔。Real-time PCR 结果分析采用经典2-∆∆Ct法。相对基因表达归一化处理以Actin mRNA 水平为内参。基因引物序列见表1。

1.4.5 cDNA文库构建及下一代测序 使用商业化的RNA-seq试剂盒。具体操作如下:配制Sample Buffer,包含18 μL Lysis Buffer,2 μL RNase inhibitor;每个样本取1 μL Sample Buffer,1 μL细胞样品,1.5 μL Nuclease-free H2O,1 μL Oligo VN Primer,1 μL dNTP Mix充分混匀并短暂离心,置于PCR仪中72 ℃ 3 min,随后冰上孵育2 min;将上一步5.5 μL产物与2 μL 1st Strand Buffer,0.5 μL DTT,0.5 μL RNase Inhibitor,0.5 μL 5’TS Oligo Primer,1 μL Sc Reverse Transcriptase充分混匀并短暂离心,置于PCR仪中42 ℃ 90 min,70 ℃ 15 min,第一链cDNA合成完成;将10 μL产物与2 μL Nuclease-free H2O,0.5 μL PCR Primer,12.5 μL 2 × Amplification Mix充分混匀并短暂离心,置于PCR仪中按照98 ℃ 1 min,(98 ℃ 10 s, 65 ℃ 15 s,72 ℃ 6 min)10个循环,72 ℃ 5 min,完成全长cDNA扩增。RNA-seq测序由北京诺禾致源科技股份有限公司完成。
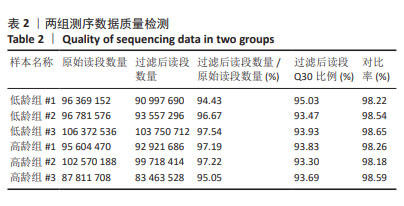
1.4.6 测序数据质控及比对定量 使用FastQC(https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)软件评估原始测序数据的质量;使用Fastp软件对原始数据进行过滤[8],删除低质量及含N(指代测序过程中,测序仪无法准确判断该位点的碱基类型,则通常会用N来代替这个位置可能的碱基)高于5%的不满足分析标准的测序读段,得到可用于后续比对及分析的高质量读段;选用人类基因组的hg38(UCSC)作为参考基因组,利用hisat2工具将高质量的纯净读段与参考基因组进行比对[9],比对过程使用hisat2默认参数。利用Featurecount工具对比对结果进行定量[10],对于蛋白质编码基因,注释文件使用的是下载自Ensembl的gtf文件。对于LncRNA,注释文件使用的是下载自LNCipedia(version 5.2)的gtf文件。
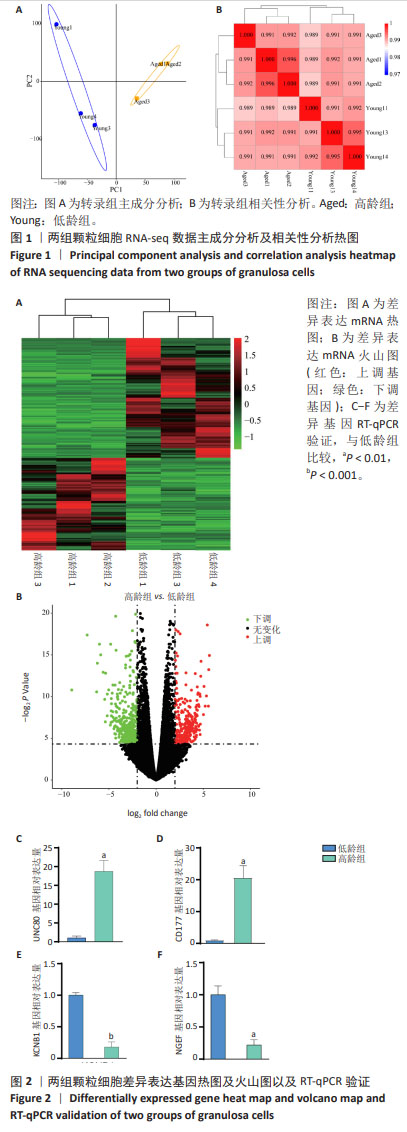
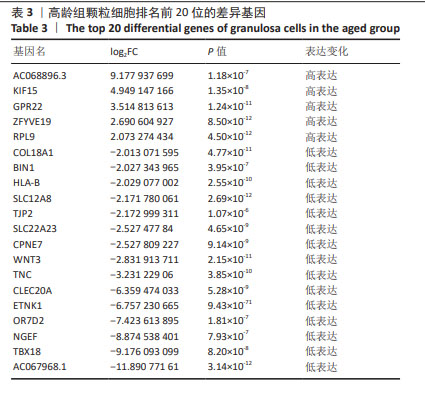
1.4.7 差异表达分析 使用R语言的edgeR软件包实现对高龄组和低龄组颗粒细胞的mRNA以及LncRNA的差异表达分析[11]。在两组中,以差异倍数|log2(fold change,FC)|≥1和P < 0.05为标准来筛选组间显著差异表达的LncRNA(DE-LncRNA)以及mRNA(DE-mRNA)。
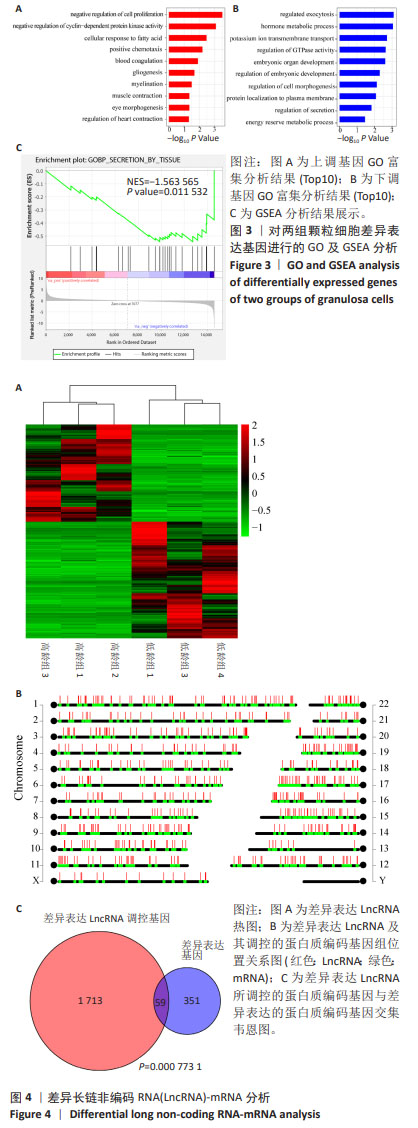
1.4.8 DE-LncRNA与mRNA共表达网络构建 基因共表达网络的构建可直观显示LncRNA和mRNA之间的关联性,其基本方法是利用bedtools工具提取差异LncRNA基因组上小于10 kb位置的mRNA,构建出LncRNA-mRNA相互作用关系。将DE-LncRNA 附近10 kb范围内的mRNA与差异表达的mRNA取交集,并通过R中的Fisher检验工具,计算其显著性。
1.4.9 差异基因功能富集分析 使用metasacpe网页版富集分析工具[12],对差异表达的mRNA进行GO富集分析,保留P < 0.05的结果,根据结果显示排名前10的GO富集结果。输入标准化后的表达值,对样本进行GSEA分析[13],GSEA本地版工具下载自https://www.gsea-msigdb.org/gsea/index.jsp,保留P < 0.05的结果。
1.5 主要观察指标 收集颗粒细胞进行RNA-seq后,对测序数据进行差异表达基因、GO、GSEA等生信分析,观察高龄组相较于年轻组变化,分析年龄因素对颗粒细胞产生的影响。
1.6 统计学分析 使用SPSS 23.0软件进行统计分析。数据以x±s表示,t检验用于评估组间差异。利用GraphPad Prism 8.0软件绘图。文章统计学方法已经通过东北农业大学生命科学学院生物统计学专家审核。
中国组织工程研究杂志出版内容重点:干细胞;骨髓干细胞;造血干细胞;脂肪干细胞;肿瘤干细胞;胚胎干细胞;脐带脐血干细胞;干细胞诱导;干细胞分化;组织工程