Chinese Journal of Tissue Engineering Research ›› 2024, Vol. 28 ›› Issue (20): 3252-2358.doi: 10.12307/2024.349
Previous Articles Next Articles
Regulation of N6-methyladenosine on non-coding RNAs in pathological cardiac remodeling
Yin Gonghua, Xu Ruoyao, Zhang Lijuan, Zhang Yifan, Qi Jie, Zhang Jun
- Institute of Physical Education, Shanghai Normal University, Shanghai 200234, China
-
Received:2023-05-09Accepted:2023-06-19Online:2024-07-18Published:2023-09-11 -
Contact:Qi Jie, PhD, Associate professor, Institute of Physical Education, Shanghai Normal University, Shanghai 200234, China Zhang Jun, PhD, Professor, Institute of Physical Education, Shanghai Normal University, Shanghai 200234, China -
About author:Yin Gonghua, Master candidate, Institute of Physical Education, Shanghai Normal University, Shanghai 200234, China -
Supported by:the National Natural Science Foundation of China, No. 32071170 (to ZJ)
CLC Number:
Cite this article
Yin Gonghua, Xu Ruoyao, Zhang Lijuan, Zhang Yifan, Qi Jie, Zhang Jun. Regulation of N6-methyladenosine on non-coding RNAs in pathological cardiac remodeling[J]. Chinese Journal of Tissue Engineering Research, 2024, 28(20): 3252-2358.
share this article
Add to citation manager EndNote|Reference Manager|ProCite|BibTeX|RefWorks
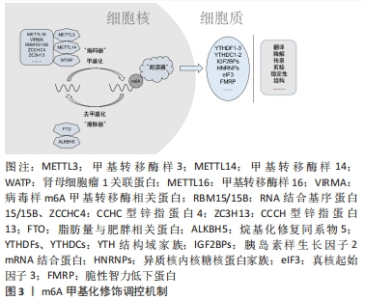
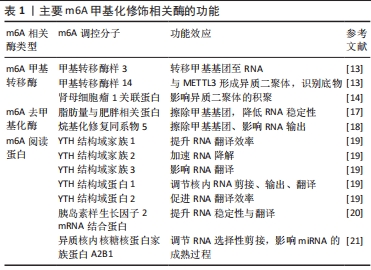
2.1 m6A甲基化修饰的生物学特征及其调控分子 2.1.1 核m6A甲基化修饰的生物学特性 m6A指腺嘌呤第6位氮原子上发生甲基化修饰,自从20世纪70年代首次在肝癌细胞中发现以来,m6A甲基化修饰已逐渐成为成为生命科学的研究热点[9]。m6A甲基化修饰被学界认为是真核生物RNA中最丰富的化学修饰,其修饰位点主要在蛋白质编码区、3’非翻译区、近终止密码子和外显子附近富集,并影响RNA的剪接、输出、翻译、降解等过程。在肥大与衰竭心脏中,m6A水平发生变化且不依赖转录翻译调控机制[10]。m6A甲基化主要由3种分子进行调控:m6A甲基转移酶、m6A去甲基化酶和m6A阅读蛋白,具体发生如下过程:首先由m6A甲基转移酶将甲基基团写入RNA中;m6A去甲基化酶可将RNA上的甲基基团擦除;最后由m6A阅读蛋白识别发生m6A修饰的碱基位点,以此发挥调节RNA的生物学功能(图3)。"
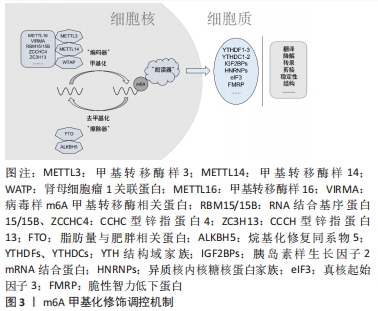
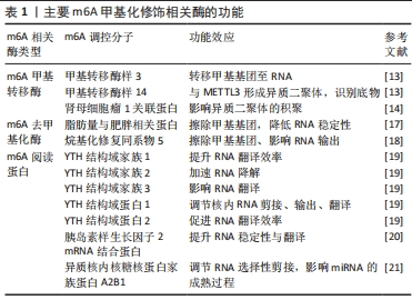
2.1.2 m6A甲基化修饰调控分子 (1)m6A甲基转移酶:主要由甲基转移酶样3、甲基转移酶样14、肾母细胞瘤1关联蛋白等组成复合物来催化甲基基团至腺嘌呤第6位氮原子上,这一类酶称为“编码器”[11]。在甲基转移酶复合物中,甲基转移酶样3首先检测出[12],通过与S-腺苷甲硫氨酸结合来发挥其甲基转移酶活性。甲基转移酶样14在底物识别中发挥重要作用[13],二者可以形成稳定的异质二聚体并增强m6A的催化。肾母细胞瘤1关联蛋白虽然不具有甲基转移酶活性,但是可以与甲基转移酶样3-甲基转移酶样14所形成的异质二聚体相互作用,并在核斑点中调节异质二聚体的积聚[14]。 此外,随着研究的进一步深入,其他甲基转移酶,例如甲基转移酶样16、病毒样m6A甲基转移酶相关蛋白、RNA结合基序蛋白15/15B、CCHC型锌指蛋白4、CCCH型锌指蛋白13等也发现于甲基转移复合物,共同发挥催化作用[15]。 (2)m6A去甲基化酶:可将已发生甲基化修饰的RNA去甲基化,所以又将这一类酶称为“擦除器”[16]。脂肪量与肥胖相关蛋白属于烷基化修复同系物蛋白家族成员,是最早被发现的m6A去甲基化酶,其主要定位于细胞核,在靶向RNA的m6A甲基化修饰残基中可有效发挥氧化去甲基化活性的作用[17]。在ALKB蛋白家族成员中,烷基化修复同系物5也在动物中检测到具有去甲基化活性[18],尽管二者都能定位于核斑点中,但是对底物的识别与功能都有所不同,其中的机制还需进一步深入讨论。 (3)m6A阅读蛋白:当RNA发生m6A甲基化修饰后,多种m6A阅读蛋白可选择性地识别已发生甲基化修饰的碱基,因此这类蛋白又称为“阅读器”,其中YTH(YT521-B homology)家族成员的研究较为充分,主要包含YTH结构域家族1-3与YTH结构域蛋白1-2。YTH结构域家族1和YTH结构域家族3可以共同影响已发生m6A甲基化修饰的mRNA的翻译,YTH结构域家族2能加速mRNA的降解[19];此外,YTH结构域蛋白1可以调节细胞核内RNA的剪接、出核,YTH结构域蛋白2不仅存在于细胞核中,也可以在细胞质中发挥作用。 近年来有研究发现其他类型的“阅读器”,胰岛素样生长因子2 mRNA结合蛋白可以在细胞质核糖核蛋白中与mRNA结合,增强mRNA的稳定性与调控其表达[20]。异质核内核糖核蛋白家族A2B1也有选择性剪接效应,还可以促进miRNA的加工[21]。此外,真核起始因子3与脆性智力低下蛋白也被相继证明是新的m6A阅读蛋白。 综上所述,m6A甲基化修饰对RNA的调节过程依赖于“编码器”“擦除器”与“阅读器”多种调控分子的相互作用,是一种动态可逆的表观遗传修饰方式(表1)。"
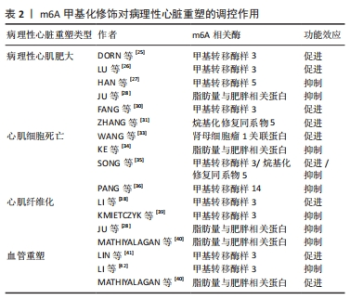
2.2 m6A甲基化修饰在病理性心脏重塑中的作用 心脏病理性重塑过程具有多种特征,包括病理性心肌肥大、心肌纤维化、心肌细胞死亡及血管重塑[22]。m6A甲基化修饰作为真核生物RNA最普遍的化学修饰,在病理性心脏重塑中已成为研究热点。 2.2.1 m6A甲基化修饰对病理性心肌肥大的调控作用 病理性心肌肥大是心脏在压力超负荷下发生的一系列复杂的代偿反应,也是病理性心脏重塑的主要发病机制,持续的病理性心肌肥大是心脏收缩功能障碍和心力衰竭的重要原因[23]。 丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)是病理性心肌肥大与重塑的关键调控分子之一[24],甲基转移酶样3介导的m6A甲基化修饰可通过激活MAPK(如MAP3K6、MAP4K5、MAPK14)的表达来促进心肌细胞代偿性肥大而无功能障碍,而心脏特异性敲除甲基转移酶样3会随着时间的推移发生心脏病理性重塑与功能障碍,并抑制心肌细胞的肥大趋势,表明甲基转移酶样3在维持心功能与心脏稳态中起重要作用[25]。此外有研究发现了病理性心肌肥大发生的新机制。泛素特异性蛋白酶12可增加甲基转移酶样3的表达,以此促进病理性心肌肥大的进程[26];除经典m6A甲基转移酶外,甲基转移酶样5也被证实可以通过促进Zeste抑制子12-肌细胞增强因子2D轴的翻译效率来抑制病理性心肌肥大[27],上述两项研究为m6A甲基转移酶调控病理性心肌肥大提供了新的证据。脂肪量与肥胖相关蛋白与YTH结构域家族2作为重要的m6A相关酶,可以有效抑制心肌细胞病理性肥大[28-29]。 现阶段已有靶向m6A甲基化修饰的药物可用于调控病理性心肌肥大。山楂酸具有广泛的药理特性,最近一项研究报道,山楂酸在体内外通过抑制甲基转移酶样3介导的m6A甲基化修饰发挥显著的抗心肌病理性肥厚作用[30]。丹参酮IIA是从丹参中提取的活性成分,可通过抑制烷基化修复同系物5的表达来治疗病理性心肌肥大[31]。 总之,m6A甲基化修饰主要通过m6A甲基转移酶来调控病理性心肌肥大的发生。当今已有针对m6A甲基化修饰的相关药物可用于病理性心肌肥大的治疗,可以进一步拓展病理性心脏重塑的靶向干预方式。 2.2.2 m6A甲基化修饰对心肌细胞死亡的调控作用 细胞死亡是各类细胞的最终阶段。心肌细胞作为一种终末分化细胞其增殖能力有限,因此控制心肌细胞的死亡对于病理性心脏重塑的干预具有重要意义。目前发现心肌细胞的死亡方式主要包括凋亡、焦亡与铁死亡等[32],关于m6A甲基化修饰相关酶参与心肌细胞死亡的调控过程多集中于对心肌细胞凋亡的研究。心肌细胞凋亡是细胞程序性死亡的一种,持续性的心肌细胞凋亡被认为是病理性心脏重塑的重要发生因素,因此阻断心肌细胞凋亡过程可以有效减轻心脏病理性重塑与心力衰竭[22]。有学者研究报道,肾母细胞瘤1关联蛋白可通过调节激活转录因子4的m6A甲基化修饰水平增加细胞凋亡率,而沉默肾母细胞瘤1关联蛋白则显著抑制了促凋亡关键分子Caspase-3的表达水平,以此减少心肌细胞凋亡[33]。效应器Yes相关蛋白是Hippo信号通路的终末效应分子,KE等[34]发现去甲基酶肪脂量与肥胖相关蛋白可擦除其下游靶点效应器Yes相关蛋白的m6A甲基化修饰,以此缓解缺氧/复氧诱导的心肌细胞凋亡过程。此外,甲基转移酶样3、烷基化修复同系物5、甲基转移酶样14也被证明是心肌细胞凋亡过程中重要的m6A甲基化修饰调控分子[35-36]。 除心肌细胞凋亡外,目前m6A甲基化相关酶对其他细胞死亡类型作用机制的相关证据并不多见,在病理性心脏重塑的研究中依然有较大前景,其中的分子机制仍有待实验进一步验证。 2.2.3 m6A甲基化修饰对心肌纤维化的调控作用 在导致病理性心脏重塑的过程中,心肌纤维化是其重要发生机制之一。心肌纤维化是由心脏成纤维细胞病理性激活与其所分泌的细胞外基质过量沉积所导致,在基于表观遗传学的心肌纤维化发生机制中m6A甲基化修饰占据了重要地位[37]。LI等[38]发现m6A甲基化修饰可影响心脏成纤维细胞的激活与分化,甲基转移酶样3可通过激活转化生长因子β/Smad 2/3途径促进心脏成纤维细胞向肌成纤维细胞的转化,从而提升心肌梗死后心肌纤维化程度。KMIETCZYK等[39]证明在主动脉弓缩窄术后,甲基转移酶样3介导的m6A甲基化修饰调控心肌纤维化程度,但可能由于实验细胞和动物模型构建方式的差异所导致的心肌纤维化机制不同,此研究中甲基转移酶样3与心肌纤维化面积呈负相关,提示同一个“编码器”“擦除器”或“阅读器”可能对一个病理过程起着相反的作用。而脂肪量与肥胖相关蛋白介导的m6A去甲基化修饰在多种模型中被证明可有效减少心肌纤维化面积[28,40]。以上结果表明,m6A甲基化修饰有潜力成为心肌纤维化新的干预方式。 2.2.4 m6A甲基化修饰对血管重塑的调控作用 血管重塑是血管内血压、血流改变多种因素共同导致的血管结构性病理生理改变,涉及许多细胞类型的变化,也是病理性心脏重塑的标志特征[22]。大量研究已证实,m6A甲基化修饰在应激条件下参与血管和心脏功能障碍的调节,例如在缺氧环境下,甲基转移酶样3可刺激脂肪源性干细胞分化为血管平滑肌细胞[41];敲降甲基转移酶样3可抑制因主动脉夹层诱导的人主动脉平滑肌细胞铁死亡的发生[42];在心力衰竭心脏中,肪脂量与肥胖相关蛋白过表达不仅可以抑制心肌纤维化,还可以促进血管生成增加[40]。但当今血管重塑过程中其他m6A相关酶的研究在心脏组织中样本量依然较少、模型不丰富,研究较为欠缺,在以后的研究中需要更多的直接证据来阐述其全面性与科学性。m6A甲基化修饰对病理性心脏重塑的调控作用,见表2。"

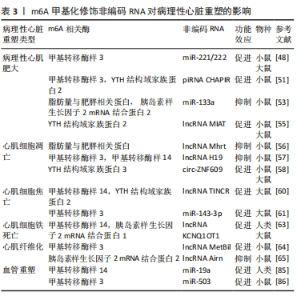
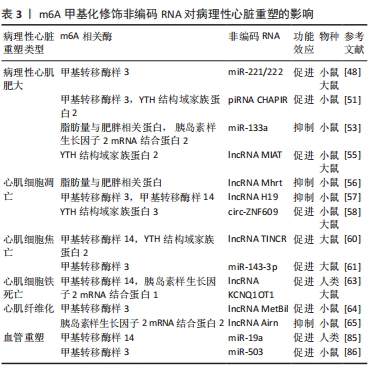
2.3 m6A甲基化修饰非编码RNA在病理性心脏重塑中的作用机制 在真核生物中,不能编码蛋白质的RNA统称为非编码RNA,常见的非编码RNA有miRNA、长链非编码RNA(long noncoding RNA,lncRNA)、环状RNA、piRNA等。由于早期的生物学手段较为落后,非编码RNA被认为缺乏开放的阅读框,是没有生物学功能的“垃圾序列”[43],但随着高通量测序的发展以及大规模转录组学的应用,研究人员观察到非编码RNA可以通过多种途径调节细胞与组织的稳态,其中转录后水平的表观遗传修饰在近年来得到了极大的研究关注。虽然m6A甲基化修饰多存在于mRNA中,但在非编码RNA中也有大量发现。已有研究证明,m6A甲基化修饰也对非编码RNA的生物学功能有调控作用[44],在生物医学领域引起了广泛关注。鉴于m6A甲基化修饰与非编码RNA之间的重要关联,并在心脏病理性重塑进程中起到不可或缺的作用,因此研究二者之间的调控机制具有深远意义。 2.3.1 m6A甲基化修饰非编码RNA在病理性心肌肥大中的作用机制 在基于病理性心肌肥大的发病机制中,多种非编码RNA发挥调控功能。QI等[45]在前期研究中发现,一些非编码RNA(例如miR-26b-5p,miR-204-5p和miR-497-3p)在维持心功能、促进心肌细胞生长以及抑制病理性心肌肥大中起到重要作用,但m6A甲基化修饰相关酶是否能通过非编码RNA作用于病理性心肌肥大的机制依旧有待深入研究挖掘。β-catenin参与了Wnt经典途径,Wnt/β-catenin信号通路的激活可促进心肌细胞的病理性肥大反应[46-47]。ZHANG等[48]发现DKK2是miR-221/222的下游靶点,同时也是Wnt/β-catenin信号通路的抑制剂,甲基转移酶样3可以通过正向调节miR-221/222的成熟过程来抑制DKK2的表达,进而激活Wnt/β-catenin信号通路并促进病理性心肌肥大。此外,糖原合成激酶3β(glycogen kinase synthase-3β,GSK-3β)与活化T细胞核因子4是已知的病理性心肌肥大调控因子[49-50],GAO等[51]发现甲基转移酶样3可与piRNA CHAPIR-PIWIL4复合物结合,催化下游因子聚腺苷酸二磷酸-核糖聚合酶10的甲基化水平来上调其表达,并抑制GSK-3β的活性,从而导致活化T细胞核因子4的积聚来促进病理性心肌肥大的发生。 miR-133a是一种已知的参与心脏发育与病理过程的关键miRNA,已有研究表明,miR-133a通过抑制病理性心肌肥大促进血管生成和心肌细胞增殖来改善心功能[52]。在血管紧张素Ⅱ诱导的肥大心肌细胞中,QIAN等[53]发现m6A甲基化修饰通过胰岛素样生长因子2 mRNA结合蛋白促进RNA诱导沉默复合物-AGO2蛋白-miR-133a复合物的聚集,以此来抑制病理性心肌肥大的发生。在对于lncRNA的研究中,lncRNA心肌梗死相关转录本(long non-coding RNA myocardial infarction associate transcript,lncRNA MIAT)于2006年首次发现[54]。YANG等[55]通过主动脉弓缩窄术与血管紧张素Ⅱ构建了体内外病理性心肌肥大模型,发现lncRNA MIAT在病理性肥大心脏中的表达显著增加,敲降lncRNA MIAT可有效减轻病理性心肌肥大表型。此外,通过RNA pull down与RIP实验证实lncRNA MIAT与YTH结构域家族蛋白2相互作用,并降低下游过氧化物酶体增殖物激活受体α信号通路中肉毒碱棕榈酰基转移酶1a的稳定性,从而抑制其表达,最终导致病理性心肌肥大。 上述研究表明,m6A甲基化修饰对病理性心肌肥大中多种非编码RNA具有重要的调控作用,m6A甲基转移酶与m6A阅读蛋白作用于不同miRNA、lncRNA、piRNA,通过多种信号通路或分子影响病理性心肌肥大的发生,这为病理性心脏重塑的发生机制提供了一个新的视角,但是否有更多种类的非编码RNA参与了m6A甲基化修饰介导的病理性心肌肥大的发生需要实验证明。此外,在病理性心肌肥大中作用最为显著的相关酶之间是否发挥协同作用依旧需要进一步探讨,后续研究中可将上述m6A相关酶与之互相作用的非编码RNA作为病理性心肌肥大的研究重点。 2.3.2 m6A甲基化修饰非编码RNA在心肌细胞死亡中的作用机制 心肌细胞受到病理性刺激后会引起心肌细胞大量死亡,是病理性心脏重塑的另一个主要特征。非编码RNA的m6A甲基化修饰主要通过心肌细胞凋亡、焦亡及铁死亡来调控病理性心脏重塑。 lncRNA肌球蛋白重链相关RNA转录本(myosin heavy chain associated RNA transcript,lncRNA Mhrt)是由肌球蛋白重链7的反义链转录而来的长链非编码RNA。心衰小鼠中m6A水平上升,SHEN等[56]推测去甲基化酶脂肪量与肥胖相关蛋白介导的m6A去甲基化修饰可能影响lncRNA Mhrt的二级结构,过表达脂肪量与肥胖相关蛋白可通过调节lncRNA Mhrt的m6A甲基化修饰水平抑制缺氧/复氧所导致的心肌细胞凋亡。缺氧预适应是一种有效提升细胞在缺氧条件下存活率的方法,缺氧预适应可抑制Caspase-3的表达水平、降低过氧化氢诱导的心肌细胞凋亡,这种保护过程依赖于甲基转移酶样3、甲基转移酶样14与lncRNA H19的生物学作用。甲基转移酶样3与甲基转移酶样14可直接与lncRNA H19结合并增加其m6A甲基化修饰水平,通过促进其表达来参与缺氧预适应的保护功能[57],提示lncRNA H19的m6A甲基化修饰在心肌细胞抗凋亡过程中发挥重要作用。现有研究已证实,在不同细胞系中m6A甲基化修饰在调控环状RNA生物发生、翻译、降解以及胞质输出等方面有着重要作用,影响多种疾病的进程[58-61]。在心脏中对环状RNA的测序已经发现其存在m6A甲基化修饰[62],有证据显示环状RNA的m6A甲基化修饰也在心肌细胞凋亡中起到重要作用。WANG等[63]发现在心肌缺血再灌注损伤后circ-ZNF609表达上调,敲除circ-ZNF609观察到心肌细胞凋亡率下降并减轻心功能障碍。进一步的研究发现,circ-ZNF609可通过Hippo-YAP信号通路与AKT信号通路之间的串扰来影响心肌细胞的存活率,YTH结构域家族蛋白3可直接与circ-ZNF609结合,并调控YAP与YTH结构域家族蛋白1和YTH结构域家族蛋白2间的相互作用,进而促进YAP的入核与翻译,介导Hippo-YAP信号通路与AKT信号通路之间的串扰。 细胞焦亡是一种具有促炎性的程序性细胞死亡方式,其特征是质膜迅速破裂,继而释放细胞内容物和促炎递质[64]。Nod样受体蛋白3(Nod like receptor protein 3,NLRP3)是Nod样受体家族成员,也是组成炎性小体的重要部分,在细胞焦亡中发挥重要调节功能。MENG等[65]发现YTH结构域家族蛋白2可识别已发生m6A甲基化修饰的lncRNA终末诱导分化长链非编码RNA(terminal differenation-induced lncRNA, lncRNA TINCR),并加速其降解,从而抑制lncRNA TINCR的表达并靶向NLRP3来参与心肌细胞的焦亡。此外,pri-miRNA的m6A甲基化修饰也参与心肌细胞焦亡过程,在心脏缺血再灌注损伤中,沉默甲基转移酶样3减轻了心肌损伤,并抑制了NLRP3、Caspase-1、GSDMD-1表达水平与炎症反应,反之过表达甲基转移酶样3通过增加成熟miR-143-3p的生成并抑制蛋白激酶C-epsilon的转录,促进心肌细胞焦亡的发生[66]。 细胞铁死亡是以铁离子积聚与脂质过氧化为主要特征的一种新型细胞死亡过程[67]。ZHUANG等[68]发现多柔比星处理后心脏中m6A修饰水平上调,甲基转移酶样14可催化其下游靶点lncRNA KCNQ1重叠转录物1(KCNQ1 overlapping transcript 1,lncRNA KCNQ1OT1)的m6A甲基化修饰,通过胰岛素样生长因子2 mRNA结合蛋白2调控lncRNA KCNQ1OT1的半衰期并抑制miR-7-5p的活性,进而促进转铁蛋白受体水平与脂质活性氧的生成,最终发生心肌细胞铁死亡。 从表观遗传学角度考虑,心肌细胞死亡过程中非编码RNA的m6A甲基化修饰可以被认为是心脏康复和治疗途径的潜在生物标志物和治疗靶点,但是关于m6A甲基化修饰与非编码RNA在某些心肌细胞死亡过程中的相互作用(例如心肌细胞焦亡、铁死亡)的直接证据依然较为单一。此外,其他一些新型细胞死亡方式(例如细胞铜死亡等)已成为研究热点,m6A甲基化修饰是否能通过非编码RNA介导其他类型的细胞死亡方式可成为进一步研究病理性心脏重塑发生机制的方向。 2.3.3 m6A甲基化修饰非编码RNA在心肌纤维化中的作用机制 虽然m6A甲基化修饰参与心肌纤维化的病理过程,但是非编码RNA的m6A甲基化修饰发生机制和功能还不明确。心肌纤维化是心肌梗死后的重要特征,在心肌梗死后心脏成纤维细胞和心脏成纤维组织中lncRNA MetBil表达上调,沉默lncRNA MetBil可抑制心肌纤维化面积。通过进一步实验证实lncRNA MetBil与甲基转移酶样3相互作用,前者可抑制甲基转移酶样3泛素化降解,以此诱导Ⅰ型胶原蛋白的表达增加,促进心肌纤维化[69]。除m6A甲基转移酶外,还有lncRNA可以与m6A阅读蛋白胰岛素样生长因子2 mRNA结合蛋白2结合并在高糖状态下发挥抗心肌纤维化作用[70]。此外据报道,氧化应激是心肌纤维化的发病机制之一。TANG等[71]发现,在心肌梗死小鼠中敲除lncRNA小核仁RNA宿主基因8(Small nucleolar RNA host gene 8,SNHG8)可减少梗死灶面积,甲基转移酶样3可以通过m6A甲基化修饰促进lncRNA SNHG8的表达和与多聚嘧啶区结合蛋白1的结合,从而调节亚铁血红素合成酶来增强氧化应激。以上结果提示,lncRNA的m6A甲基化修饰是心肌纤维化的可能发生机制。 有学者证实,lncRNA 肺腺癌转移相关转录本1(metastasis-associated lung adenocarcinoma transcript 1,lncRNA MALAT1)作为分子海绵可吸附miR-145激活转化生长因子β信号通路,促进心肌梗死后心肌纤维化,敲低其可改善改善受损的心脏功能,并抑制血管紧张素Ⅱ诱导的成纤维细胞增殖、胶原蛋白沉积和心脏成纤维细胞中α平滑肌肌动蛋白的表达[72];lncRNA生长阻滞特异性转录因子5(growth arrest-special transcript 5,lncRNA GAS5)在心肌纤维化过程中水平下调,过表达lncRNA GAS5可通过张力蛋白同源磷酸酶基因/基质金属蛋白酶2信号通路抑制心肌纤维化[73]。值得注意的是在其他组织纤维化进程中,甲基转移酶样3与脂肪量与肥胖相关蛋白可调控相关lncRNA,例如:甲基转移酶样3可促进lncRNA MALAT1的m6A甲基化水平并上调其表达;脂肪量与肥胖相关蛋白减少了lncRNA GAS5的m6A甲基化修饰来抑制其表达[74-75]。以上结果提示,某些lncRNA的m6A甲基化修饰可能通过不同途径诱导/抑制心肌纤维化进程。另外,其他miRNA(例如miRNA-133a、miR-221/222、miR-503、miR-143-3p)[76-79]、lncRNA(例如lncRNA Mhrt、lncRNA MIAT、lncRNA H19、lncRNA KCNQ1OT1)同样也在心肌纤维化过程中发挥效应[80-83],鉴于m6A甲基化修饰对不同miRNA与lncRNA的调控作用及在心脏中所发挥的功能,所以推测m6A甲基化修饰可能影响多种miRNA和lncRNA来参与心肌纤维化进程。 尽管环状RNA在心肌纤维化过程中已有研究,且发现circRNA CELF1可促进心脏成纤维细胞中脂肪量与肥胖相关蛋白的表达水平[84],但m6A甲基化修饰是否通过影响circRNA的生物学加工过程来诱导心肌纤维化暂无直接证据。综上所述,心肌纤维化过程中往往伴随m6A异常修饰,m6A甲基化修饰与多种非编码RNA之间存在潜在关,其中甲基转移酶样3修饰lncRNA调控心肌纤维化过程有潜力成为心肌纤维化与心血管疾病的靶向干预方式。尽管一些非编码RNA的m6A修饰是否调控心肌纤维化暂无直接证据,但已有研究已显示出其之间存在相关性,进一步研究二者在心肌纤维化进程中的作用机制对完善心肌纤维化的发生机制具有重要意义。 2.3.4 m6A甲基化修饰非编码RNA在血管重塑中的作用机制 血管重塑是多种因素联合驱动导致血管壁发生适应性变化的病理生理过程。尽管已证实m6A在血管重塑中发挥相关作用,但非编码RNA的m6A甲基化修饰在血管重塑过程中的潜在机制主要集中在miRNA中,其他类型非编码RNA暂无直接证据。 血管内皮细胞的增殖与迁移参与的血管重塑是导致病理性心脏重塑的重要过程,有实验表明血管内皮细胞的增殖离不开甲基转移酶样14对miR-19a成熟度的调控作用。在动脉粥样硬化模型中,甲基转移酶样14水平显著上调,并与RNA结合蛋白DGCR8结合来促进成熟miR-19a的表达,以此诱导血管内皮细胞的增殖与迁移[85]。此外,SUN等[86]通过生物信息学分析发现了在小鼠心脏中位于初级miR-503剪接位点附近的潜在m6A基序,通过实验证实甲基转移酶样3过表达可诱导小鼠血管内皮细胞中的成熟miR-503的生物发生,提示miRNA的m6A甲基化修饰对血管重塑具有可能调控作用。 m6A甲基化修饰对心血管重塑中关键非编码RNA的调控作用已有一定研究,甲基转移酶样3与甲基转移酶样14促进成熟miRNA的加工来缓解血管内皮细胞功能障碍,但其他非编码RNA是否直接参与其机制调控的可查询证据较不明确,且在血管重塑中是否作用于其他类型的细胞以及在病理性心脏重塑过程中发挥的功能也尚未阐明,需要通过更多分子生物学手段进行深入的讨论与分析。 m6A甲基化修饰非编码RNA对病理性心脏重塑的影响,见表3。m6A甲基化修饰非编码RNA调控病理性心脏重塑的作用机制,见图4。"
| [1] 王世强,常芸,饶志坚,等.运动性心脏重塑:microRNA的调节[J].体育科学, 2017,37(11):81-90. [2] 孟昶,朱磊,田雪文,等.运动诱导miRNAs调控PI3K/Akt/mTOR通路防治病理性心肌肥大的研究进展[J].中国体育科技,2021,57(9):67-75. [3] FIGUEIREDO PA, APPELL CORIOLANO HJ, DUARTE JA. Cardiac regeneration and cellular therapy: is there a benefit of exercise? Int J Sports Med. 2014;35(3):181-190. [4] 韩韦钰,赵永超,赵然尊.mRNA m6A甲基化修饰对干细胞生物学特性的影响[J].中国组织工程研究,2023,27(10):1584-1592. [5] TANG Y, CHEN K, SONG B, et al. m6A-Atlas: a comprehensive knowledgebase for unraveling the N6-methyladenosine (m6A) epitranscriptome. Nucleic Acids Res. 2021;49(D1):D134-D143. [6] CHEN YS, OUYANG XP, YU XH, et al. N6-Adenosine Methylation (m6A) RNA Modification: an Emerging Role in Cardiovascular Diseases. J Cardiovasc Transl Res. 2021;14(5):857-872. [7] 王剑,杨晓.非编码RNA在心脏稳态维持中的功能和机制[J].生命科学, 2018,30(11):1193-1201. [8] COKER H, WEI G, BROCKDORFF N. m6A modification of non-coding RNA and the control of mammalian gene expression. Biochim Biophys Acta Gene Regul Mech. 2019;1862(3):310-318. [9] DESROSIERS R, FRIDERICI K, ROTTMAN F. Identification of methylated nucleosides in messenger RNA from Novikoff hepatoma cells. Proc Natl Acad Sci U S A. 1974; 71(10):3971-3975. [10] BERULAVA T, BUCHHOLZ E, ELERDASHVILI V, et al. Changes in m6A RNA methylation contribute to heart failure progression by modulating translation. Eur J Heart Fail. 2020;22(1):54-66. [11] SHI H, WEI J, HE C. Where, When, and How: Context-Dependent Functions of RNA Methylation Writers, Readers, and Erasers. Mol Cell. 2019;74(4):640-650. [12] BOKAR JA, RATH-SHAMBAUGH ME, LUDWICZAK R, et al. Characterization and partial purification of mRNA N6-adenosine methyltransferase from HeLa cell nuclei. Internal mRNA methylation requires a multisubunit complex. J Biol Chem. 1994;269(26):17697-17704. [13] MA Z, LI Q, LIU P, et al. METTL3 regulates m6A in endometrioid epithelial ovarian cancer independently of METTL14 and WTAP. Cell Biol Int. 2020;44(12):2524-2531. [14] PING XL, SUN BF, WANG L, et al. Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase. Cell Res. 2014;24(2):177-189. [15] KNUCKLES P, LENCE T, HAUSSMANN IU, et al. Zc3h13/Flacc is required for adenosine methylation by bridging the mRNA-binding factor Rbm15/Spenito to the m6A machinery component Wtap/Fl(2)d. Genes Dev. 2018;32(5-6):415-429. [16] MELSTROM L, CHEN J. RNA N6-methyladenosine modification in solid tumors: new therapeutic frontiers. Cancer Gene Ther. 2020;27(9):625-633. [17] LI J, HAN Y, ZHANG H, et al. The m6A demethylase FTO promotes the growth of lung cancer cells by regulating the m6A level of USP7 mRNA. Biochem Biophys Res Commun. 2019;512(3):479-485. [18] ZHANG J, GUO S, PIAO HY, et al. ALKBH5 promotes invasion and metastasis of gastric cancer by decreasing methylation of the lncRNA NEAT1. J Physiol Biochem. 2019;75(3):379-389. [19] BERLIVET S, SCUTENAIRE J, DERAGON JM, et al. Readers of the m6A epitranscriptomic code. Biochim Biophys Acta Gene Regul Mech. 2019;1862(3): 329-342. [20] HUANG H, WENG H, SUN W, et al. Recognition of RNA N6-methyladenosine by IGF2BP proteins enhances mRNA stability and translation. Nat Cell Biol. 2018; 20(3):285-295. [21] ALARCÓN CR, GOODARZI H, LEE H, et al. HNRNPA2B1 Is a Mediator of m(6)A-Dependent Nuclear RNA Processing Events. Cell. 2015;162(6):1299-1308. [22] CHOY M, XUE R, WU Y, et al. Role of N6-methyladenosine Modification in Cardiac Remodeling. Front Cardiovasc Med. 2022;9:774627. [23] THAM YK, BERNARDO BC, OOI JY, et al. Pathophysiology of cardiac hypertrophy and heart failure: signaling pathways and novel therapeutic targets. Arch Toxicol. 2015;89(9):1401-1438. [24] ROMERO-BECERRA R, SANTAMANS AM, FOLGUEIRA C, et al. p38 MAPK Pathway in the Heart: New Insights in Health and Disease. Int J Mol Sci. 2020;21(19):7412. [25] DORN LE, LASMAN L, CHEN J, et al. The N6-Methyladenosine mRNA Methylase METTL3 Controls Cardiac Homeostasis and Hypertrophy. Circulation. 2019;139(4): 533-545. [26] LU P, XU Y, SHENG ZY, et al. De-ubiquitination of p300 by USP12 Critically Enhances METTL3 Expression and Ang II-induced cardiac hypertrophy. Exp Cell Res. 2021;406(1):112761. [27] HAN Y, DU T, GUO S, WANG L, et al. Loss of m6A Methyltransferase METTL5 Promotes Cardiac Hypertrophy Through Epitranscriptomic Control of SUZ12 Expression. Front Cardiovasc Med. 2022;9:852775. [28] JU W, LIU K, OUYANG S, et al. Changes in N6-Methyladenosine Modification Modulate Diabetic Cardiomyopathy by Reducing Myocardial Fibrosis and Myocyte Hypertrophy. Front Cell Dev Biol. 2021;9:702579. [29] XU H, WANG Z, CHEN M, et al. YTHDF2 alleviates cardiac hypertrophy via regulating Myh7 mRNA decoy. Cell Biosci. 2021;11(1):132. [30] FANG M, DENG J, ZHOU Q, et al. Maslinic acid protects against pressure-overload-induced cardiac hypertrophy by blocking METTL3-mediated m6A methylation. Aging (Albany NY). 2022;14(6):2548-2557. [31] ZHANG M, CHEN Y, CHEN H, et al. Tanshinone IIA alleviates cardiac hypertrophy through m6A modification of galectin-3. Bioengineered. 2022;13(2):4260-4270. [32] MISHRA PK, ADAMEOVA A, HILL JA, et al. Guidelines for evaluating myocardial cell death. Am J Physiol Heart Circ Physiol. 2019;317(5):H891-H922. [33] WANG J, ZHANG J, MA Y, et al. WTAP promotes myocardial ischemia/reperfusion injury by increasing endoplasmic reticulum stress via regulating m6A modification of ATF4 mRNA. Aging (Albany NY). 2021;13(8):11135-11149. [34] KE WL, HUANG ZW, PENG CL, et al. m6A demethylase FTO regulates the apoptosis and inflammation of cardiomyocytes via YAP1 in ischemia-reperfusion injury. Bioengineered. 2022;13(3):5443-5452. [35] SONG H, FENG X, ZHANG H, et al. METTL3 and ALKBH5 oppositely regulate m6A modification of TFEB mRNA, which dictates the fate of hypoxia/reoxygenation-treated cardiomyocytes. Autophagy. 2019;15(8):1419-1437. [36] PANG P, QU Z, YU S, et al. Mettl14 Attenuates Cardiac Ischemia/Reperfusion Injury by Regulating Wnt1/β-Catenin Signaling Pathway. Front Cell Dev Biol. 2021; 9:762853. [37] LI X, YANG Y, CHEN S, et al. Epigenetics-based therapeutics for myocardial fibrosis. Life Sci. 2021;271:119186. [38] LI T, ZHUANG Y, YANG W, et al. Silencing of METTL3 attenuates cardiac fibrosis induced by myocardial infarction via inhibiting the activation of cardiac fibroblasts. FASEB J. 2021;35(2):e21162. [39] KMIETCZYK V, RIECHERT E, KALINSKI L, et al. m6A-mRNA methylation regulates cardiac gene expression and cellular growth. Life Sci Alliance. 2019;2(2):e201800233. [40] MATHIYALAGAN P, ADAMIAK M, MAYOURIAN J, et al. FTO-Dependent N6-Methyladenosine Regulates Cardiac Function During Remodeling and Repair. Circulation. 2019;139(4):518-532. [41] LIN J, ZHU Q, HUANG J, et al. Hypoxia Promotes Vascular Smooth Muscle Cell (VSMC) Differentiation of Adipose-Derived Stem Cell (ADSC) by Regulating Mettl3 and Paracrine Factors. Stem Cells Int. 2020;2020:2830565. [42] LI N, YI X, HE Y, et al. Targeting Ferroptosis as a Novel Approach to Alleviate Aortic Dissection. Int J Biol Sci. 2022;18(10):4118-4134. [43] 范吉林,朱婷婷,田晓玲,等.非编码RNA调节心肌缺血再灌注损伤中自噬的作用及机制[J].中国组织工程研究,2022,26(35):5716-5723. [44] MA S, CHEN C, JI X, et al. The interplay between m6A RNA methylation and noncoding RNA in cancer. J Hematol Oncol. 2019;12(1):121. [45] QI J, LUO X, MA Z, et al. Downregulation of miR-26b-5p, miR-204-5p, and miR-497-3p Expression Facilitates Exercise-Induced Physiological Cardiac Hypertrophy by Augmenting Autophagy in Rats. Front Genet. 2020;11:78. [46] ZELARAYAN L, GEHRKE C, BERGMANN MW. Role of beta-catenin in adult cardiac remodeling. Cell Cycle. 2007;6(17):2120-2126. [47] LEE CY, KUO WW, BASKARAN R, et al. Increased β-catenin accumulation and nuclear translocation are associated with concentric hypertrophy in cardiomyocytes. Cardiovasc Pathol. 2017;31:9-16. [48] ZHANG R, QU Y, JI Z, et al. METTL3 mediates Ang-II-induced cardiac hypertrophy through accelerating pri-miR-221/222 maturation in an m6A-dependent manner. Cell Mol Biol Lett. 2022;27(1):55. [49] BLANKESTEIJN WM, VAN DE SCHANS VA, TER HORST P, et al. The Wnt/frizzled/GSK-3 beta pathway: a novel therapeutic target for cardiac hypertrophy. Trends Pharmacol Sci. 2008;29(4):175-180. [50] LI M, HE HP, GONG HQ, et al. NFATc4 and myocardin synergistically up-regulate the expression of LTCC α1C in ET-1-induced cardiomyocyte hypertrophy. Life Sci. 2016;155:11-20. [51] GAO XQ, ZHANG YH, LIU F, et al. The piRNA CHAPIR regulates cardiac hypertrophy by controlling METTL3-dependent N6-methyladenosine methylation of Parp10 mRNA. Nat Cell Biol. 2020;22(11):1319-1331. [52] IZARRA A, MOSCOSO I, LEVENT E, et al. miR-133a enhances the protective capacity of cardiac progenitors cells after myocardial infarction. Stem Cell Reports. 2014;3(6):1029-1042. [53] QIAN B, WANG P, ZHANG D, et al. m6A modification promotes miR-133a repression during cardiac development and hypertrophy via IGF2BP2. Cell Death Discov. 2021;7(1):157. [54] ISHII N, OZAKI K, SATO H, et al. Identification of a novel non-coding RNA, MIAT, that confers risk of myocardial infarction. J Hum Genet. 2006;51(12):1087-1099. [55] YANG Y, MBIKYO MB, ZHANG J, et al. The lncRNA MIAT regulates CPT-1a mediated cardiac hypertrophy through m6A RNA methylation reading protein Ythdf2. Cell Death Discov. 2022;8(1):167. [56] SHEN W, LI H, SU H, et al. FTO overexpression inhibits apoptosis of hypoxia/reoxygenation-treated myocardial cells by regulating m6A modification of Mhrt. Mol Cell Biochem. 2021;476(5):2171-2179. [57] SU Y, XU R, ZHANG R, et al. N6-methyladenosine methyltransferase plays a role in hypoxic preconditioning partially through the interaction with lncRNA H19. Acta Biochim Biophys Sin (Shanghai). 2020;52(12):1306-1315. [58] DI TIMOTEO G, DATTILO D, CENTRÓN-BROCO A, et al. Modulation of circRNA Metabolism by m6A Modification. Cell Rep. 2020;31(6):107641. [59] ZHAO J, LEE EE, KIM J, et al. Transforming activity of an oncoprotein-encoding circular RNA from human papillomavirus. Nat Commun. 2019;10(1):2300. [60] PARK OH, HA H, LEE Y, et al. Endoribonucleolytic Cleavage of m6A-Containing RNAs by RNase P/MRP Complex. Mol Cell. 2019;74(3):494-507.e8. [61] CHEN RX, CHEN X, XIA LP, et al. N6-methyladenosine modification of circNSUN2 facilitates cytoplasmic export and stabilizes HMGA2 to promote colorectal liver metastasis. Nat Commun. 2019;10(1):4695. [62] JAKOBI T, SIEDE D, ESCHENBACH J, et al. Deep Characterization of Circular RNAs from Human Cardiovascular Cell Models and Cardiac Tissue. Cells. 2020; 9(7):1616. [63] WANG L, YU P, WANG J, et al. Downregulation of circ-ZNF609 Promotes Heart Repair by Modulating RNA N6-Methyladenosine-Modified Yap Expression. Research (Wash D C). 2022;2022:9825916. [64] DEL RE DP, AMGALAN D, LINKERMANN A, et al. Fundamental Mechanisms of Regulated Cell Death and Implications for Heart Disease. Physiol Rev. 2019; 99(4):1765-1817. [65] MENG L, LIN H, HUANG X, et al. METTL14 suppresses pyroptosis and diabetic cardiomyopathy by downregulating TINCR lncRNA. Cell Death Dis. 2022;13(1):38. [66] WANG X, LI Y, LI J, et al. Mechanism of METTL3-Mediated m6A Modification in Cardiomyocyte Pyroptosis and Myocardial Ischemia-Reperfusion Injury. Cardiovasc Drugs Ther. 2023;37(3):435-448. [67] GAO M, MONIAN P, QUADRI N, et al. Glutaminolysis and Transferrin Regulate Ferroptosis. Mol Cell. 2015;59(2):298-308. [68] ZHUANG S, MA Y, ZENG Y, et al. METTL14 promotes doxorubicin-induced cardiomyocyte ferroptosis by regulating the KCNQ1OT1-miR-7-5p-TFRC axis. Cell Biol Toxicol. 2021.doi: 10.1007/s10565-021-09660-7. [69] ZHUANG Y, LI T, HU X, et al. MetBil as a novel molecular regulator in ischemia-induced cardiac fibrosis via METTL3-mediated m6A modification. FASEB J. 2023; 37(3):e22797. [70] PENG T, LIU M, HU L, et al. LncRNA Airn alleviates diabetic cardiac fibrosis by inhibiting activation of cardiac fibroblasts via a m6A-IMP2-p53 axis. Biol Direct. 2022;17(1):32. [71] TANG J, TANG QX, LIU S. METTL3-modified lncRNA-SNHG8 binds to PTBP1 to regulate ALAS2 expression to increase oxidative stress and promote myocardial infarction. Mol Cell Biochem. 2023;478(6):1217-1229. [72] HUANG S, ZHANG L, SONG J, et al. Long noncoding RNA MALAT1 mediates cardiac fibrosis in experimental postinfarct myocardium mice model. J Cell Physiol. 2019;234(3):2997-3006. [73] LIU HL, CHEN CH, SUN YJ. Overexpression of lncRNA GAS5 attenuates cardiac fibrosis through regulating PTEN/MMP-2 signal pathway in mice. Eur Rev Med Pharmacol Sci. 2019;23(10):4414-4418. [74] LIU P, ZHANG B, CHEN Z, et al. m6A-induced lncRNA MALAT1 aggravates renal fibrogenesis in obstructive nephropathy through the miR-145/FAK pathway. Aging (Albany NY). 2020;12(6):5280-5299. [75] LI X, LI Y, WANG Y, et al. The m6A demethylase FTO promotes renal epithelial-mesenchymal transition by reducing the m6A modification of lncRNA GAS5. Cytokine. 2022;159:156000. [76] YU BT, YU N, WANG Y, et al. Role of miR-133a in regulating TGF-β1 signaling pathway in myocardial fibrosis after acute myocardial infarction in rats. Eur Rev Med Pharmacol Sci. 2019;23(19):8588-8597. [77] VERJANS R, PETERS T, BEAUMONT FJ, et al. MicroRNA-221/222 Family Counteracts Myocardial Fibrosis in Pressure Overload-Induced Heart Failure. Hypertension. 2018;71(2):280-288. [78] ZHOU Y, DENG L, ZHAO D, et al. MicroRNA-503 promotes angiotensin II-induced cardiac fibrosis by targeting Apelin-13. J Cell Mol Med. 2016;20(3):495-505. [79] LI C, LI J, XUE K, et al. MicroRNA-143-3p promotes human cardiac fibrosis via targeting sprouty3 after myocardial infarction. J Mol Cell Cardiol. 2019;129:281-292. [80] LANG M, OU D, LIU Z, et al. LncRNA MHRT Promotes Cardiac Fibrosis via miR-3185 Pathway Following Myocardial Infarction. Int Heart J. 2021;62(4):891-899. [81] YAO L, ZHOU B, YOU L,et al. LncRNA MIAT/miR-133a-3p axis regulates atrial fibrillation and atrial fibrillation-induced myocardial fibrosis. Mol Biol Rep. 2020; 47(4):2605-2617. [82] GUO F, TANG C, HUANG B, et al. LncRNA H19 Drives Proliferation of Cardiac Fibroblasts and Collagen Production via Suppression of the miR-29a-3p/miR-29b-3p-VEGFA/TGF-β Axis. Mol Cells. 2022;45(3):122-133. [83] YANG F, QIN Y, LV J, et al. Silencing long non-coding RNA Kcnq1ot1 alleviates pyroptosis and fibrosis in diabetic cardiomyopathy. Cell Death Dis. 2018;9(10):1000. [84] LI XX, MU B, LI X, et al. circCELF1 Inhibits Myocardial Fibrosis by Regulating the Expression of DKK2 Through FTO/m6A and miR-636. J Cardiovasc Transl Res. 2022;15(5):998-1009. [85] ZHANG BY, HAN L, TANG YF, et al. METTL14 regulates M6A methylation-modified primary miR-19a to promote cardiovascular endothelial cell proliferation and invasion. Eur Rev Med Pharmacol Sci. 2020;24(12):7015-7023. [86] SUN P, WANG C, MANG G, et al. Extracellular vesicle-packaged mitochondrial disturbing miRNA exacerbates cardiac injury during acute myocardial infarction. Clin Transl Med. 2022;12(4):e779. |
| [1] | Yu Weijie, Liu Aifeng, Chen Jixin, Guo Tianci, Jia Yizhen, Feng Huichuan, Yang Jialin. Advantages and application strategies of machine learning in diagnosis and treatment of lumbar disc herniation [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(9): 1426-1435. |
| [2] | Lin Zeyu, Xu Lin. Research progress in gout-induced bone destruction mechanism [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(8): 1295-1300. |
| [3] | Liu Jianhong, Liao Shijie, Li Boxiang, Tang Shengping, Wei Zhendi, Ding Xiaofei. Extracellular vesicles carrying non-coding RNA regulate the activation of osteoclasts [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(7): 1076-1082. |
| [4] | Ma Shuwei, He Sheng, Han Bing, Zhang Liaoyun. Exosomes derived from mesenchymal stem cells in treatment of animals with acute liver failure: a meta-analysis [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(7): 1137-1142. |
| [5] | Zhang Kefan, Shi Hui. Research status and application prospect of cytokine therapy for osteoarthritis [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(6): 961-967. |
| [6] | Li Jiaqi, Huang Yuanli, Li Yan, Wang Chunren, Han Qianqian. Mechanism and influencing factors in molecular weight degradation of non-cross-linked hyaluronic acid [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(5): 747-752. |
| [7] | Xu Rong, Wang Haojie, Geng Mengxiang, Meng Kai, Wang Hui, Zhang Keqin, Zhao Huijing. Research advance in preparation and functional modification of porous polytetrafluoroethylene artificial blood vessels [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(5): 759-765. |
| [8] | Chen Xiaofang, Zheng Guoshuang, Li Maoyuan, Yu Weiting. Preparation and application of injectable sodium alginate hydrogels [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(5): 789-794. |
| [9] | Liu Chuang, Shan Shuo, Yu Tengbo, Zhou Huan, Yang Lei. Advantages, discomfort and challenges of clinical application of orthopedic hemostatic materials [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(5): 795-803. |
| [10] | Zhang Ming, Wang Bin, Jia Fan, Chen Jie, Tang Wei. Application of brain-computer interface technology based on electroencephalogram in upper limb motor function rehabilitation of stroke patients [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(4): 581-586. |
| [11] | He Yuanjie, Chen Yuheng, Zhao Yongchao, Wang Zhenglong. Progress in epigenetic regulation of vascular smooth muscle cell remodeling in the occurrence and development of aortic aneurysms [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(4): 602-608. |
| [12] | Ma Sicong, Chen Jing, Li Yunqing. Functions and roles of connective tissue growth factor in nervous systems [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(4): 615-620. |
| [13] | Yan Binghan, Li Zhichao, Su Hui, Xue Haipeng, Xu Zhanwang, Tan Guoqing. Mechanisms of traditional Chinese medicine monomers in the treatment of osteoarthritis by targeting autophagy [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(4): 627-632. |
| [14] | Wang Xinyi, Xie Xianrui, Chen Yujie, Wang Xiaoyu, Xu Xiaoqing, Shen Yihong, Mo Xiumei. Electrospun nanofiber scaffolds for soft and hard tissue regeneration [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(3): 426-432. |
| [15] | Long Jundong, Shi Yehong, Wang Cheng, Chen Shijiu. Effects of different freezing techniques on the rejection of allogeneic vascular transplantation [J]. Chinese Journal of Tissue Engineering Research, 2024, 28(3): 433-438. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||

