Chinese Journal of Tissue Engineering Research ›› 2021, Vol. 25 ›› Issue (7): 1122-1128.doi: 10.3969/j.issn.2095-4344.2180
Previous Articles Next Articles
Advances in research and application of breast cancer organoids
Jiao Hui, Zhang Yining, Song Yuqing, Lin Yu, Wang Xiuli
- College of Basic Medical Sciences, Dalian Medical University, Dalian 116044, Liaoning Province, China
-
Received:2020-04-01Revised:2020-04-09Accepted:2020-05-13Online:2021-03-08Published:2020-12-09 -
Contact:Wang Xiuli, MD, Professor, College of Basic Medical Sciences, Dalian Medical University, Dalian 116044, Liaoning Province, China -
About author:Jiao Hui, Master candidate, College of Basic Medical Sciences, Dalian Medical University, Dalian 116044, Liaoning Province, China -
Supported by:the National Natural Science Foundation of China, No. 31771068
CLC Number:
Cite this article
Jiao Hui, Zhang Yining, Song Yuqing, Lin Yu, Wang Xiuli. Advances in research and application of breast cancer organoids[J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1122-1128.
share this article
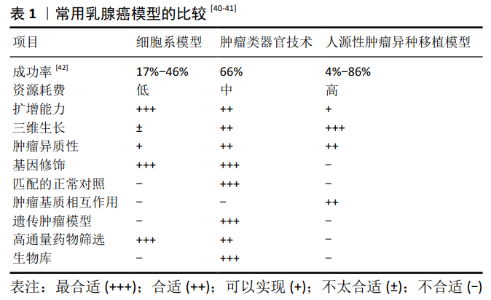
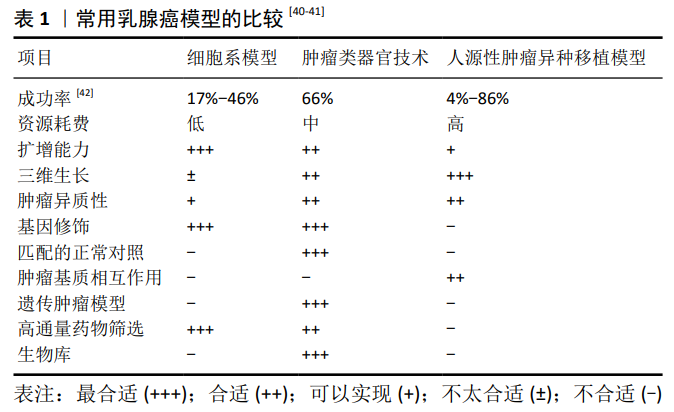
2.1 类器官技术 目前,传统的体外肿瘤细胞培养模型主要是细胞系模型(cell lines,CLs)和人源性肿瘤异种移植模型(patient-derived xenografts,PDXs)。细胞系模型简单、经济,可以进行高通量药物筛选,最常用。然而,细胞系通常来自单个细胞,缺乏细胞-细胞、细胞-细胞基质间的相互作用,不能重述肿瘤细胞的多样性[8]。此外,长期的体外平面培养,使肿瘤细胞丧失异质性,不能维持其特有的组织来源特征,与原肿瘤生长状态不一致,因此无法模拟患者体内复杂的三维环境及组织细胞功能和相关的信号通路[9]。最近有研究表明,乳腺癌细胞系的遗传和转录进化可显著改变药物应答[10]。例如,二维培养导致不同实验室间的MCF-7细胞系在生长速度、激素受体状态、核型和克隆方面存在着差异。人源性肿瘤异种移植模型虽在模拟体内微环境方面具有独特的优势,但动物实验研究培养周期长、花费高,不能进行高通量药物筛选;再者具有种属差异,动物体内环境体系复杂,变异因素众多,在数据采集分析和阐明机制方面均存在很大难度,无法进行单因素分析[11]。这些模型存在肿瘤异质性缺乏等缺陷,致使它们都不能很好地还原患者体内肿瘤的生长状态,给乳腺癌研究带来了模型失真性等问题,导致在体外模型开发的大多数药物最终在人类临床试验中失败。 近年来,伴随组织工程研究的不断深入,体外三维培养模型愈来愈受到研究者的关注,其中类器官技术发展最为迅猛。该技术以干细胞或器官祖细胞为原料,经过体外培养,能发生类似体内细胞分选及谱系定向,即自组装成器官样结构的细胞团,表现出与来源组织相似的结构和功能,为模拟活体器官提供新的体外模型[12-13]。2009年,HANS CLEVERS团队将来源于小鼠肠道的成体lgr5阳性干细胞培养在含有R-spondin、EGF、Noggin因子的三维基质凝胶中,形成了类似小肠的显微结构,即隐窝-绒毛复合体,成功建立了肠类器官培养系统[14]。由于成人干细胞培养缺乏间充质细胞,一般需要在培养基中加入细胞因子和细胞外基质形成干细胞培养微环境,这是类器官更新和分化的物质基础,通过人为调控培养系统的成分,细胞可以自动分化为特定结构并完成类器官自组装过程[15]。研究人员通过修改加入的因子成分和细胞分离程序,成功摸索出其他上皮类器官体外生成和长期扩增的培养条件[16],如肝[17]、肺[18]、肾[19]、胰腺等[20]。 由类器官衍生而来的肿瘤类器官技术(patient-derived organoids,PDOs),主要是利用来源于患者的新鲜肿瘤组织,在特定的液体培养条件下进行体外三维培养,形成微型肿瘤模型,具有自我更新和自我组织能力,并且维持了其来源组织的生理结构和功能[13],用于模拟体内肿瘤组织的生物学特征[21],开启肿瘤个性化治疗新纪元。肿瘤类器官技术代表了在体外细胞系模型和体内人源性肿瘤异种移植模型之间的第3种临床前模型,且能兼具两者优点。它取材灵活,可通过手术、活检等方式获取样本[22-23];与细胞系模型相比,能够实现和患者的匹配,再现原始肿瘤异质性[24]。与人源性肿瘤异种移植模型相比,小鼠培养周期长达4-8个月,而类器官培养周期较短,一般为一两个月,成本低,能进行高通量药物筛选和长期传代培养,且具有稳定的表型和遗传学特性,是肿瘤生物学和干细胞生物学研究的有力工具[25]。2011年SATO等[26]首次成功培养出肿瘤来源的类器官——结肠癌类器官,随后又建立了胰腺癌[27]、前列腺癌[28]、肝癌等肿瘤类器官[29],涉及药物敏感性检测[30-31]、肿瘤发展与耐药机制等各个研究方向[32-36],为乳腺癌类器官的建立和研究提供了可能。 2.2 乳腺癌类器官的建立 2.2.1 培养现状 2018年,HANS CLEVERS团队中SACHS等[37] 从知情同意下接受肿瘤切除术的患者中获得乳腺癌组织,通过机械破坏和酶消化相结合的方法分离乳腺癌细胞。将分离的细胞接种在Matrigel中,在类器官通用培养基的基础 上[14, 16],进行了乳腺癌类器官培养条件的探索。他们发现,神经调节蛋白1(Neuregulin 1)是一个关键的补充因子,它是一种跨膜蛋白,是人表皮生长因子受体(human epidermal growth factor receptor,HER)酪氨酸激酶3和4的配体,与乳房发育和肿瘤发生有关[38]。有研究表明,它通过激活HER2/HER3信号通路,促进细胞增殖和自我更新[39]。将神经调节蛋白1添加到乳腺癌类器官培养基中,使乳腺癌类器官高效生成,并使其能长期扩增>20代。另外,他们发现表皮生长因子质量浓度高于5 μg/L虽然能促进细胞增殖,但会使乳腺癌类器官逐渐在Matrigel中下沉,失去三维培养条件;p38抑制剂SB202190浓度高于1 μmol/L会降低乳腺癌类器官建立的效率;添加特异性的ROCK蛋白抑制剂Y-27632也能促进乳腺癌类器官在体外长期增殖,而添加Wnt-3A并未明显改善培养条件。SACHS等[37]通过对>150例患者的乳腺癌类器官培养条件的探索,进一步加深了对乳腺癌细胞生长环境的认识,使乳腺癌类器官建立的成功率达80%以上。但相对于其他肿瘤类器官,乳腺癌类器官平台的建立时间相对比较晚,培养条件还不是特别成熟,其他实验室按照HANS CLEVERS团队的配方和操作流程建立的乳腺癌类器官成功率远低于此,见表1[40-42],尤其在样本量少的情况下。2019年有研究者认为组织的类别、样本量和培养条件完善程度是影响乳腺癌类器官成功率的关键因素[23],结合课题组对培养乳腺癌类器官探索的过程,作者认为乳腺癌类器官的操作流程仍需进一步细化,明确操作规范,以降低培养的失败率。 2.2.2 具体研究内容 研究者成功建立了乳腺癌类器官生物样本库,这种临床前模型能否捕获疾病的异质性,能否更有效地将基础癌症研究转化为临床应用,研究者对纳入类器官样本库的样本进行如下评估: (1)组织病理学:乳腺癌被分为不同的亚型,结合受体状态,组织学分级等组织病理学特征,有助于描述乳腺癌的恶性程度,结合临床表现可进行干预治疗,并估计患者预后。①苏木精-伊红染色:为了检验乳腺癌类器官是否与原肿瘤的组织学类型(如浸润性导管癌、浸润性小叶癌)相匹配,研究人员用苏木精-伊红染色对原肿瘤组织和类器官切片进行组织病理学分析,结果发现乳腺癌类器官可见典型的恶性形态特征,细胞核异型性,乳腺癌类器官的表型常与原肿瘤组织一致[37]。②免疫组织化学染色:除了组织学特点外,一个有代表性的乳腺癌模型应该保留最重要和最普遍的乳腺癌生物学标志物:ER、PR和HER2。这3个指标是乳腺癌病理检查的常用指标,可以帮助医生了解肿瘤的恶性程度以及制定治疗方案。激素受体ER和PR的状态对激素/内分泌治疗的结果具有预测价值,而HER2的状态可以预测全身化疗的结果,其本身就是癌症治疗的一个靶向目标[43]。通过免疫组织化学染色,研究发现75% ER/PR阳性的乳腺癌形成的类器官呈 ER/PR阳性,90% ER/PR 阴性的乳腺癌形成的类器官呈ER/PR阴性,80% HER2阳性的乳腺癌形成的类器官呈HER2阳性,90% HER2阴性的乳腺癌形成的类器官呈HER2阴性,这说明激素受体和HER2状态在大多数乳腺癌类器官都有保留,与原乳腺癌组织的相符率为75%-90%以上[43]。③活/死染色:对乳腺癌类器官进行活/死染色,了解肿瘤细胞团的活性状态[44]。 (2)基因测序:肿瘤的发生发展和耐药性常常由基因组变异所导致,通过二代测序获取乳腺癌基因组信息,有助于研究乳腺癌发生发展的相关机制,有助于通过分析突变基因来进行乳腺癌的诊断和预后评估,有助于通过获取抗肿瘤药物的靶向位点来进行肿瘤靶向药物的治疗。单核苷酸变异(single nucleotide variant,SNV)是指在基因组上单个核苷酸变异,拷贝数变异(copy number variation,CNV)是指基因组中大片段DNA的拷贝数改变[45]。既往研究表明,乳腺癌中拷贝数变异和单核苷酸变异与疾病预后、药物耐受性有着密切联系[46]。为了进一步表征类器官,SACHS等[37]对乳腺癌组织和类器官进行了全基因组测序,进行拷贝数变异、单核苷酸变异分析。研究发现拷贝数变异的增加和减少被保留在整个基因组中,乳腺癌类器官经常显示比原乳腺癌组织更清晰和更直接的拷贝数信号,并且乳腺癌类器官的拷贝数变异信号经过连续传代后(>20代,每代1-4周)依然保持稳定。他们还发现某些乳腺癌特异的突变位点在多个肿瘤组织-类器官配对中都有保留,包括CDKN2A、ERBB2、NF1和SNX31。乳腺癌类器官显示与其来源肿瘤组织相似的突变特征,其缺失、过表达或野生型的状态均与原肿瘤一致。比如在抑癌基因TP53和CDH1中发现失活突变,在致癌基因PIK3CA中发现MLL2/3表观遗传复合物和激活突变。因此,拷贝数变异、单核苷酸变异检测表明了乳腺癌类器官基因水平的高拟合度和稳定性[47]。 (3)基因表达:基因表达谱对乳腺癌的分类具有重要贡献[48]。为了确定类器官是否为肿瘤异质性的真正模型,除了将类器官的基因组谱与肿瘤组织比较,还需要将表达谱与肿瘤组织进行比较。SACHS等[37]通过对22个乳腺癌类器官进行RNA测序,并根据> 25 000个基因组装相关热图,比较了乳腺癌组织和乳腺癌类器官的基因表达,发现乳腺癌类器官的基因表达谱几乎与原乳腺癌组织的基因表达谱相一致。前人已提出基于RNA表达的乳腺癌分类系统:PAM50、SCMGENE、SCMOD1和IntClust[49],前3个分类系统主要与受体状态相关,以基底型、管腔型A和正常亚型为主导的PAM50,以ER/HER2阴性为主导的SCMGENE和HER阳性亚型为主导的SCMOD1。SACHS等[37]发现分类系统中几乎所有亚型均可找到相对应的乳腺癌类器官。因此,乳腺癌类器官具有代表性的基因表达谱,可以在不存在明显培养偏差的情况下,分级聚类成大多数基于RNA表达的乳腺癌亚型,并有助于上皮细胞生物学现象的表征,进一步强调了它们作为乳腺癌代表模型的潜力。 上述研究表明,乳腺癌类器官始终保持着原始组织的组织学、基因组、转录组特征,为人们更好地探究乳腺癌提供了一种非常有前景的新模型。"
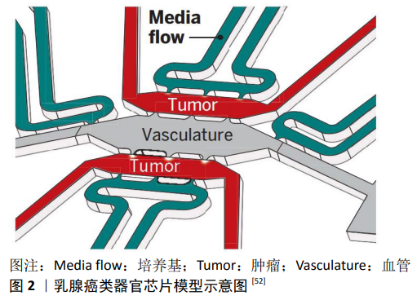
2.3 乳腺癌类器官前沿进展 2.3.1 乳腺癌类器官共培养模型 肿瘤微环境对肿瘤的生长、转移和耐药的产生发挥重要作用。肿瘤类器官模型通常缺乏完整的微环境。研究表明,类器官与肿瘤微环境细胞共培养模型,更接近肿瘤在体内的状态[24]。KOLODOVA[50]将乳腺癌类器官与癌症相关成纤维细胞共培养,发现癌症相关成纤维细胞能促进乳腺癌细胞的增殖,其原因是乳腺癌细胞受上皮-基质相互作用的密切调节,包括旁分泌信号、细胞外基质重塑和机械感应。CHATTERJEE等[51]通过构建乳腺癌类器官-成纤维细胞(正常乳腺成纤维细胞和癌症相关成纤维细胞)共培养模型,发现了一种促肿瘤性旁分泌信号机制,并且这种旁分泌信号机制在正常乳腺成纤维细胞和癌症相关成纤维细胞之间是共享的。研究发现,正常乳腺成纤维细胞或癌症相关成纤维细胞分泌C-C基序趋化因子配体7、白细胞介素6和白细胞介素8,导致ER阳性乳腺癌细胞释放血小板衍生生长因子BB,然后血小板衍生生长因子BB以旁分泌的方式作用,导致从正常乳腺成纤维细胞和癌症相关成纤维细胞产生白细胞介素1b。白细胞介素1b作为促进增殖的因子,促进ER阳性癌细胞的增殖。 2.3.2 新乳腺癌类器官模型 目前常规的乳腺癌类器官模型大多依赖基于细胞外基质做底物,如Matrigel。虽然这种基于细胞外基质的底物对体外细胞的存活有很大的贡献,但它们需要锁定不必要的信号通路,以确保与原始肿瘤的一致性。此外,它们与乳腺肿瘤组织的弹性系数不匹配,导致不同的机械转导,从而改变细胞信号[52]。因此,它不是理想的可模仿原发肿瘤的模型。最近,有研究者开发了一种可生物降解的三维机械支架来研究乳腺癌的转移[44]。首先制作聚己内酯多孔蛋白支架。在乳腺癌中,细胞外基质主要由癌症相关成纤维细胞分泌和重塑,其分泌物在肿瘤微环境中具有多向性功能[53]。为了提高多孔聚己内酯支架的表面生物活性,同时保持机械性能,需要在支架上提供一个额外的、天然的细胞外基质涂层来促进肿瘤细胞的附着;接下来在支架上培养乳腺癌症相关成纤维细胞,当支架上沉积胶原和纤维连接蛋白时,洗脱掉支架上的癌症相关成纤维细胞,生成优化后的混合支架,其保留了癌症相关成纤维细胞沉积物;最后把患者来源的乳腺癌细胞在混合支架和裸支架上进行培养。结果发现,与裸支架相比,乳腺癌细胞在混合支架上大量附着并形成类器官,并且细胞在混合支架上表现出更高的增殖活力;随后进行多种化疗药物反应分析,发现混合支架上乳腺癌类器官可以捕捉到药物反应时患者间的异质性。综合起来得出结论,聚己内酯支架涂上天然细胞外基质用于乳腺癌类器官的培养,能更好地模拟体内微环境,这表明癌症相关成纤维细胞衍生的细胞外基质为细胞提供了显著的生长优势。该模型可作为一种新的乳腺癌类器官模型,以发展有效和个性化的治疗方案。 2.3.3 乳腺癌类器官芯片 器官芯片(Organs-on-a-chip)是仿生生物学和微加工技术的结合,利用微流控技术控制流体流动,结合细胞与细胞相互作用、基质特性以及生物化学和生物力学特性,在芯片上构建三维的人体器官生理微系统。简单来说,器官芯片是一种用于体外模拟人体器官功能单元的微型细胞培养装置。类器官缺乏上皮肿瘤细胞与周围血管系统和基质之间的组织界面,而这些组织界面是癌症控制和进展的重要因素,不能很好地复制器官发展过程中复杂又动态的微环境,为了解决这一缺点,科学家将器官芯片技术与类器官相整合而形成“类器官芯片”技术[54]。随着类器官的增大,其仅仅依靠被动扩散来获取养分和氧气并去除代谢产物的方法不足以满足它们对代谢物日益增加的需求,最终导致它们的生长和成熟失败[55]。而用器官芯片来模拟灌注血管被证明可以解决这一问题[56],利用微流控平台使类器官血管化,并允许生理流动条件下的灌注,从而再现了肿瘤微环境在体内的运输特性。SHIRURE等[57]开发的乳腺癌类器官芯片也证实了这一点。1个乳腺癌肿瘤类器官芯片模型包含3个相互联系的微液流装置,支持内皮细胞的血管生成自组装可灌流血管的三维网络,并长成乳腺癌类器官结构,见图2,利用此装置通过血管灌流紫杉醇可以抑制肿瘤的生长,表明该模型可以用来筛选患者个性化的药物。血运化是一种很有前途的策略,可以解决乳腺癌类器官中营养供应和寿命有限的问题。 2.4 乳腺癌类器官的应用 2.4.1 药物筛选 乳腺癌类器官的主要用途之一是药物敏感检测。在乳腺癌治疗方式中,靶向药物治疗是一个重要的组成部分。SACHS等[37]利用乳腺癌类器官生物样本库,对作用于靶向HER信号通路的药物(如阿法替尼、吉非替尼、依维莫司、AZD8055等)、腺苷二磷酸核糖聚合酶抑制剂(如奥拉帕尼、尼拉帕尼等)进行了药敏检测。他们发现,多数HER2依赖的类器官对HER2靶向治疗敏感,而HER2阴性的类器官是耐药的。同样,大部分具有高BRCA1/2信号的类器官对腺苷二磷酸核糖聚合酶抑制剂敏感,而来自低BRCA1/2信号的类器官对腺苷二磷酸核糖聚合酶抑制剂不敏感。值得注意的是,一小部分类器官对药物的敏感性与HER2、BRCA1/2基因特征不相符,尽管受体高表达,但对靶向治疗不敏感,这表明耐药机制存在于类器官中,与人源性肿瘤异种移植模型和临床研究一致。他们又把乳腺癌类器官移植到小鼠体内,结果发现在小鼠体内实验的药敏结果与体外类器官结果一致。WALSH等[58]通过乳腺癌类器官利用光学代谢成像技术来评估药物反应,他们的研究证实类器官体外药物反应可以预测体内反应。CHATTERJEE等[51]发现他莫昔芬联合白细胞介素1b和血小板衍生生长因子BB受体阻滞剂,对恶性ER阳性乳腺癌类器官作用效果明显,这表明阻断ER阳性乳腺癌细胞-癌症相关成纤维细胞旁分泌信号可以显著增强他莫西芬的作用,对临床治疗具有启示意义。把类器官药敏检测结果和基因检测结果相结合进行分析,尽早获得有效药物的信息,避免盲目用药、延误治疗,对患者个性化用药具有深远意义。 使用类器官模型检测药物对患者正常组织的毒性是药物筛选的另一研究方向。乳腺癌研究中尚缺乏针对正常组织的药物毒理实验数据,如果同时获得肿瘤和肿瘤周围正常组织,并分别进行类器官培养和药物处理,分析正常组织和肿瘤类器官的存活率和突变频率等,评估药物疗效、药代动力学、药物不良反应和继发肿瘤的风险,从而避免患者受到不必要的伤害,对临床用药具有重大意义。 2.4.2 机制研究 除了药敏检测以外,乳腺类器官也被用来进行与肿瘤发生和转移相关的信号通路研究。DEKKERS等[59]获取正常乳腺组织上皮,并进行长期培养与扩增,借助的CRISPR/Cas9基因编辑技术,靶向敲除乳腺癌相关抑癌基因P53、PTEN、RB1、NF1,借此可以清楚地评估单个基因突变的致瘤能力。乳腺类器官突变成乳腺癌类器官,重述了肿瘤发生过程。这些突变的类器官形成ER阳性的肿瘤,能在体外长期培养;把它们移植到小鼠体内,经验证1/6小鼠发生了P53/PTEN/RB1突变,3/6发生了P53/PTEN/RB1/NF1突变,并且这些突变的类器官对内分泌治疗或化疗均有反应。该研究证实至少3个抑癌基因必须失活才能发生癌变,增加了对乳腺肿瘤发生过程中关键驱动因素的认识。剪接因子的错误调控是肿瘤发生的重要标志,PARK等[60]利用乳腺类器官分析其致癌功能,他们发现50%的乳腺肿瘤至少有1个SR蛋白家族成员的改变,通过上调SRSF4、SRSF6或TRA2β这3种剪接因子,破坏了乳腺腺泡的形成,并能促进乳腺细胞增殖和侵袭。CHEUNG等[61]通过培养乳腺癌类器官发现表达CK14和P63的细胞是入侵发生最早的细胞,同时发现原乳腺癌组织在入侵边界处高表达CK14。将敲除CK14的乳腺癌类器官移植到小鼠体内发现该肿瘤失去了侵袭性,证明该入侵行为是由一群基底样的上皮细胞所“领导”起始的,该研究为研究乳腺癌转移的起始机制奠定了理论基础。 目前,对乳腺癌机制的研究比较缺乏,如果乳腺癌类器官技术能够进一步推广应用,将有助于进一步识别新的癌症驱动基因和新的药物靶点,将有助于更好地理解乳腺肿瘤细胞侵袭、转移和耐药的机制。 2.4.3 新药研发 乳腺癌的治疗尽管取得了诸多进展,但癌症依旧是全世界的一大健康难题。除预防和早期诊断之外,在癌症的防治上还需要更多创新靶向疗法。然而新药研发并不是一件容易的事,将创新科学转化为有效疗法是新药上市之路的一大瓶颈。这背后的原因之一在于使用的癌症模型往往不能很好地复制肿瘤的组织复杂性与遗传异质性。类器官平台的建立使这一情况出现了转机,并且类器官在机制研究中新发现的基因或许是新药物治疗的靶点。 在癌症治疗中,利用患者本身的免疫系统来清除癌细胞的做法已经取得了巨大成功。考虑到肿瘤的新抗原未必能带来想要的免疫反应,科学家们正在尝试在体外对免疫细胞进行激活,再应用到患者体内。而体外激活的部分可以通过类器官来实现。例如ZUMWALDE等[62]将正常乳腺导管上皮形成的类器官与Vδ 2+T淋巴细胞共培养,发现这些T细胞能对三阴性乳腺癌细胞进行有效的杀伤。这一研究结果让研究者看到了类器官在肿瘤免疫疗法里的巨大应用前景。另外,也能利用类器官平台对研发的新药进行大规模测试。建立大规模乳腺癌类器官生物样本库,为研究者提供新药测试平台,从而显著缩短样本收集、受试人群招募的时间,降低前期研究成本。对于患者而言,如果类器官测试能取代新药研发Ⅰ期、Ⅱ期试验,则能避免对受试人群带来的潜在损害[63]。未来,进一步优化类器官平台上的新药筛选流程,无疑将更快、更好地把个体化疗法带向临床。"

| [1] BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 2018;68(6):394-424. [2] CHEN W, ZHENG R, BAADE PD, et al. Cancer statistics in China, 2015. CA Cancer J Clin. 2016;66(2):115-132. [3] 郑荣寿,孙可欣,张思维,等. 2015年中国恶性肿瘤流行情况分析[J].中华肿瘤杂志,2019,41(1):19-28. [4] TELANG N. Putative cancer-initiating stem cells in cell culture models for molecular subtypes of clinical breast cancer. Oncol Lett. 2015;10(6):3840-3846. [5] ZHANG J, LIU D, FENG Z, et al. MicroRNA-138 modulates metastasis and EMT in breast cancer cells by targeting vimentin. Biomed Pharmacother. 2016;77:135-141. [6] GNANT M, HARBECK N, THOMSSEN C. St. Gallen 2011: Summary of the Consensus Discussion. Breast Care (Basel). 2011;6(2):136-141. [7] NIK-ZAINAL S, DAVIES H, STAAF J, et al. Landscape of somatic mutations in 560 breast cancer whole-genome sequences. Nature. 2016;534(7605):47-54. [8] GOODSPEED A, HEISER LM, GRAY JW, et al. Tumor-Derived Cell Lines as Molecular Models of Cancer Pharmacogenomics. Mol Cancer Res. 2016;14(1):3-13. [9] NAGLE PW, PLUKKER JTM, MUIJS CT, et al. Patient-derived tumor organoids for prediction of cancer treatment response. Semin Cancer Biol. 2018;53:258-264. [10] BEN-DAVID U, SIRANOSIAN B, HA G, et al. Genetic and transcriptional evolution alters cancer cell line drug response. Nature. 2018;560(7718): 325-330. [11] SCHUTGENS F, CLEVERS H. Human Organoids: Tools for Understanding Biology and Treating Diseases. Annu Rev Pathol. 2020;15:211-234. [12] LANCASTER MA, KNOBLICH JA. Organogenesis in a dish: modeling development and disease using organoid technologies. Science. 2014; 345(6194):1247125. [13] LI M, IZPISUA BELMONTE JC. Organoids - Preclinical Models of Human Disease. N Engl J Med. 2019;380(6):569-579. [14] SATO T, VRIES RG, SNIPPERT HJ, et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 2009;459(7244):262-265. [15] 董研博,王健,郑梦竹,等.类器官研究进展及应用[J].中国药理学与毒理学杂志,2019,33(2):152-160. [16] SATO T, CLEVERS H. SnapShot: Growing Organoids from Stem Cells. Cell. 2015;161(7):1700-1700.e1. [17] HUCH M, GEHART H, VAN BOXTEL R, et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 2015; 160(1-2):299-312. [18] NADKARNI RR, ABED S, DRAPER JS. Organoids as a model system for studying human lung development and disease. Biochem Biophys Res Commun. 2016;473(3):675-682. [19] MORIZANE R, BONVENTRE JV. Kidney Organoids: A Translational Journey. Trends Mol Med. 2017;23(3):246-263. [20] SEINO T, KAWASAKI S, SHIMOKAWA M, et al. Human Pancreatic Tumor Organoids Reveal Loss of Stem Cell Niche Factor Dependence during Disease Progression. Cell Stem Cell. 2018;22(3):454-467.e6. [21] CARRANZA-ROSALES P, GUZMÁN-DELGADO NE, CARRANZA-TORRES IE,et al. Breast Organotypic Cancer Models. Curr Top Microbiol Immunol. 2018 Mar 20. doi: 10.1007/82_2018_86. Online ahead of print. [22] KASAGI Y, CHANDRAMOULEESWARAN PM, WHELAN KA, et al. The Esophageal Organoid System Reveals Functional Interplay Between Notch and Cytokines in Reactive Epithelial Changes. Cell Mol Gastroenterol Hepatol. 2018;5(3):333-352. [23] MAZZUCCHELLI S, PICCOTTI F, ALLEVI R, et al. Establishment and Morphological Characterization of Patient-Derived Organoids from Breast Cancer. Biol Proced Online. 2019;21:12. [24] TUVESON D, CLEVERS H. Cancer modeling meets human organoid technology. Science. 2019;364(6444):952-955. [25] VLACHOGIANNIS G, HEDAYAT S, VATSIOU A, et al. Patient-derived organoids model treatment response of metastatic gastrointestinal cancers. Science. 2018;359(6378):920-926. [26] SATO T, STANGE DE, FERRANTE M, et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett’s epithelium. Gastroenterology. 2011;141(5):1762-1772. [27] BOJ SF, HWANG CI, BAKER LA, et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 2015;160(1-2):324-338. [28] PARK JW, LEE JK, PHILLIPS JW, et al. Prostate epithelial cell of origin determines cancer differentiation state in an organoid transformation assay. Proc Natl Acad Sci U S A. 2016;113(16):4482-4487. [29] BROUTIER L, MASTROGIOVANNI G, VERSTEGEN MM, et al. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nat Med. 2017;23(12):1424-1435. [30] FRANCIES HE, GARNETT MJ. What role could organoids play in the personalization of cancer treatment? Pharmacogenomics. 2015; 16(14):1523-1526. [31] ASTASHKINA A, GRAINGER DW. Critical analysis of 3-D organoid in vitro cell culture models for high-throughput drug candidate toxicity assessments. Adv Drug Deliv Rev. 2014;69-70:1-18. [32] ZHANG L, ZHAO J, LIANG C, et al. A novel biosensor based on intestinal 3D organoids for detecting the function of BCRP. Drug Deliv. 2017;24(1):1453-1459. [33] KARKI R, MAN SM, MALIREDDI RKS, et al. NLRC3 is an inhibitory sensor of PI3K-mTOR pathways in cancer. Nature. 2016;540(7634):583-587. [34] YOSHIDA T, SOPKO NA, KATES M, et al. Three-dimensional organoid culture reveals involvement of Wnt/β-catenin pathway in proliferation of bladder cancer cells. Oncotarget. 2018;9(13):11060-11070. [35] GAO D, VELA I, SBONER A, et al. Organoid cultures derived from patients with advanced prostate cancer. Cell. 2014;159(1):176-187. [36] DATE S, SATO T. Mini-gut organoids: reconstitution of the stem cell niche. Annu Rev Cell Dev Biol. 2015;31:269-289. [37] SACHS N, DE LIGT J, KOPPER O, et al. A Living Biobank of Breast Cancer Organoids Captures Disease Heterogeneity. Cell. 2018;172(1-2): 373-386.e10. [38] ROSKOSKI R JR. The ErbB/HER family of protein-tyrosine kinases and cancer. Pharmacol Res. 2014;79:34-74. [39] LEE CY, LIN Y, BRATMAN SV, et al. Neuregulin autocrine signaling promotes self-renewal of breast tumor-initiating cells by triggering HER2/HER3 activation. Cancer Res. 2014;74(1):341-352. [40] DROST J, CLEVERS H. Organoids in cancer research. Nat Rev Cancer. 2018;18(7):407-418. [41] 刘宏飞,陈晓红,黄志刚,等.类器官和人源性肿瘤组织异种移植模型在肿瘤研究中的应用[J].中国比较医学杂志,2019,29(3): 103-108. [42] WEEBER F, OOFT SN, DIJKSTRA KK, et al. Tumor Organoids as a Pre-clinical Cancer Model for Drug Discovery. Cell Chem Biol. 2017; 24(9): 1092-1100. [43] DAI X, XIANG L, LI T, et al. Cancer Hallmarks, Biomarkers and Breast Cancer Molecular Subtypes. J Cancer. 2016;7(10):1281-1294. [44] NAYAK B, BALACHANDER GM, MANJUNATH S, et al. Tissue mimetic 3D scaffold for breast tumor-derived organoid culture toward personalized chemotherapy. Colloids Surf B Biointerfaces. 2019;180:334-343. [45] COE BP, WITHERSPOON K, ROSENFELD JA, et al. Refining analyses of copy number variation identifies specific genes associated with developmental delay. Nat Genet. 2014;46(10):1063-1071. [46] GOSSAGE L, EISEN T, MAHER ER. VHL, the story of a tumour suppressor gene. Nat Rev Cancer. 2015;15(1):55-64. [47] ROELOFS C, HOLLANDE F, REDVERS R, et al. Breast tumour organoids: promising models for the genomic and functional characterisation of breast cancer. Biochem Soc Trans. 2019;47(1):109-117. [48] SCHMIDT M, THOMSSEN C, UNTCH M. Intrinsic Subtypes of Primary Breast Cancer--Gene Expression Analysis. Oncol Res Treat. 2016;39(3): 102-110. [49] ALI HR, RUEDA OM, CHIN SF, et al. Genome-driven integrated classification of breast cancer validated in over 7,500 samples. Genome Biol. 2014;15(8):431. [50] KOLEDOVA Z. 3D Coculture of Mammary Organoids with Fibrospheres: A Model for Studying Epithelial-Stromal Interactions During Mammary Branching Morphogenesis. Methods Mol Biol. 2017;1612:107-124. [51] CHATTERJEE S, BHAT V, BERDNIKOV A, et al. Paracrine Crosstalk between Fibroblasts and ER+ Breast Cancer Cells Creates an IL1β-Enriched Niche that Promotes Tumor Growth. iScience. 2019;19: 388-401. [52] UMESH V, RAPE AD, ULRICH TA, et al. Microenvironmental stiffness enhances glioma cell proliferation by stimulating epidermal growth factor receptor signaling. PLoS One. 2014;9(7):e101771. [53] BUESS M, NUYTEN DS, HASTIE T, et al. Characterization of heterotypic interaction effects in vitro to deconvolute global gene expression profiles in cancer. Genome Biol. 2007;8(9):R191. [54] PARK SE, GEORGESCU A, HUH D. Organoids-on-a-chip. Science. 2019; 364(6444):960-965. [55] SONTHEIMER-PHELPS A, HASSELL BA, INGBER DE. Modelling cancer in microfluidic human organs-on-chips. Nat Rev Cancer. 2019;19(2):65-81. [56] CHEN MB, WHISLER JA, FRÖSE J, et al. On-chip human microvasculature assay for visualization and quantification of tumor cell extravasation dynamics. Nat Protoc. 2017;12(5):865-880. [57] SHIRURE VS, BI Y, CURTIS MB, et al. Tumor-on-a-chip platform to investigate progression and drug sensitivity in cell lines and patient-derived organoids. Lab Chip. 2018;18(23):3687-3702. [58] WALSH AJ, COOK RS, SANDERS ME, et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Res. 2014;74(18):5184-5194. [59] DEKKERS JF, WHITTLE JR, VAILLANT F, et al. Modeling Breast Cancer Using CRISPR-Cas9-Mediated Engineering of Human Breast Organoids. J Natl Cancer Inst. 2020;112(5):540-544. [60] PARK S, BRUGIOLO M, AKERMAN M, et al. Differential Functions of Splicing Factors in Mammary Transformation and Breast Cancer Metastasis. Cell Rep. 2019;29(9):2672-2688.e7. [61] CHEUNG KJ, GABRIELSON E, WERB Z, et al. Collective invasion in breast cancer requires a conserved basal epithelial program. Cell. 2013; 155(7):1639-1651. [62] ZUMWALDE NA, HAAG JD, SHARMA D, et al. Analysis of Immune Cells from Human Mammary Ductal Epithelial Organoids Reveals Vδ2+ T Cells That Efficiently Target Breast Carcinoma Cells in the Presence of Bisphosphonate. Cancer Prev Res (Phila). 2016;9(4):305-316. [63] 吴宇琪.乳腺癌的体外类器官培养和个性化药物选择[D].北京:北京协和医学院中国医学科学院;清华大学医学部;北京协和医学院, 2018. [64] QU Y, HAN B, GAO B, et al. Differentiation of Human Induced Pluripotent Stem Cells to Mammary-like Organoids. Stem Cell Reports. 2017;8(2):205-215. [65] CHOU JL, SHEN ZX, STOLFI RL, et al. Effects of extracellular matrix on the growth and casein gene expression of primary mouse mammary tumor cells in vitro. Cancer Res. 1989;49(19):5371-5376. [66] YOSHIDA GJ. Applications of patient-derived tumor xenograft models and tumor organoids. J Hematol Oncol. 2020;13(1):4. |
| [1] | Pu Rui, Chen Ziyang, Yuan Lingyan. Characteristics and effects of exosomes from different cell sources in cardioprotection [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(在线): 1-. |
| [2] | Lin Qingfan, Xie Yixin, Chen Wanqing, Ye Zhenzhong, Chen Youfang. Human placenta-derived mesenchymal stem cell conditioned medium can upregulate BeWo cell viability and zonula occludens expression under hypoxia [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(在线): 4970-4975. |
| [3] | Zhang Xiumei, Zhai Yunkai, Zhao Jie, Zhao Meng. Research hotspots of organoid models in recent 10 years: a search in domestic and foreign databases [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(8): 1249-1255. |
| [4] | Wang Zhengdong, Huang Na, Chen Jingxian, Zheng Zuobing, Hu Xinyu, Li Mei, Su Xiao, Su Xuesen, Yan Nan. Inhibitory effects of sodium butyrate on microglial activation and expression of inflammatory factors induced by fluorosis [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1075-1080. |
| [5] | Wang Xianyao, Guan Yalin, Liu Zhongshan. Strategies for improving the therapeutic efficacy of mesenchymal stem cells in the treatment of nonhealing wounds [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1081-1087. |
| [6] | Liao Chengcheng, An Jiaxing, Tan Zhangxue, Wang Qian, Liu Jianguo. Therapeutic target and application prospects of oral squamous cell carcinoma stem cells [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1096-1103. |
| [7] | Xie Wenjia, Xia Tianjiao, Zhou Qingyun, Liu Yujia, Gu Xiaoping. Role of microglia-mediated neuronal injury in neurodegenerative diseases [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1109-1115. |
| [8] | Li Shanshan, Guo Xiaoxiao, You Ran, Yang Xiufen, Zhao Lu, Chen Xi, Wang Yanling. Photoreceptor cell replacement therapy for retinal degeneration diseases [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1116-1121. |
| [9] | Wang Shiqi, Zhang Jinsheng. Effects of Chinese medicine on proliferation, differentiation and aging of bone marrow mesenchymal stem cells regulating ischemia-hypoxia microenvironment [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1129-1134. |
| [10] | Zeng Yanhua, Hao Yanlei. In vitro culture and purification of Schwann cells: a systematic review [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1135-1141. |
| [11] | Kong Desheng, He Jingjing, Feng Baofeng, Guo Ruiyun, Asiamah Ernest Amponsah, Lü Fei, Zhang Shuhan, Zhang Xiaolin, Ma Jun, Cui Huixian. Efficacy of mesenchymal stem cells in the spinal cord injury of large animal models: a meta-analysis [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1142-1148. |
| [12] | Hou Jingying, Yu Menglei, Guo Tianzhu, Long Huibao, Wu Hao. Hypoxia preconditioning promotes bone marrow mesenchymal stem cells survival and vascularization through the activation of HIF-1α/MALAT1/VEGFA pathway [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 985-990. |
| [13] | Shi Yangyang, Qin Yingfei, Wu Fuling, He Xiao, Zhang Xuejing. Pretreatment of placental mesenchymal stem cells to prevent bronchiolitis in mice [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 991-995. |
| [14] | Liang Xueqi, Guo Lijiao, Chen Hejie, Wu Jie, Sun Yaqi, Xing Zhikun, Zou Hailiang, Chen Xueling, Wu Xiangwei. Alveolar echinococcosis protoscolices inhibits the differentiation of bone marrow mesenchymal stem cells into fibroblasts [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 996-1001. |
| [15] | Fan Quanbao, Luo Huina, Wang Bingyun, Chen Shengfeng, Cui Lianxu, Jiang Wenkang, Zhao Mingming, Wang Jingjing, Luo Dongzhang, Chen Zhisheng, Bai Yinshan, Liu Canying, Zhang Hui. Biological characteristics of canine adipose-derived mesenchymal stem cells cultured in hypoxia [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(7): 1002-1007. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||