Chinese Journal of Tissue Engineering Research ›› 2013, Vol. 17 ›› Issue (24): 4529-4538.doi: 10.3969/j.issn.2095-4344.2013.24.023
Previous Articles Next Articles
Molecular mechanism of spinal cord injury detected using microarray technology
Xi Yue
- School of Biological Science and Medical Engineering, Beihang University, Beijing 100191, China
-
Received:2012-10-17Revised:2012-11-27Online:2013-06-11Published:2013-06-11 -
About author:Xi Yue★, Studying for master’s degree, School of Biological Science and Medical Engineering, Beihang University, Beijing 100191, China jackson@be.buaa.edu.cn
CLC Number:
Cite this article
Xi Yue. Molecular mechanism of spinal cord injury detected using microarray technology[J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4529-4538.
share this article
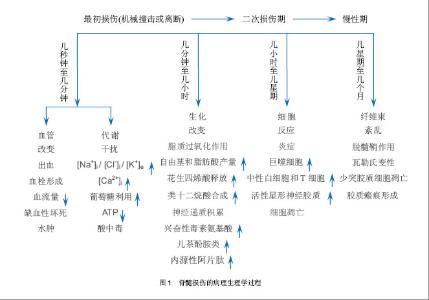
外伤性脊髓损伤在损伤后致成病理生理的变化会从几分钟延伸至几年[1]。脊髓损伤后的病理生理学反应可分为3个时期:急性期、二次损伤期(次生组织损伤期)及慢性期[2]。有的将脊髓损伤分为原发性和继发性。 2.1.1 急性期 急性期开始于损伤时刻至最初的两三天,这期间很多病理生理学过程启动。如机械性损伤导致的神经元损伤、传导束断裂属于直接损伤。进一步导致血流量减少造成缺血性坏死[3]。大脑和脊髓的创伤性损伤可导致血脑屏障的破坏,在脊髓损伤时,血脊髓屏障可受到破坏,改变中枢神经系统正常内皮的选择通透性,富含蛋白的血浆样液体在血管外间隙聚集,形成水肿。代谢紊乱可发生包括神经元内Ca2+堆积和细胞外K+浓度增加[1]。有假说认为,脊髓损伤时水肿的形成之所以有害,是因为其可导致神经组织的压迫和电解质环境的紊乱。急性期可看成一个标准神经功能的总体缺失及脊髓休克阶段[4]。代谢方面,Rawe等[5]报道狗在中度脊髓损伤1 h后,白质和灰质的葡萄糖利用增加。随后,灰质的葡萄糖利用逐渐下降,持续3-8 h后又恢复到基线水平。 2.1.2 二次损伤期 在二次损伤期,自由基产量增加。脊髓损伤和创伤后缺血将消耗ATP使AMP的产量增加,并降解为次黄嘌呤。自由基过量的病理状态可导致脂类过氧化和细胞膜的损伤。已有实验证明,自由基的产生及其有害作用参与了中枢神经系统缺血的病理生理过程。二次损伤同时会使兴奋性神经递质过度释放,发生炎症反应。脊髓损伤有炎性成分的参与,包括炎症细胞的浸润和炎性递质的释放。除此之外可观察到大块的缺血性坏死和大量细胞死亡。星形胶质细胞的大小和胶质纤维酸性蛋白阳性程度在活性神经胶质增生阶段有所增加。脊髓的外伤性损伤同样会导致强烈的炎症应答,伴随周围衍生的免疫细胞如中性白细胞(6-24 h)、巨噬细胞(24 h-2周)和T细胞的募集反应[6-7]。 2.1.3 慢性期 慢性期(损伤后几天到几年)的变化主要是细胞持续性死亡,通道和受体功能被损坏,形成瘢痕和脱髓鞘作用,并伴随瓦勒氏变性,这些过程促成了传导障碍[8]。最近有证据表明,肿瘤坏死因子参与脊髓继发性损伤的病理生理变化过程。脊髓损伤后可检测到肿瘤坏死因子及其mRNA的升高。这一细胞因子可刺激星形胶质细胞增生、肥大,从而导致神经胶质瘢痕的形成。它的具体作用是双向的,因为它可通过增强血管生成作用和诱导生长因子的表达而促进神经的修复和再生。 2.1.4 形态学改变 脊髓损伤几分钟内,就可出现各种形态学的改变。这包括灰质的出血瘀斑、小静脉壁的破裂、灰质和白质细胞外间隙的增大、髓鞘的变薄、轴突周间隙增大、轴浆内陷和轴浆破裂。脊髓受伤1 h后的主要变化是染色质溶解,腹侧前角细胞开始出现缺血。4 h出血性改变向四周蔓延并融合。急性破坏损伤多位于中心区,依赖于损伤的严重程度,将会缓慢地累及邻近的白质。轴突破碎变化将继发于轴突或轴突周围肿胀。轴突增大包括多个细胞器(线粒体、神经微丝、溶酶体和平滑内织网),Bresnahan等[9]将其归咎于轴浆淤滞。免疫应答方面,最初可出现急性多形核白细胞的浸润,伤后几天变为巨噬细胞的浸润。伤后第1周坏死区可出现囊性退变,4周后囊性变更明显,残存的白质表现出脱髓鞘作用和微囊形成。4个月后,囊肿被星形神经胶质细胞包围,损伤区出现硬膜增厚。在损伤较重的部位软脊膜细胞构成的增加也很明显。 虽然广泛的研究旨在更好的了解脊髓损伤的病理生理学过程,但目前的研究成果还不能更加完全的阐明其详细分子机制。鉴于大量的生物化学级联及细胞反应,需要用一种高准确性、高通量分析来阐明相关现象。近来在基因组学水平上的技术进步使这种方法成为可能。 2.2 利用微阵列芯片技术观察脊髓损伤后的基因表达变化 在最近的研究中,脊髓损伤后的mRNA表达谱表现出有变化的特征。一些研究证实基因表达的变化在损伤后几分钟到几天内随着时间的发展而加剧[10-13],更多的研究则聚焦于观察到的仅一个变化非常剧烈或具有特异性的时间点上[14-18]。 2.2.1 脊髓损伤后急性期内的基因表达变化 一些研究调查了成年大鼠或小鼠脊髓在损伤后从30 min到6 h的基因表达变化,在损伤的急性期有4个主要类型的基因表达有持续改变。首先,在早期时间点炎症相关分子中的转录本有变化。到损伤后30 min出现环氧化酶2的强烈上调(3倍),在4 h时强化到4倍。促炎细胞因子白细胞介素1β、白细胞介素6和白细胞介素受体4R/2Ra的表达水平在脊髓损伤后30 min和6 h时提高了3到9倍[10,12,14,18]。最近研究表明脊髓损伤后细胞外谷氨酸盐的升高能促进细胞的死亡和功能减退,而白细胞介素1β介导了脊髓损伤后谷氨酸盐的损害作用。这提示白细胞介素1浓度的升高加速了细胞的死亡和功能减退,阻断白细胞介素1β可能减轻脊髓继发性损伤。Mukaino等[19]报道白细胞介素6过度表达引起脊髓继发性损伤的同时也促进神经干细胞向神经胶质细胞分化。最近有研究发现白细胞介素6通过Erk1/2介导JAK-STAT通路刺激脊髓前体细胞增殖,并促进伤后脊髓组织功能恢复,为白细胞介素6在脊髓损伤后的保护作用机制提供了新的思路,但白细胞介素6在脊髓损伤后的致炎性作用机制尚不清楚。先前Pan等[20]用RNA酶保护测定白细胞介素1β和白细胞介素6,Bartholdi和Schwab[21]利用原位杂交测定白细胞介素1β都得到相同数据。传统测定技术与微阵列技术相结合是必要的,这样能够证明微阵列数据的准确性。 早期时间点的抗炎细胞因子(白细胞介素10、白细胞介素13或白细胞介素4)没有发现表达上的修饰。除了触发炎症应答之外,低浓度的促炎症反应分子如白细胞介素1β和白细胞介素6是有益的[22],但如果是高浓度就同样会活化转录因子如AP-1、核因子κB,这样就会介导细胞死亡[23]。实际上,强烈上调的尤其是与细胞损伤和死亡相关的转录因子,如NF-kB、c-jun和SOCS-3会发生在脊髓损伤的急性期 中[12,15,18]。这与先前报道的利用原位杂交和免疫组织化学技术得到的结果相一致[24-25]。强烈过表达(7倍[14])的转录因子JAK和信号转导蛋白及STAT家族转录本的激活可能表示了一个早期的脊髓修复和再生行为[26-28]。 2.2.2 脊髓损伤后二次损伤期的基因表达变化 在急性期观察到的一些持续变化在以后的几天中进一步提高。这个现象明显表现在即早基因或促炎症反应调节物如白细胞介素1β或环氧化酶2中。同时观察到离子通道和神经递质受体及转运蛋白在1-3 d 之间持续下调。特别的是,谷氨酸盐的几个亚基(AMPA和促代谢型)的受体、GABA受体和甘氨酸受体的轻微下调在急性期进一步缩小[12,16-18]。转运蛋白如Na+、K+、Na+/Ca2+和Na+/HCO-中也有相同的模式[17-18]。这很可能是神经递质受体和转运蛋白的下调促成了长期的神经元损伤,破坏了突触传递。二次损伤时期同样存在突触分子如Rab3、SNAP-25A和SNAP-25B、突触蛋白2A、突触蛋白2B和突触融合蛋白B的表达降低,这些蛋白都是突触分泌装置的部件,参与囊泡融合及神经递质的释放[29-30]。这是一个突触功能的主要损坏。 在脊髓损伤后12 h之前神经微丝蛋白NFL和NFH、tau和微管相关蛋白2有强烈下调,这证实了先前的研究利用传统技术如免疫组织化学显示损伤后细胞骨架蛋白损失的真实性[31]。髓鞘少突胶质细胞糖蛋白MOG的表达同样降低[12],指示了髓磷脂合成的障碍及可能的脱髓鞘作用[32-33]。Nogo-A的表达在损伤中心丧失[34],但是在周围组织中不会改变[35];然而,被损坏的髓磷脂层同样可以释放这样的蛋白质类活性片段,进一步抑制轴突生长[36]。硫酸软骨素蛋白聚糖、主要轴突生长抑制因子的表达水平在脊髓损伤后损伤区周围的瘢痕组织中升高,这就限制了脊髓进一步的再生行为[37]。所有这些变化反映了在脊髓损伤后的12-72 h内,脊髓的完整性高度的缺乏。然而在24-48 h内,生长相关分子和生长因子开始表达,显示了损伤的中枢神经系统再生和继续生长的行为。在24 h之前,可探测到生长因子的上调如可诱导的神经生长因子(8倍增加)、脑源性神经营养因子(3倍增加)、血小板衍生生长因子、骨形态发生蛋白质类、运动神经元存活因子和成纤维细胞生长因子受体1(5倍增加)。在48 h,他们的表达进一步增加,伴随胰岛素样生长因子1和胰岛素样生长因子2均上调[11,14,18]。在48 h之前神经再生相关分子(如发动蛋白)和黏附分子(如VCAM和F3)有上调[10,16],而nestin有10倍上调[12],这或许反映了神经前体细胞的增殖[38]。 2.2.3 脊髓损伤7 d及之后的基因表达变化 很少的研究者观察到脊髓损伤后的慢性期中有转录本的变化。脊髓损伤后7 d,炎症和氧化性应激基因,如编码白细胞抗原MRC-OX44基因、补体蛋白C1q、主要组织相容性复合物(MHC)I和II类及白细胞介素6依然上调[10]。一些朊酶类的表达增加,多数可能会促成进一步二次组织损伤或组织重塑。血管发生和神经发生基因开始强烈表达,反映了脊髓修复自身组织的行为。在一个选择性部分损伤的特殊例子中,Bareyre等[11]用椎体束切断术切断皮质脊髓束,发现去神经支配后的1周,脊髓表现出来生长因子和受体(如trkB:25倍、血管内皮生长因子受体和脑源性神经营养因子)、轴突导向分子(如Sema3和slit1)、胞外基质蛋白(如核心蛋白聚糖、基膜聚糖和胶原)的显著上调。在大鼠慢性脊髓损伤中,Wnt信号、雌激素受体、胰岛素样生长因子1及成骨细胞中的骨形态生成蛋白信号通路均出现下调[39]。总的来说,轴突导向、神经元存活以及轴突生长相关分子的上调证明了脊髓适应损伤和轴突再生的努力。即使就面积而言是有限的,至少对于一些类型的轴突来说这些生长的进程或许是有靶向性和非随机性的,在这个时间里可以促成功能上的恢复。 2.3 实验设计及微阵列芯片检测技术上的革新 虽然微阵列芯片分析提供了重要的新的洞察脊髓损伤病理生理学的方法,但在很多研究的数据分析结果中,都存在数据的不一致,即数据的可重复性低。不充分的实验设计因素导致输出数据集的偏差是微阵列在技术上的不幸后果之一[40]。因此生物学重复、芯片标记、检测、图像扫描、比率计算和数据标准化的合理设计至关重要。另外的一个原因是实验动物模型制作方法没有一个统一的标准,不同的损伤模型、动物物种、性别影响了结果的统一性和重复性。如在大鼠脊髓损伤模型中,选用成年雄性Wistar大鼠与成年雌性SD大鼠、脊髓半段、全断或砸伤得到的结果必然不同。因为损伤的本质(包括挫伤、切断或切除)极其严重性影响了脊髓损伤的病理生理特征和基因表达。因此一个好的标准化的实验条件决定了将来的研究成果。目前国际上只有少数几例动物模型可作为标准。另外,芯片本身的质量也是决定性的因素之一。微阵列芯片近几年来在其制作方法和检测精确程度上也有所突破,从最初的被动式生物芯片的制作方法发展到利用光引导原位合成技术制备核酸芯片,现已出现基于微阵列芯片的微缩芯片实验室和主动式生物芯片(如电磁芯片),它具有分析灵敏度高、样品分析时间大为缩短等特点,是生物芯片发展的大势所趋[41]。 在检测方法上,最常用的是荧光显微和激光共聚焦扫描检测法。目前还出现了CCD成像扫描检测、表面等离子共振吸收检测(SPR)及金胶银染检测法、薄膜技术检测法。这些新方法克服了前者的不足,提高了杂交信号检测的准确性、灵敏性和速度。随着生物芯片技术的发展与应用需求的多样化,新的生物芯片检测技术还在出现,而相关仪器设备则以小型化与集成化发展为特征[41]。 2.4 数据分析方法上的改进 随着芯片技术的发展,芯片实验成本逐渐降低。很多芯片实验都会采用重复芯片的策略,如何从重复芯片中识别差异表达基因,是现在芯片研究的一个重要的课题。 在数据分析中,差异程度的标准化起到决定性作用。许多研究选择2倍的差异是显著的界限。然而这个选择会导致丢失大量的相同表达变化基因及未剔除假阳性结果。杂交信号大于400、变化倍数在1.5倍以上且显著性系数p值<0.05相结合是较合理的一种筛选方式。这样能保留在脊髓损伤修复过程中可能参与并尚未被发现的基因。 另外目前广泛使用的一些数据分析软件如Affymetrix GCOS系统、GeneSpring、MATLAB生物信息工具箱、基因芯片图像分析软件GenePix Pro、SpotFire DecisionSuite、SAM及Cluster和Tree View程序可帮助研究者们简单、快速、高效的完成数据采集、数据比对、差异挑选、聚类分析、图像处理等工作。差异基因功能注释分析可通过网络公共数据库信息(如NCBI、KEGG、EBI ArrayExpress和SMD等)或网络基因注释数据库(如DAVID系统、mas3.0系统等)实现[42]。在基因网络分析中,目前常用的方法是权重基因共表达网络分析(Weighted Correlation Network Analysis, WGCNA),它是在R软件运行环境下建立的一个软件包,由很多分析程序组成。其中包括分析基因共表达网络、定义模块、分析相关模块与外部信息、分析模块相关性、感兴趣模块中驱动基因的寻找这几个方面的程序及软件。其作用包括:寻找高度相关基因的聚类(模块);通过模块本征基因或内部中心基因来概括模块特征;分析模块与另一个模块及外部样本之间的关联特征(运用本征基因网络方法学);计算模块从属关系的程度[43]。近年来出现的新方法也为改进数据分析提供了思路,如Ujjwal[44]提出用软计算结构(包括遗传算法、模拟退火算法和差异演化算法)来分析基因微阵列数据,José等[45]利用一个基因表达数据聚类的可视化分析学结构,提出了寻找基于距离空间性质的聚类边界的新方法。 处理微阵列数据时的一个重要内容是质量和精确性分析,目前国际上通常用适当的补充研究来证明观察到的变化。感兴趣基因的差异表达应该用典型方法在独立样本中证实,尤其是利用非严谨杂交分析或只发现轻微表达变化时,更需验证其真实性。一些研究利用定量RT-PCR或核糖核酸酶保护测定(RPA)来证实他们的数据。Western blot和免疫组织化学同样被用来测量相应蛋白水平,而原位杂交用来确定区域及细胞类型特异性的mRNA,见图2。 "

2.5 对微阵列芯片帮助寻找有效治疗靶点、改良脊髓损伤治疗方法的能力评价 微阵列芯片检测数据经过程序筛选和聚类分析后,可得到差异表达显著的基因。将基因以其所在信号通路分类,就能推测促进神经再生的分子机制。反过来寻找差异表达最显著的一些基因,就能找到可能的治疗学靶点。 在周围神经系统中,轴突可以通过长距离有效再生,重建正确的联合。但中枢神经系统的轴突再生和功能修复是有限制的[1]。微阵列、差异筛选和免疫组织化学研究显示在轴突切断术后背根神经节神经元不仅上调即早基因和热休克蛋白,而且上调神经突生长和再生相关基因(编码GAP-43、CAP-23和Sprrp1A) [47-49]。在尝试识别外周和中枢神经损伤的不同调节基因时,Fan等[16]比较了小鼠的坐骨神经损伤和脊髓损伤。他们鉴定了对侧方位的一组基因的变化;在这些基因中有转录因子和受体及细胞黏附分子。Armin等[50]探究了在脊髓损伤前后环单磷酸腺苷(cAMP)介导的外周神经轴突再生效应,利用微阵列芯片技术检测了腰段背根神经节中的基因表达变化,发现在条件性损伤后1 d就有733个探针表现上调,497个表现下调。Colak等[51]发现在外伤性脑损伤后 1 h、12 h和48 h,C1ql2、Cbnl、Sdc1、Bdnf、MMP9和Cd47这些基因均出现显著差异表达。更广泛的比较(如时间进程研究)或许能给出感兴趣的关键发现,有助于进一步了解外周和中枢神经系统再生的不同进程。 轴突生长抑制因子在中枢神经系统再生的背景下起到了至关重要的作用[1]。一个增加脊髓损伤后再生和提供允许的微环境的方法是中和或阻止这些抑制因子。一些髓磷脂相关抑制因子已被鉴定,其中有NI-250(现在被称为Nogo-A) [50]。这个分子具有离体情况下防止神经纤维生长的能力[51]。在体内其抑制效应的证据最初来自在成年大鼠脊髓损伤后利用抗体IN-1抵消髓磷脂生长抑制因子的实验,由此可增强轴突再生、补偿备用纤维的可塑性以及显著改善运动功能。为阐明施用IN-1抗体后组织的反应,Bareyre等[11]利用了Affymetrix微阵列芯片来检测。在没有任何损伤的条件下,IN-1控制上调了细胞骨架蛋白(actin和肌球蛋白)、Ca2+结合蛋白、炎症相关基因和胶原蛋白。延髓水平皮质脊髓束中断配以IN-1治疗,可观察到颈部脊髓通过上调细胞骨架蛋白类(肌动蛋白、肌球蛋白、肌原蛋白和微管相关蛋白2)、一个生长相关蛋白(GAP-43)、一些生长因子(脑源性神经营养因子,血管内皮生长因子,trkB,胰岛素样生长因子1与胰岛素样生长因子2,胶质细胞源性神经营养因子、胶质成熟作用因子和内皮素受体)、转录因子(STATs、MRP14)、轴突导向分子(Sema3a、Slit)、细胞外基质和膜蛋白(神经细胞黏附分子NCAM、F3、淀粉样前蛋白APP、胶原蛋白、层粘连蛋白和TIMP1)以及突触蛋白(囊泡相关膜蛋白1、突触蛋白3a)的mRNA来起作用[11]。 在损害控制和神经保护方面,Nesic等[14]研究了MK801的效果,它是一个NMDA受体阻断剂。报道在大鼠脊髓损伤后注射MK801一个小时可以逆转脊髓损伤的效果,改变约50%脊髓损伤影响的基因。特别的是,他们发现MK801减弱了脊髓损伤对于细胞因子类(如白细胞介素6、β型转化生长因子和胰岛素样生长因子1)、细胞死亡因子(caspase1或Bad)、谷氨酸受体(NMDAR-2D1、GLUR-C和GLUR6)的上调的效果,同样损伤脊髓的Ca2+和K+通道也通过施加MK801下调。利用微阵列芯片检测到差异表达的转录因子Sox11b被证明能够促进成体脊髓再生性神经发生与运动恢复[54],而在小鼠脊髓损伤后7d到28d的慢性期中,炎症相关基因C1qB、p22phox和galectin-3显示出持续性的表达高峰,而galectin-3或许会促成二次损伤[55]。 目前广泛接受的治疗人类脊髓损伤的药理学疗法是运用甲泼尼龙,它是一种皮质类固醇。在损伤后6-8 h内使用大剂量使用会诱导一个轻微的功能恢复的改善。Almon等[56]进行了一个cDNA微阵列芯片检测甲泼尼龙用于大鼠的实验。当给予快速灌注方式注射时,它升高了炎症相关基因如初前补体C、MHCI类及凝血酶的表达,同时增加了a1和a2巨球蛋白类(可抑制炎症性蛋白酶)的表达。这些抗炎作用暗示了甲泼尼龙在脊髓损伤患者的临床效治疗中似乎起到一定作用。除此之外,利用生物材料修复脊髓损伤也是当今神经再生基础研究的热点。但无论是何种治疗方法,都需要更多脊髓损伤后分子生物学机制的研究,不仅在治疗方法和效果上要有创新和突破,更需阐明促进神经再生是通过激活或抑制哪些基因或分子信号通路来起到作用。"

| [1] Schwab ME, Bartholdi D. Degeneration and regeneration of axons in the lesioned spinal cord. Physiol Rev. 1996; 76(2): 319-370.http://www.ncbi.nlm.nih.gov/pubmed/8618960[2] Hulsebosch CE. Recent advances in pathophysiology and treatment of spinal cord injury. Adv. Physiol Educ. 2002; 26(4): 238-255.http://www.ncbi.nlm.nih.gov/pubmed/12443996 [3] Sandler AN,Tator CH. Review of the effect of spinal cord trauma on the vessels and blood flow in the spinal cord. J Neurosurg. 1976; 45(3): 638-646.http://www.ncbi.nlm.nih.gov/pubmed/824416 [4] Bach-y-Rita P, Illis LS. Spinal shock: possible role of receptor plasticity and non synaptic transmission. Paraplegia.1993; 31(2): 82-87.http://www.ncbi.nlm.nih.gov/pubmed/8383312[5] Rawe SE, Lee WA, Perot PL. Spinal cord glucose utilization after experimental spinal cord injury. Neurosurgery. 1981; 9(1): 40-47.http://www.ncbi.nlm.nih.gov/pubmed/7279171 [6] Bethea JR, Dietrich WD. Targeting the host inflammatory response in traumatic spinal cord injury. Curr Opin Neurol. 2002; 15(3): 355-360.http://www.ncbi.nlm.nih.gov/pubmed/12045737[7] Schnell L, Fearn S, Klassen H, et al. Acute inflammatory responses to mechanical lesions in the CNS: differences between brain and spinal cord. Eur J Neurosci.1999; 11(10): 3648-3658.http://www.ncbi.nlm.nih.gov/pubmed/10564372 [8] Taoka Y, Okajima K. Spinal cord injury in the rat . Prog Neurobiol. 1998; 56(3): 341-358.http://www.ncbi.nlm.nih.gov/pubmed/9770243[9] Bresnahan JC, Beattie MS. Spinal cord injury: taking a detour to recovery (Commentary on Schnell et al.). Eur J Neurosci. 2011; 34(8): 1254-1255.http://www.ncbi.nlm.nih.gov/pubmed/?term=Spinal+cord+injury%3A+taking+a+detour+to+recovery [10] Di Giovanni S, Knoblach SM, Brandoli C, et al. Gene profiling in spinal cord injury shows role of cell cycle in neuronal death. Ann Neurol. 2003; 53(4): 454-468.http://www.ncbi.nlm.nih.gov/pubmed/12666113 [11] Bareyre FM, Haudenschild B, Schwab ME, et al. Long-lasting sprouting and gene expression changes induced by the monoclonal antibody IN-1 in the adult spinal cord. J Neurosci. 2002; 22(16): 7097-7110.http://www.ncbi.nlm.nih.gov/pubmed/12177206 [12] Carmel JB, Galante A, Soteropoulos P, et al.Gene expression profiling of acute spinal cord injury reveals spreading inflammatory signals and neuron loss. Physiol Genomics. 2001; 7(2): 201-213.http://www.ncbi.nlm.nih.gov/pubmed/11773606 [13] Miranda M, Christophe L, Elaine DB, et al. Developmental changes of gene expression after spinal cord injury in neonatal opossums. Brain Research. 2010; 1363: 20-39.http://www.ncbi.nlm.nih.gov/pubmed/20849836 [14] Nesic O, Svrakic NM, Xu GY, et al. DNA microarray analysis of the contused spinal cord: effect of NMDA receptor inhibition. J Neurosci Res. 2002; 68(4): 406-423.http://www.ncbi.nlm.nih.gov/pubmed/11992467 [15] Liu CL, Jin AM, Tong BH, et al. Detection of gene expression pattern in the early stage after spinal cord injury by gene chip. Chin J Traumatol. 2003; 6(1): 18-22.http://www.ncbi.nlm.nih.gov/pubmed/12542959 [16] Fan M, Mi RF, Yew DT, et al. Analysis of gene expression following sciatic nerve crush and spinal cord hemisection in the mouse by microarray expression profiling. Cell Mol Neurobiol. 2001; 21(5): 497-508.http://www.ncbi.nlm.nih.gov/pubmed/11860187 [17] Tachibana T, Noguchi K, Ruda MA. Analysis of gene expression following spinal cord injury in rat using complementary DNA microarray. Neurosci Lett. 2002; 327(2): 133-137.http://www.ncbi.nlm.nih.gov/pubmed/12098653 [18] Song GQ, Cechvala C, Resnick DK, et al. GeneChip analysis after acute spinal cord injury in rat. J Neurochem. 2001; 79(4): 804-815.http://www.ncbi.nlm.nih.gov/pubmed/11723173 [19] Mukaino M, Nakamura M, Okada S, et al. Role of IL-6 in regulation of inflammation and stem cell differentiation in CNS trauma. Jap J Clinical Immunology. 2008; 31(2): 93-98.http://www.ncbi.nlm.nih.gov/pubmed/18446011 [20] Pan JZ, Ni L, Sodhi A, et al. Cytokine activity contributes to induction of inflammatory cytokine mRNAs in spinal cord following contusion. J Neurosci Res. 2002; 68(3): 315-322.http://www.ncbi.nlm.nih.gov/pubmed/12111861 [21] Bartholdi D, Schwab ME. Expression of pro-inflammatory cytokine and chemokine mRNA upon experimental spinal cord injury in mouse: an in situ hybridization study. Eur J Neurosci. 1997; 9(7): 1422-1438.http://www.ncbi.nlm.nih.gov/pubmed/9240400 [22] Murphy PG, Borthwick LA, Altares M, et al. Reciprocal actions of interleukin-6 and brain-derived neurotrophic factor on rat and mouse primary sensory neurons. Eur J Neurosci. 2000; 12(6): 1891-1899.http://www.ncbi.nlm.nih.gov/pubmed/10886330 [23] Li X, Mairead C, Jiang ZF, et al. IL-1-induced NFkB and c-Jun N-terminal kinase (JNK) activation diverge at IL-1 receptor-associated kinase (IRAK). Proc Natl Acad Sci U S A. 2001; 98(8): 4461-4465.http://www.ncbi.nlm.nih.gov/pubmed/11287640[24] Hayashi M, Ueyama T, Nemoto K, et al. Sequential mRNA expression for immediate early genes, cytokines, and neurotrophins in spinal cord injury. J Neurotrauma 2000; 17(3): 203-218.http://www.ncbi.nlm.nih.gov/pubmed/10757326 [25] Schmitt AB, Breuer S, Voell M, et al. GAP-43 (B-50) and c-Jun are up-regulated in axotomized neurons of Clarke’s nucleus after spinal cord injury in the adult rat. Neurobiol Dis. 1999; 6(2): 122-130.http://www.ncbi.nlm.nih.gov/pubmed/10343327 [26] Suzuki S, Tanaka K, Nogawa S, et al. Phosphorylation of signal transducer and activator of transcription-3 (Stat3) after focal cerebral ischemia in rats. Exp Neurol. 2001; 170(1): 63-71.http://www.ncbi.nlm.nih.gov/pubmed/11421584 [27] Sheu JY, Kulhanek DJ, Eckenstein FP. Differential patterns of ERK and STAT3 phosphorylation after sciatic nerve transection in the rat. Exp Neurol. 2000; 166(2): 392-402.http://www.ncbi.nlm.nih.gov/pubmed/11085904 [28] Schwaiger FW, Horvat A, Hager G, et al. Peripheral but not central axotomy induces changes in Janus kinases (JAK) and signal transducers and activators of transcription (STAT) . Eur J Neurosci. 2000; 12(4): 1165-1176.http://www.ncbi.nlm.nih.gov/pubmed/10762348[29] Grabs D, Bergmann M, Urban M, et al. Rab3 proteins and SNAP-25, essential components of the exocytosis machinery in conventional synapses, are absent from ribbon synapses of the mouse retina. Eur J Neurosci. 1996; 8(1): 162-168.http://www.ncbi.nlm.nih.gov/pubmed/8713460 [30] Porton B, Kao HT, Greenqard P, et al. Cloning of cDNAs encoding human synapsins IIa and IIb. DNA Seq. 1999;10: 49-54.http://www.ncbi.nlm.nih.gov/pubmed/10565545 [31] Zhang SX, Underwood M, Landfield A, et al. Cytoskeletal disruption following contusion injury to the rat spinal cord. J Neuropathol Exp Neurol. 2000; 59(4): 287-296.http://www.ncbi.nlm.nih.gov/pubmed/10759184 [32] Gledhill RF, Harrison BM, McDonald WI. Demyelination and remyelination after acute spinal cord compression. Exp Neurol. 1973; 38(3): 472-487.http://www.ncbi.nlm.nih.gov/pubmed/4696114 [33] Grossman SD, Rosenberg LJ, Wrathall JR. Temporal-spatial pattern of acute neuronal and glial loss after spinal cord contusion. Exp Neurol. 2001; 168(2): 273-282.http://www.ncbi.nlm.nih.gov/pubmed/11259115 [34] Josephson A, Widenfalk J, Widmer HW, et al. NOGO mRNA expression in adult and fetal human and rat nervous tissue and in weight drop injury. Exp Neurol. 2001; 169(2): 319-328.http://www.ncbi.nlm.nih.gov/pubmed/11358445 [35] Huber AB, Weinmann O, Brosamle C, et al. Patterns of Nogo mRNA and protein expression in the developing and adult rat and after CNS lesions. J Neurosci. 2002; 22(9):3553-3567.http://www.ncbi.nlm.nih.gov/pubmed/11978832 [36] Tang S, Qiu J, Nikulina E, et al. Soluble myelin-associated glycoprotein released from damaged white matter inhibits axonal regeneration. Mol Cell Neurosci. 2001; 18(3): 259-269.http://www.ncbi.nlm.nih.gov/pubmed/11591127 [37] Fawcett JW, Asher RA. The glial scar and central nervous system repair. Brain Res. Bull. 1999; 49(6): 377-391.http://www.ncbi.nlm.nih.gov/pubmed/10483914 [38] Horner PJ, Power AE, Kempermann G, et al. Proliferation and differentiation of progenitor cells throughout the intact adult rat spinal cord. J Neurosci. 2000;20(6): 2218-2228.http://www.jneurosci.org/content/20/6/2218.full.pdf [39] Jiang SD, Yan J, Jiang LS, et al. Down-regulation of the Wnt, estrogen receptor, insulin-like growth factor-I, and bone morphogenetic protein pathways in osteoblasts from rats with chronic spinal cord injury. Joint Bone Spine. 2011; 78(5): 488-492.http://www.ncbi.nlm.nih.gov/pubmed/21273111 [40] Tobias KK, Robert MF, Susan ED, et al. An introduction to DNA microarrays for gene expression analysis. Chemometrics and Intelligent Laboratory Systems. 2010; 104(1): 28-52.http://www.sciencedirect.com/science/article/pii/S0169743910000523[41] 程京, 黄国亮. 生物芯片:生命信息CPU [M]. 上海:少年儿童出版社, 2006. 247-249.http://www.tmall.com[42] 李瑶. 基因芯片数据分析与处理[M]. 北京:化学工业出版社, 2006. 295-307.http://download.cip.com.cn/ [43] Peter L, Steve H. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 2008; 9: 559.http://www.ncbi.nlm.nih.gov/pubmed/19114008 [44] Ujjwal M. Analysis of gene microarray data in a soft computing framework. Applied Soft Computing. 2011; 11(6): 4152-4160.http://libra.msra.cn/Publication/39311347/analysis-of-gene-microarray-data-in-a-soft-computing-framework [45] José AC, Carlos AG, Paulo N, et al. A visual analytics framework for cluster analysis of DNA microarray data. Expert Systems with Applications. 2012; 40(2): 758-744.http://www.sciencedirect.com/science/article/pii/S0957417412009992 [46] Jeffrey KH, Andrew AJ, Brahim NO, et al. Retinoid X receptor gamma signaling accelerates CNS remyelination. Nature Neurosci. 2011; 14: 45-53.http://www.ncbi.nlm.nih.gov/pubmed/21131950 [47] Costigan M, Befort K, Karchewski L, et al.Replicate high-density rat genome oligonucleotide microarrays reveal hundreds of regulated genes in the dorsal root ganglion after peripheral nerve injury. BMC Neurosci. 2002; 3: 16.http://www.ncbi.nlm.nih.gov/pubmed/?term=Replicate+high-density+rat+genome+oligonucleotide+microarrays+reveal+hundreds+of+regulated+genes+in+the+dorsal+root+ganglion+after+peripheral+nerve+injury [48] Bonilla IE, Tanabe K, Strittmatter SM. Small proline-rich repeat protein 1A is expressed by axotomized neurons and promotes axonal outgrowth. J Neurosci. 2002; 22(4): 1303-1315.http://www.ncbi.nlm.nih.gov/pubmed/?term=Small+proline-rich+repeat+protein+1A+is+expressed+by+axotomized+neurons+and+promotes+axonal+outgrowth [49] Xiao HS, Huang QH, Zhang FX, et al. Identification of gene expression profile of dorsal root ganglion in the rat peripheral axotomymodel of neuropathic pain. Proc Natl Acad Sci U S A. 2002; 99(12): 8360-8365.http://www.ncbi.nlm.nih.gov/pubmed/12060780 [50] Armin B, Paul L, Shingo T, et al. Conditioning lesions before or after spinal cord injury recruit broad genetic mechanisms that sustain axonal regeneration: Superiority to camp-mediated effects. Experimental Neurology. 2012; 235(1): 162-173.http://www.ncbi.nlm.nih.gov/pubmed/22227059 [51] Colak T, Cine N, Bamac B, et al. Microarray-based gene expression analysis of an animal model for closed head injury. Injury. 2012; 43(8): 1264-1270.http://www.ncbi.nlm.nih.gov/pubmed/?term=Microarray-based+gene+expression+analysis+of+an+animal+model+for+closed+head+injury [52] Wang KC, Koprivica V, Kim JA, et al. Oligodendrocyte-myelin glycoprotein is a Nogo receptor ligand that inhibits neurite outgrowth. Nature. 2002; 417(6892): 941-944.http://www.ncbi.nlm.nih.gov/pubmed/12068310 [53] Bandtlow CE, Schwab ME. NI-35/250/nogo-a: a neurite growth inhibitor restricting structural plasticity and regeneration of nerve fibers in the adult vertebrate CNS. Glia. 2000; 29(2): 175-181.http://www.ncbi.nlm.nih.gov/pubmed/?term=NI-35%2F250%2Fnogo-a%3A+a+neurite+growth+inhibitor+restricting+structural+plasticity+and+regeneration+of+nerve+fibers+in+the+adult+vertebrate+CNS [54] Guo Y, Ma L, Cristofanilli M, et al. Transcription factor Sox11b is involved in spinal cord regeneration in adult zebrafish. Neuroscience. 2011; 172: 329-341.http://www.ncbi.nlm.nih.gov/pubmed/?term=Transcription+factor+Sox11b+is+involved+in+spinal+cord+regeneration+in+adult+zebrafish [55] Ahdeah PG, Susan MK, Alan IF, et al. Characterization of inflammatory gene expression and galectin-3 function after spinal cord injury in mice. Brain Research. 2012; 1475: 96-105.http://www.ncbi.nlm.nih.gov/pubmed/22884909 [56] Almon RR, DuBois DC, Brandenburg EH, et al. Pharmacodynamics and pharmacogenomics of diverse receptor-mediated effects of methylprednisolone in rats using microarray analysis. Journal of pharmacokinetics and pharmacodynamics. 2002; 29(2): 103-129.http://www.ncbi.nlm.nih.gov/pubmed/12361239 |
| [1] | Qiu Xiao-feng, Liu Xue-mei. Fiber post versus metal post in repairing the maxillary premolars [J]. Chinese Journal of Tissue Engineering Research, 2015, 19(8): 1245-1249. |
| [2] | Li Hong-shi, Chen Xin, Feng Yan, Yin Yin, Cao Jun-kai. Changes of cartilage matrix components, related growth factors and inflammatory factors in the temporomandibular joint under high acceleration [J]. Chinese Journal of Tissue Engineering Research, 2015, 19(11): 1663-1667. |
| [3] | Ouyang Hai-chun, Wu Wo-dong, Zhong Dong-mei, Wu Yan-xian, Li Wen-jie, Chen Xi-ming. Effects of Xingnaojing on recombinant human tumor necrosis factor-mediated tissue-type plasminogen activator and plasminogen activator inhibitor-1 expression in human umbilical cord vein endothelial cells [J]. Chinese Journal of Tissue Engineering Research, 2015, 19(11): 1717-1721. |
| [4] | Wu Guang-wen, Ye Jin-xia, Zheng Chun-song, Chen Wen-lie, Liu Xian-xiang, Ye Hong-zhi . Effect of Tougu Xiaotong capsule on articular cartilage changes in rat models of osteoarthritis [J]. Chinese Journal of Tissue Engineering Research, 2014, 18(49): 7924-7929. |
| [5] | Liu Xia, Jiang Jia-ye, Lu Hai-ying, Gu Cui-ying, Cai Mei-qin. Response of the structure of rat kidney and inflammatory factors to Blackcurrant Extract at 24 hours after exhaustive exercise [J]. Chinese Journal of Tissue Engineering Research, 2014, 18(49): 8020-8025. |
| [6] | Cui Jing-fu, Xu Yao-zeng, Zhu Shi-jun, Zhu Feng, Fu Wen, Shao Hong-guo, Geng De-chun. Icariin inhibits titanium particle-induced inflammatory reaction [J]. Chinese Journal of Tissue Engineering Research, 2014, 18(16): 2563-2569. |
| [7] | Zhou Zhi, Liu Kai-xiang, Xie Yue, Wang Li-ming. Protective effects of low-concentration hydrogen sulfide on rheumatoid joint disease [J]. Chinese Journal of Tissue Engineering Research, 2014, 18(11): 1659-1664. |
| [8] | Zheng Peng-xiang, Zhou Huan, Tan Jian-ming. Impact of mesenchymal stem cells on the proliferation, invasion and biological behaviors of hepatocellular carcinoma cells [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(36): 6521-6526. |
| [9] | Wei Wei, Shi Geng-hu, Li Yu-tang, Zhang Bing, Gao Chuang. Research and application progress of visual fixation component separation [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4553-4560. |
| [10] | . Culture of bone marrow mesenchymal stem cells plus allogenic decalcified bone matrix in the knee cavity of rabbits for tissue engineered cartilage [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4371-4379. |
| [11] | Wen Cai-xiong. Resistance training and protein supplement: Improvement of elderly muscle protein metabolism [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4545-4552. |
| [12] | Lu Shou-ming, Lu Shou-liang, Sun Tian-wei, Zhang Hang, Wang Qi-ming. Estrogen effects on serum interleukin-8 and interleukin-10 expression in ovariectomized rats with osteoporosis [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4394-4400. |
| [13] | Adila•Azhati, Zhao Long, Wang Qian, Ma Yi-tong. Cardiac function and electrophysiological characteristics in myocardial infarction rats after tissue engineered cardiac patch transplantation [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4401-4408. |
| [14] | Shi Yu-lu, Li Xiao-yuan, Cao Mei-na, Yu Shu-yuan, Wang Ping. Simultaneous isolation of myocardial cells and cardiac fibroblasts from neonatal rats [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4414-4420. |
| [15] | Wang Qi-feng, Wang Zhi-gang, Huang Jing. Left ventricular papillary muscle ablation in canines by ultrasound ablation catheter [J]. Chinese Journal of Tissue Engineering Research, 2013, 17(24): 4409-4413. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||