中国组织工程研究 ›› 2025, Vol. 29 ›› Issue (7): 1429-1436.doi: 10.12307/2025.010
• 细胞相关实验/试验研究Cell related experimental/trial studies • 上一篇 下一篇
藏西北绒山羊子宫内膜容受性相关基因和可变剪接事件的综合分析
德 吉1,索朗达1,魏宇辰2,王 斌2,阿旺措吉1,仁青措姆1,崔久增3,张 磊3,巴 贵1
- 1西藏自治区农牧科学院畜牧兽医研究所,西藏自治区拉萨市 850009;2西北农林科技大学动物医学院,陕西省杨凌示范区 712100;3西北农林科技大学动物科技学院,陕西省杨凌示范区 712100
Comprehensive analysis of genes related to endometrial receptivity and alternative splicing events in northwest Tibetan cashmere goats
De Ji1, Suo Langda1, Wei Yuchen2, Wang Bin2, Awangcuoji1, Renqingcuomu1, Cui Jiuzeng3, Zhang Lei3, Ba Gui1
- 1Institute of Animal Sciences, Tibet Academy of Agricultural and Animal Husbandry Sciences, Lhasa 850009, Xizang Autonomous Region, China; 2College of Veterinary Medicine, Northwest A&F University, Yangling 712100, Shaanxi Province, China; 3College of Animal Science and Technology, Northwest A&F University, Yangling 712100, Shaanxi Province, China
摘要:
文题释义:
子宫内膜容受性:是指子宫内膜允许囊胚定位、黏附、穿透、植入,使胚胎着床、发育的能力。子宫内膜容受性与胚胎植入密切相关,是决定辅助生殖技术成功与否的关键因素之一。可变剪接:是指从一个mRNA前体中通过不同的剪接方式(选择不同的剪接位点组合)产生不同的mRNA剪接异构体的过程。可变剪接是调节基因表达和产生蛋白质组多样性的重要机制。
背景:藏西北绒山羊子宫内膜容受性是胚胎植入的关键因素,目前对于藏西北绒山羊子宫内膜容受性相关基因表达及可变剪接的认识还未明确。
目的:分析并挖掘藏西北绒山羊子宫内膜容受性相关的基因和可变剪接事件。
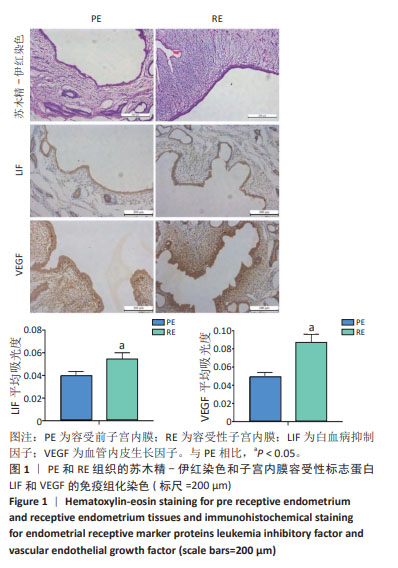
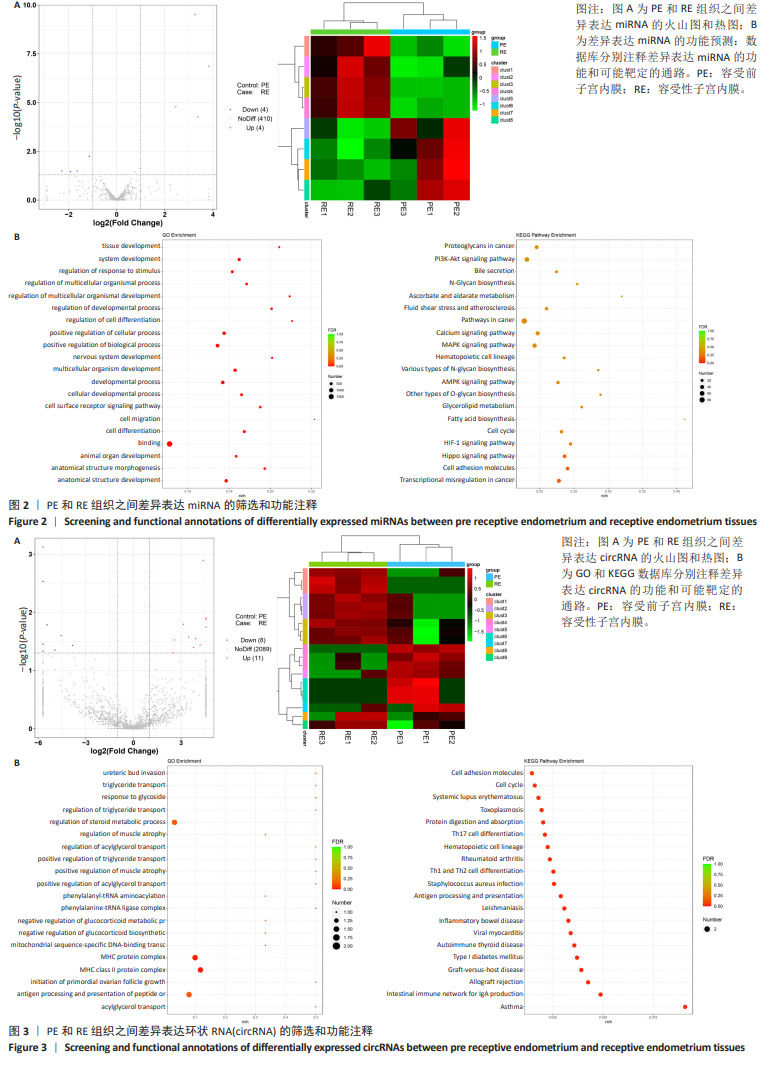
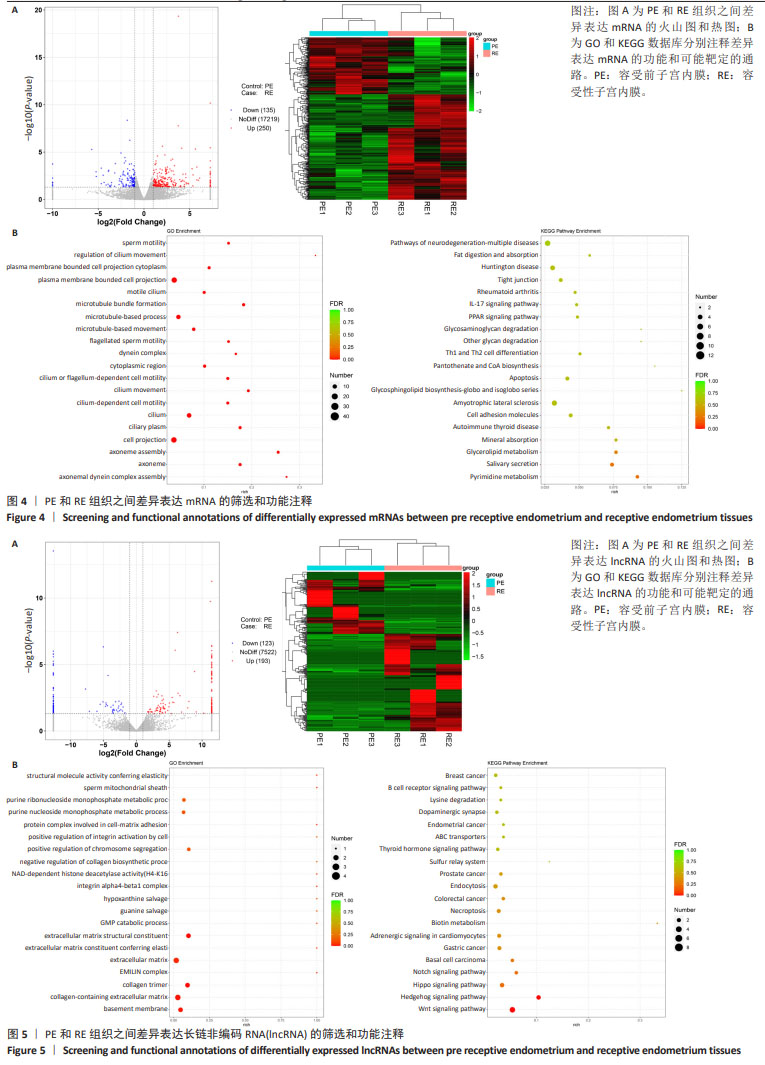
方法:在妊娠第5天和第15天(分别代表容受前子宫内膜组和容受性子宫内膜组),分别随机选取3只藏西北绒山羊,采集子宫内膜组织,苏木精-伊红染色观察组织形态,免疫组织化学检测子宫内膜容受性标志蛋白白血病抑制因子、血管内皮生长因子的表达水平;提取子宫内膜组织总RNA,质量检测合格后进行转录组测序,寻找差异表达的mRNA、长链非编码RNA、环状RNA和miRNA,进行功能预测,并分析与子宫内膜容受性相关的可变剪接mRNA和长链非编码RNA。
结果与结论:①与容受前子宫内膜组比较,容受性子宫内膜组子宫内膜组织中白血病抑制因子和血管内皮生长因子蛋白的表达水平明显升高;②测序结果显示,差异表达基因多为mRNA和长链非编码RNA,包括250个上调的mRNA、193个上调的长链非编码RNA、135个下调的mRNA和123个下调的长链非编码RNA,显著富集于Wnt、Hedgehog和Hippo信号通路;③可变剪接事件分析显示有8个差异表达的可变剪接转录本,均为mRNA分子,其中2个下调、6个上调,与血管内皮生长因子受体信号、细胞运动和胚胎发育有关。
https://orcid.org/0009-0009-2099-478X (德吉)
中国组织工程研究杂志出版内容重点:干细胞;骨髓干细胞;造血干细胞;脂肪干细胞;肿瘤干细胞;胚胎干细胞;脐带脐血干细胞;干细胞诱导;干细胞分化;组织工程
中图分类号: