Chinese Journal of Tissue Engineering Research ›› 2020, Vol. 24 ›› Issue (31): 5057-5063.doi: 10.3969/j.issn.2095-4344.2096
Previous Articles Next Articles
Single cell analysis applied to stem cell heterogeneity
Gao Shijun1, 2, Zhang Haiying2, Wang Tan1, Chen Li1, 2, Peng Yanan2, Tang Yunyi1, 2, Chen Zhibin1,Zhao Zhenqiang1
- 1Department of Neurology, the First Affiliated Hospital of Hainan Medical University, Haikou 570100, Hainan Province, China; 2Hainan Provincial Key Laboratory of Tropical Brain Research and Translation, Haikou 570100, Hainan Province, China
-
Received:2019-07-30Revised:2019-08-02Accepted:2019-12-13Online:2020-11-08Published:2020-09-04 -
Contact:Zhao Zhenqiang, MD, Chief physician, Professor, Master’s supervisor, Department of Neurology, the First Affiliated Hospital of Hainan Medical University, Haikou 570100, Hainan Province, China Chen Zhibin, MD, Chief physician, Professor, Master’s supervisor, Department of Neurology, the First Affiliated Hospital of Hainan Medical University, Haikou 570100, Hainan Province, China -
About author:Gao Shijun, Master candidate, Physician, Department of Neurology, the First Affiliated Hospital of Hainan Medical University, Haikou 570100, Hainan Province, China; Hainan Provincial Key Laboratory of Tropical Brain Research and Translation, Haikou 570100, Hainan Province, China -
Supported by:the National Natural Science Foundation of China, No. 81860238; the Key Research & Development Program of Hainan Province, No. ZDYF2018233; the International Cooperation Project of Hainan Province, No. KJHZ2015-09; the Health and Family Planning Research Project of Hainan Province, No. [2018]1018A200171; the Research Cultivation Foundation of Hainan Medical University, No. HY2018-20; the Postgraduate Innovation Research Project of Hainan Province, No. Hys2019-311; the National Natural Science Foundation of China, No. 8186050175; the Key Research & Development Program of Hainan Province, No. ZDYF2017170
CLC Number:
Cite this article
Gao Shijun, Zhang Haiying, Wang Tan, Chen Li, Peng Yanan, Tang Yunyi, Chen Zhibin, Zhao Zhenqiang . Single cell analysis applied to stem cell heterogeneity[J]. Chinese Journal of Tissue Engineering Research, 2020, 24(31): 5057-5063.
share this article
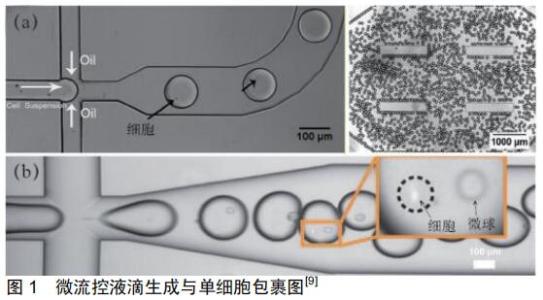
2.1 单细胞分离与收集技术 通过单细胞分析了解干细胞分化的异质性至关重要,而单个细胞的分离和收集是进行单细胞分析的前提,其技术主要包括传统的手动分离和流式细胞分选,以及近几年发展起来的微流控技术和单细胞机器人技术等。 2.1.1 手动分离和流式细胞分选 手动挑拣和极限稀释是最传统的单细胞分离技术。前者通过最大限度的稀释来获得单细胞悬液,该方法效率低且准确度差;后者通过显微镜观察细胞,利用微滴管吸取得到特定细胞,分离效率极低。流式细胞术是目前常用的单细胞筛选技术,通过光学系统和激光系统对单个细胞的荧光信号和散射信号进行检测,散射信号反映细胞的自身信息,包括形态、大小、内部结构等,而荧光信号反映了特定的细胞表面或内部抗原或抗体的特异信号[6]。 2.1.2 微流控技术 微流控技术根据分离的原理分为2类,一类是不需要借助外力的微孔法和微滴法;另一类为借助外力的声、光、电、磁法。微流控芯片技术具有准确、高通量等特征,但需要借助特殊的设备或器械。 (1)微孔法(流体力学捕获法):微孔法是在微流控芯片内制备障碍微结构,在细胞悬液中将单个感兴趣的细胞进行分离捕获的方法,包含2种结构,分别为微筛式结构和微坑式结构。微筛式结构通过微挡板阵列拦截和容纳单个目标细胞,在悬液通过后便自动形成了单细胞捕获阵列;微坑式结构是凹槽型结构阵列,当细胞悬液通过芯片时,细胞在重力下进入凹槽中,形成单细胞阵列,由于流体的冲击力小,使被捕获的单个细胞难以被冲走,每个凹槽与单细胞的尺寸相当。WLODKOWIC等[7]开发了含有上百个捕获位点的微流控芯片,应用于肿瘤单细胞的研究,证明了在细胞凋亡评估上这种芯片平台的效果与流式细胞仪相似。DEUTSCH等[8]通过刻蚀的方式在玻璃基底上设计了蜂窝状微坑阵列,将细胞悬液滴在微坑阵列表面,盖上盖玻片静置数分钟,细胞便在重力下沉降落入微坑内,产生了单个细胞的阵列,而每个微坑的大小可以根据不同细胞的种类进行调整。这种捕获阵列的构建有利于接下来的单细胞检测分析,如酶动力学研究、荧光标记等。 微孔法可以不用特殊的缓冲液,不依赖细胞标志物的表达水平,且操作方法简易,通过精准设计芯片中微通道与捕获微结构,实现目标单细胞的高通量捕获,而且单细胞的固定捕获位点可以进一步进行实时观察与追踪分析。传统的微孔芯片已被证明可用于筛选干细胞的蛋白质/基因标记物,但存在加工难度较大、成本较高、交叉污染、费力等问题,且必须手动冲洗芯片[9]。对于某些应用研究中,少量细胞易附着在孔的倾斜壁上。还很多研究缺乏自动检测功能(例如用于自动x/y运动的平板读数器或编程显微镜表),限制了在合理时间范围内可检测井的数量[10]。 (2)微滴法(液滴微流控捕获法):微流控技术是在微通道网络中产生纳升至皮升级的液滴,将单个感兴趣的细胞包裹后置于液滴内。该装置的核心是只容许单个细胞通过的通道,其直径可根据细胞的大小进行调节,并且通道可根据需要检测的细胞类型进行修饰。实验中为了减少流体用量、提高样品浓度,可以将微流控装置结合到生物芯片上[11]。此技术利用了物理学上可以在微米级尺度上忽略流体惯性的原理[12],人为控制细胞流体的流动,来实现单细胞的分离[13],每个液滴能够满足多种目标单细胞检测的研究,并能高效地避免交叉污染。在微灌注系统的辅助下,微流控芯片已经可以实现动态细胞培养作为恒化器。目前,微流体细胞培养设备已用于干细胞增殖和分化[14],已经有很多科学家在微流控芯片内成功培养了干细胞[15-17],并可以模拟细胞之间的相互作用。十字型流体聚焦法和T型通道法是生成微液滴最常见的方法,两者都可通过调控两相流速获得性质稳定、大小均一的液滴。用于包裹单个细胞的液滴一般用油包水的形式来处理,以保证细胞存活与生物相容性[18]。 当前的微流体技术已经加强了对细胞谱系的研究,但在微流体内单独检索姐妹细胞进一步分析的技术未完善,还不能排除单细胞或单独回收的细胞之间的相互作用,仍具有进一步分析的特异性[19]。液滴微流控捕获法的单细胞捕获芯片成本低、捕获速度快,且消耗的试剂和样品量较少,适用于需要将单个细胞和相关试剂单独包裹在微液滴再进行单细胞检测分析的研究。但此方法在单个液滴内没有或存在多个细胞包裹的现象,在包裹细胞后微液滴位置不易固定,不利于实时观察。 (3)单光束激光捕获法:单光束激光捕获技术是一种通过高度汇聚的激光束形成三维势阱,通过束腰附近存在强大的梯度力捕获并移动单个细胞的技术[9],见图1。通过调节光束,能够精准地利用光镊捕获细胞群中的单个细胞,再将目标细胞移动到特定的位置进一步分析检测[20]。 光镊捕获技术在完全密封的容器内进行,具备在微米级范围内定位的能力,能在不接触细胞的情况下,精确地捕获并移动单个细胞,不易污染且不会损伤细胞。但是,该技术成本较高,对设备要求高,不适用于高通量的单细胞捕获和分析。 (4)介电电泳捕获法:介电电泳现象是非均匀电场中介电粒子被极化而受力产生的定向移动[21]。在不同电极结构产生不同的非均匀电场下,介电粒子会根据正介电电泳力(p-DEP)或负介电电泳力(n-DEP)的作用向高电场或低电场运动,最终在相应的部位被捕获和固定,实现了粒子的有效操控[22]。而介电粒子的细胞在非均匀电场时,只需改变施加电压的大小和频率等条件,细胞便可通过所受到的介电电泳力来移动。当前,微流控芯片技术与介电电泳操纵技术的结合,得到了可靠的单细胞研究平台[23]。 介电电泳技术可以进行实时观察,并且捕获效率高、操作简单、对细胞损伤小。但是,这种方法需要特殊的缓冲液,精准设计电极结构与分析细胞所受到的介电电泳力,在确保细胞活性的同时实现高效地捕获单个目标细胞。 2.1.3 单细胞机器人技术 将单个靶细胞与细胞群分离,对建立高产细胞和进行细胞分析非常重要。传统的细胞筛选方法广泛地使用荧光激活细胞分选仪(FACS),但荧光激活细胞分选仪不能用于靶细胞含量百分比< 0.1%的样品,且不能基于细胞特性的时间依赖性变化对细胞进行分类。单细胞操纵支持机器人(SMSR)是一种用于在活细胞中显微注射特定基因和蛋白质以分析它们各自功能的有用机器。但传统的单细胞操纵支持机器人有较大缺陷。MATSUOKA等[24]将单细胞操纵支持机器人应用于显微注射到水稻原生质体和小鼠胚胎干细胞中。结果表明对这些细胞的显微注射比通常的动物细胞如成纤维细胞更困难。因此,TATEMATSU等[25]开发了新的自动单细胞分析和分离系统(单细胞机器人),可以对具有最高多能性的干细胞进行高通量筛选,可以促进单个细胞的非侵入性分离,从含有>105个细胞的阵列中获得最有利的特性,从而可以为生物药物建立新的细胞筛选方法。单细胞机器人在2013年成功开发并实现商业化[26]。 单细胞机器人作为一门高通量筛选的新兴技术,较微流体技术在操作上更简便,未来将更广泛的应用于干细胞领域。 2.2 单细胞分析技术 在过去的几年中,各种单细胞技术揭示了体外和体内不同干细胞群体的异质性,这些技术涵盖了从单个细胞的基因组学分析[27]、全转录组学分析和蛋白质组学分析[28-30]。近几年随着单细胞蛋白质组学分析和RNA测序发展迅速,单细胞分析检测技术被广泛用于不同细胞类型的鉴定与发现。该技术推进了蛋白质功能与特征的研究,为单细胞异质性的分析与鉴定奠定基础。通过质谱流式技术,结合多种表面抗体和胞内抗体,为细胞图谱绘制提供了表型数据。这些技术提供了大量关于组织内细胞异质性的信息,但它们无法提供细胞动力学的任何信息,因为它们代表单个细胞快 照[31]。新技术的最新发展旨在整合细胞的转录组学特征和它们的空间位置。 2.2.1 单细胞DNA分析技术 单细胞DNA分析技术是通过高通量测序平台对生物基因组中的基因进行测序,测定其DNA的碱基序列。该技术可在基因组水平上检测单核苷酸变异、插入缺失、拷贝数变异和结构变异等多种突变信息。 相同个体不同细胞间基因的组成与突变存在着差异,而鉴定细胞的类型与解决细胞异质性的重要方法是分析单个细胞的表观遗传信息和DNA。单细胞DNA测序技术检测包括外显子组测序、靶向重测序、全基因组测序等。全基因组测序可以获得整个基因组的信息,用于检测DNA变异、插入或缺失和拷贝数的改变,为当前应用广泛的单细胞测序分析技术[6]。 在2012年,ZONG等[32]提出了多重退火与成环循环扩增技术(MALBAC),在添加引物进行拟线性扩增后,再进行指数扩增。这种方法降低了序列的偏好性,并能覆盖单个细胞绝大部分的基因组。在2014年,HAN等[33]报道了微流体促进的方法,允许控制分离单个细胞的细胞质和核内容物,然后片上扩增基因组DNA和细胞质mRNA。当与片外聚合酶链反应、凝胶电泳和Sanger测序结合时,可以共同检测和测序来自相同单细胞的一组基因和转录物。在2017年,VITAK等[34]利用单细胞组合标记技术(SCI-seq)分析了单个目标细胞拷贝数的变异(CNV),利用锂辅助核小体耗尽(LAND)和SDS交联(xSDS)方法,清除了与DNA结合的蛋白质,同时保证了细胞核的完整性,得到了较为精准的CNV测序结果。这种方法可同时建立上千种低通量的单细胞CNV检测文库,可同时分析大约10 000个单细胞,极大地降低了文库的构建成本,并增加了检测通量。这种方法可应用于单细胞检测和不同类型细胞的表观遗传差异,为解决干细胞领域诸多问题例如异质性开辟了新的机会,有助于解决干细胞治疗的临床挑战。 2.2.2 单细胞RNA分析技术 RNA检测技术可以检测样本中所有的RNA转录本,开展基因表达分析或发现新型RNA。但是,测序的对象往往是细胞群或组织样本,这使细胞之间的差异可能会被细胞群的总体平均值所掩盖。 通过同时测量多个相关基因的mRNA水平,可以分析共同控制细胞事件调节的潜在基因相互作用,在理想情况下,应该研究单个细胞,以便与其他细胞进行比较。单细胞RNA-seq技术是根据基因表达谱来对细胞进行鉴定和分析。单细胞RNA测序技术有2种,一种是先反转录为cDNA,再通过二代测序技术进行测序;另一种是通过单分子测序技术,直接对RNA分子进行分析[6]。 高通量单细胞RNA测序技术需结合高效的单细胞分离技术和后续准确的测序分析。KLEIN等[35]利用微流控技术把带有条码的微珠与细胞装入微小的液滴内,建立了快速、高通量的单细胞RNA-seq方法。将细胞捕获入由反转录试剂、溶解液与条码标记的寡核苷酸引物组成的微滴内,在微滴中溶解细胞,释放mRNA,再通过条码标记的寡核苷酸引物将其合成出有标记的cDNA,接着利用CEL-seq对释放出的cDNA进行测序。在2013年,TANG等[36]开发了新的单细胞RNA-Seq技术,利用寡(dT)引物将含有poly(A)尾的mRNA反转录成cDNA,通过将cDNA片段从3'末端限制为0.85 kb来实现定量准确性,这相当于所有全长cDNA约7%,通过延长反转录的孵育时间和PCR的延伸时间可以将cDNA长度增加到3 kb并不会降低计数mRNA拷贝数的准确性。能够捕获大部分表达基因的全长cDNA,在相同细胞周期阶段的单细胞中,数百个基因同时表达至少2种转录物同种型。在2014年,STREETS等[37]提出了一种基于微流体的系统,用于从单个细胞制备cDNA和RNA测序,具有更高的精确度和灵敏度,通过微流体平台与单细胞全转录组分析的高通量测序相结合解决了这些局限性。对提取的总RNA和62个单小鼠胚胎细胞进行了技术重复测序,以对该技术的性能进行基准测试,使用TANG团队[36]的方案进行反转录和cDNA扩增,并显示出精确度和灵敏度的提高。2016年HABIB等[38]根据Nuc-Seq技术(一种高通量的单核单细胞RNA测序技术)开发了适用于神经细胞的Div-Seq技术,用于固定后或冰冻的神经细胞检测。HABIB等[39]继续开发了DroNc-Seq单核RNA测序方法,可以进行大规模的单细胞RNA测序,能够应用于复杂样品的测序。高效灵敏的测序技术是鉴定细胞类型的利器,2017年VILLANI等[40]通过单细胞RNA测序技术发现了新的人血树突状细胞和单核细胞。 2.2.3 单细胞蛋白质组学分析技术 蛋白质组学是在大规模水平上研究蛋白质的特征,包含蛋白质表达水平、翻译后修饰以及蛋白之间的相互作用等,得到蛋白质水平上有关细胞代谢、疾病发生机制等过程的全面认识。 从单细胞层面了解细胞特征与彼此之间的影响,可为生物系统细胞间的异质性提供可靠的信息。单细胞蛋白质组学技术是后基因组计划之一,单个细胞的蛋白质作为生命体内功能的直接执行者,与基因相比更有意义去揭示生命发育的发生发展的机制。该技术对单个细胞的蛋白质表达种类与表达量进行检测,分析不同细胞的表达差异,鉴定细胞类型。干细胞的单细胞蛋白质分析对了解细胞间异质性与细胞信号通路有很大作用[30]。 常用的单细胞蛋白质组学检测技术包括流式细胞技术和化学蛋白质组学。化学蛋白质组学采用化学探针研究蛋白质功能和机制。单细胞蛋白免疫印迹可以直接检测单个目标细胞内特定蛋白的表达水平或通过抗原抗体反应对细胞群分类筛选,来实现对单个目标细胞的蛋白质组学分析。流式细胞技术可以将细胞群在流体力学和超声波的作用下形成单细胞阵列通过激光束,使细胞通过激发的信号被检测器快速捕获,再根据细胞大小、荧光标记抗体等对单细胞表面或胞内蛋白进行筛选、鉴定、定性和定量分析,具有速度快、灵敏度高和准确度高的特点,是目前单细胞分析的重要手段。 随着单细胞高通量技术的发展,通过将蛋白质组学分析与活体成像和谱系追踪研究相结合,可以了解与周围微环境相关的哺乳动物干细胞行为[41]。多种技术的相互结合有助于在稳态条件、组织修复与微环境中调节干细胞活性并探索和揭示其新的机制[42]。 2.2.4 单细胞机器人分析技术 多基因和相关因子的相互作用参与胚胎干细胞的去分化与分化。这些基因和因子的定量分析对于阐明其机制至关重要。单细胞分析是生物医学科学中的一个新兴领域,需要具有不同分析能力的同等范围的仪器设备[43-44]。随着纳米技术的不断发展,基于原子力显微镜的纳米级别的操作机器人技术在单细胞等领域取得了极大成功。 已经被开发与应用的RoboSCell是一种高通量、基于单细胞微阵列的机器人仪器[43]。该仪器采用与机器人生物操纵器集成的部分透明单细胞微阵列(SCM),用于体外分析捕获在阵列位点的活单细胞。在透明单细胞微阵列中通过封装的坡莫合金通道引导磁场,在阵列位置捕获用免疫磁性粒子标记的细胞。RoboSCell将为动态单细胞分析提供准确和有效的手段。YASUKAWA等[45]在2002年根据单细胞微阵列开发了用于使细胞保持特定状态的装置,便于单个细胞更精准的分析。MATSUOKA等[24]在2005年开发了一种单细胞操纵支持机器人(SMSR),让操作员专注于显微注射,并在2007年研究了用于小鼠胚胎干细胞的高效显微注射的各种实验条件,成功证明了小鼠胚胎干细胞中EGFP基因表达的半定量控制及其敲低[46]。总而言之,目前单细胞操纵支持机器人的性能已经满足具有亚微空间分辨率的活胚胎干细胞中的动态基因表达分析。 新的自动单细胞分析和分离系统(单细胞机器人)极大地丰富了应用于干细胞的单细胞分析,但自动细胞检测和跟踪是高通量分析的主要瓶颈[47-48],这对于准确评估细胞的形态特征和迁移模式是必不可少的,并且在很大程度上减少了主观偏倚。在将来,随着自动细胞检测和跟踪技术的提高,单细胞机器人将发挥更大的作用。 2.2.5 相关技术在干细胞研究中的应用 干细胞是具有无限自我更新能力的细胞,可以演化成表型不同的细胞群。受细胞分化、克隆演化和微环境等因素影响,导致表型与功能的异质性。通过流式细胞仪把中脑多巴胺能神经元分为多个亚群,包括A8亚群、A9亚群、A10亚群和5-HT亚群等。已知只有A9中脑多巴胺神经元可以治疗帕金森病,但现有的分化方案尚未克服异质性问题,影响体内移植,是细胞源性多巴胺替代疗法进入临床的难点。单细胞分析对了解干细胞中的异质性至关重要,而微流控芯片是一种在高分辨率下比较测量和表征整个转录组模式的有效方法[49]。 在2009年,GLOTZBACH等[50]开发了一种微流体技术应用,结合信息理论的分析原理,定义了单个细胞的转录特征,并提供了辨别单细胞水平有意义的变异(信号)的能力。通过这种方法,检测到小鼠长期造血干细胞(LT-HSC)群体中基因表达谱的变化,并确定了几个转录定义的亚群,这些亚群与长期造血干细胞的已知功能异质性一致[51]。该方法在组织和器官水平上定义复杂细胞群体的转录组织的效用,以阐明转录和表型变异之间的关系。在2011年,MOON等[52]提出了一种基于微滴的方法,从异质细胞悬浮液(10%靶细胞混合物)中分离提取单细胞,可以保留提取细胞的活力(约97%),并从非分离细胞获得基因组信息-图案控件。在分离的细胞1 000个基因中鉴定出11个干细胞标记物,并与对照组进行比较。与现有的方法相比,这种自动化平台能够更有效地进行高通量细胞操作,以进行后续的基因组分析。微流体实时阵列已被用于人类诱导多能干细胞的基因表达谱分析[53],通过分析大型多能性和分化标记物的表达,为定量评估人类诱导多能干细胞的特征提供了一个新平台。WHITE等[49]提出了一个完全集成的微流体装置,能够执行每次运行数百个单细胞的基因表达的高精度RT-qPCR测量。在超过3 300个单细胞实验中,黏附和悬浮细胞系以及临床样品中精确比较了GAPDH和miR-16表达的分布,发现miR-16在K562细胞中被精确调节。该装置以纳升体积处理降低了测量噪音,提高了灵敏度,并提供了单核苷酸特异性,为干细胞的微流体单细胞转录分析奠定了基础。在2012年,SANCHEZ-FREIRE等[54]描述了用于评估单个多能细胞中基因表达谱的微流体平台,可以提供关于定义细胞群(例如人类诱导多能干细胞集落)内多能性变化的重要信息。该技术仅需要提取和表征相对少量的细胞,允许通过体内成像在动物模型中监测人类诱导多能干细胞增殖和存活方面的深入分析,可以直接比较人类诱导多能干细胞和人胚胎干细胞基因表达谱[53]。 另外,随着新一代测序成本的降低,文库制备正成为最终实验总成本中越来越重要的因素。MORA- CASTILLA等[55]提出了一个高通量的工作方案,应用于单细胞的转录组测序,其中使用单细胞自动制备系统(Fluidigm)制备单细胞RNA-seq文库,能够显著降低文库制备反应体积并增加反应数量。为了确定文库的质量,将该系统应用于对体外分化为胰腺细胞早期阶段的人胚胎干细胞进行分析。分析所得到的单细胞RNA-seq数据,确定该系统的可重复性及其区分不同分化阶段的细胞之间以及每个阶段内各个细胞之间的能力。 总之,随着干细胞的异质性问题得到广泛的重视,单细胞基因组测序与转录组分析扮演越来越重要的角色。近年来技术的发展使基因组测序与转录组分析获得了可靠的结果,解决干细胞分化的异质性问题指日可待。 "

|
[1] NOLBRANT S, HEUER A, PARMAR M, et al. Generation of high-purity human ventral midbrain dopaminergic progenitors for in vitro maturation and intracerebral transplantation. Nat Protoc. 2017;12(9): 1962-1979.
[2] 文路,汤富酬.单细胞转录组分析研究进展[J].生命科学, 2014,26(3): 228-233.
[3] KUMAR RM, CAHAN P, SHALEK AK,et al. Nature:揭示干细胞惊人异质性[J].岭南现代临床外科,2014,14(6):722.
[4] ZHANG JZ, TERMGLINCHAN V, SHAO NY, et al. A Human iPSC Double-Reporter System Enables Purification of Cardiac Lineage Subpopulations with Distinct Function and Drug Response Profiles. Cell Stem Cell. 2019;24(5):802-811.e5.
[5] WEN L, TANG F. Single-cell sequencing in stem cell biology. Genome Biol. 2016;17:71.
[6] 刘念,王园园,胡新笑,等.单细胞分析技术在人类细胞图谱计划中的应用[J].中国科学:化学,2018,48(10):1217-1225.
[7] WLODKOWIC D, FALEY S, ZAGNONI M, et al. Microfluidic single-cell array cytometry for the analysis of tumor apoptosis. Anal Chem. 2009; 81(13):5517-5523.
[8] DEUTSCH M, DEUTSCH A, SHIRIHAI O, et al. A novel miniature cell retainer for correlative high-content analysis of individual untethered non-adherent cells. Lab Chip. 2006;6(8):995-1000.
[9] 吴春卉,姜有为,程鑫.微流控芯片在单细胞捕获中的应用[J].科技导报, 2018,36(16):39-45.
[10] CHEN JY, TSAI WS, SHAO HJ, et al. Sensitive and Specific Biomimetic Lipid Coated Microfluidics to Isolate Viable Circulating Tumor Cells and Microemboli for Cancer Detection. PLoS One. 2016;11(3):e0149633.
[11] FENG X, DU W, LUO Q, et al. Microfluidic chip: next-generation platform for systems biology. Anal Chim Acta. 2009;650(1):83-97.
[12] 董燕,宋程程,黄鹤.单细胞测序技术研究进展[J].化学工业与工程, 2015, 32(1):71-78.
[13] BENÍTEZ JJ, TOPOLANCIK J, TIAN HC, et al. Microfluidic extraction, stretching and analysis of human chromosomal DNA from single cells. Lab Chip. 2012;12(22):4848-4854.
[14] JIANG CY, DONG L, ZHAO JK, et al. High-Throughput Single-Cell Cultivation on Microfluidic Streak Plates. Appl Environ Microbiol. 2016; 82(7):2210-2218.
[15] TATÁROVÁ Z, ABBUEHL JP, MAERKL S, et al. Microfluidic co-culture platform to quantify chemotaxis of primary stem cells. Lab Chip. 2016; 16(10):1934-1945.
[16] RICO-VARELA J, HO D, WAN LQ. In Vitro Microscale Models for Embryogenesis. Adv Biosyst. 2018;2(6):1700235.
[17] ZHANG Y, GAO S, XIA J, et al. Hematopoietic Hierarchy - An Updated Roadmap. Trends Cell Biol. 2018;28(12):976-986.
[18] HU Y, AZADI G, ARDEKANI AM. Microfluidic fabrication of shape-tunable alginate microgels: effect of size and impact velocity. Carbohydr Polym. 2015;120:38-45.
[19] ZHUANG J, WU Y, CHEN L,et al. Single-Cell Mobility Analysis of Metastatic Breast Cancer Cells. Adv Sci (Weinh). 2018;5(12):1801158.
[20] WANG XL, GOU X, CHEN SX, et al. Cell manipulation tool with combined microwell array and optical tweezers for cell isolation and deposition. Journal of Micromechanics & Microengineering. 2013; 23(7):075006.
[21] POHL HA. The Motion and Precipitation of Suspensoids in Divergent Electric Fields. Journal of Applied Physics. 1951;22(7): 869-871.
[22] ZHANG C, KHOSHMANESH K, MITCHELL A, et al. Dielectrophoresis for manipulation of micro/nano particles in microfluidic systems. Anal Bioanal Chem. 2010;396(1):401-420.
[23] WU C, CHEN R, LIU Y, et al. A planar dielectrophoresis-based chip for high-throughput cell pairing. Lab Chip. 2017;17(23):4008-4014.
[24] MATSUOKA H, KOMAZAKI T, MUKAI Y, et al. High throughput easy microinjection with a single-cell manipulation supporting robot. J Biotechnol. 2005;116(2):185-194.
[25] TATEMATSU K, KURODA S. Automated Single-Cell Analysis and Isolation System: A Paradigm Shift in Cell Screening Methods for Bio-medicines. Adv Exp Med Biol. 2018;1068:7-17.
[26] YOSHIMOTO N, KIDA A, JIE X, et al. An automated system for high-throughput single cell-based breeding. Sci Rep. 2013;3:1191.
[27] MACAULAY IC, HAERTY W, KUMAR P, et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 2015;12(6):519-522.
[28] KANG HM, SUBRAMANIAM M, TARG S, et al. Multiplexed droplet single-cell RNA-sequencing using natural genetic variation. Nat Biotechnol. 2018;36(1):89-94.
[29] SATO K, TSUYUZAKI K, SHIMIZU K, et al. CellFishing.jl: an ultrafast and scalable cell search method for single-cell RNA sequencing. Genome Biol. 2019;20(1):31.
[30] CUNHA B, AGUIAR T, CARVALHO SB, et al. Bioprocess integration for human mesenchymal stem cells: From up to downstream processing scale-up to cell proteome characterization. J Biotechnol. 2017;248:87-98.
[31] DONATI G. The niche in single-cell technologies. Immunol Cell Biol. 2016;94(3):250-255.
[32] ZONG C, LU S, CHAPMAN AR, et al. Genome-wide detection of single-nucleotide and copy-number variations of a single human cell. Science. 2012;338(6114):1622-1626.
[33] HAN L, ZI X, GARMIRE LX, et al. Co-detection and sequencing of genes and transcripts from the same single cells facilitated by a microfluidics platform. Sci Rep. 2014;4:6485.
[34] VITAK SA, TORKENCZY KA, ROSENKRANTZ JL, et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 2017;14(3):302-308.
[35] KLEIN AM, MAZUTIS L, AKARTUNA I, et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 2015;161(5):1187-1201.
[36] TANG F, BARBACIORU C, WANG Y, et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nat Methods. 2009;6(5):377-382.
[37] STREETS AM, ZHANG X, CAO C, et al. Microfluidic single-cell whole-transcriptome sequencing. Proc Natl Acad Sci U S A. 2014; 111(19):7048-7053.
[38] HABIB N, LI Y, HEIDENREICH M, et al. Div-Seq: Single-nucleus RNA-Seq reveals dynamics of rare adult newborn neurons. Science. 2016;353(6302):925-928.
[39] HABIB N, AVRAHAM-DAVIDI I, BASU A, et al. Massively parallel single-nucleus RNA-seq with DroNc-seq. Nat Methods. 2017;14(10): 955-958.
[40] VILLANI AC, SATIJA R, REYNOLDS G, et al. Single-cell RNA-seq reveals new types of human blood dendritic cells, monocytes, and progenitors. Science. 2017;356(6335): eaah4573.
[41] HOPPE PS, COUTU DL, SCHROEDER T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 2014;16(10): 919-927.
[42] KRETZSCHMAR K, WATT FM. Lineage tracing. Cell. 2012;148(1-2): 33-45.
[43] SAKAKI K, FOULDS IG, LIU W, et al. RoboSCell: an automated single cell arraying and analysis instrument. Biomed Microdevices. 2009; 11(6):1317-1330.
[44] GOONERATNE CP, KODZIUS R, LI F, et al. On-Chip Magnetic Bead Manipulation and Detection Using a Magnetoresistive Sensor-Based Micro-Chip: Design Considerations and Experimental Characterization. Sensors (Basel). 2016;16(9): E1369.
[45] YASUKAWA T, GLIDLE A, COOPER JM, et al. Electroanalysis of metabolic flux from single cells in simple picoliter-volume microsystems. Anal Chem. 2002;74(19):5001-5008.
[46] MATSUOKA H, SHIMODA S, OZAKI M, et al. Semi-quantitative expression and knockdown of a target gene in single-cell mouse embryonic stem cells by high performance microinjection. Biotechnol Lett. 2007;29(3):341-350.
[47] SCHROEDER T. Long-term single-cell imaging of mammalian stem cells. Nat Methods. 2011;8(4 Suppl):S30-35.
[48] LILJA AM, RODILLA V, HUYGHE M, et al. Clonal analysis of Notch1-expressing cells reveals the existence of unipotent stem cells that retain long-term plasticity in the embryonic mammary gland. Nat Cell Biol. 2018;20(6):677-687.
[49] WHITE AK, VANINSBERGHE M, PETRIV OI, et al. High-throughput microfluidic single-cell RT-qPCR. Proc Natl Acad Sci U S A. 2011; 108(34):13999-14004.
[50] GLOTZBACH JP, JANUSZYK M, VIAL IN, et al. An information theoretic, microfluidic-based single cell analysis permits identification of subpopulations among putatively homogeneous stem cells. PLoS One. 2011;6(6):e21211.
[51] GLAUCHE I, MOORE K, THIELECKE L, et al. Stem cell proliferation and quiescence--two sides of the same coin. PLoS Comput Biol. 2009; 5(7):e1000447.
[52] MOON S, KIM YG, DONG L, et al. Drop-on-demand single cell isolation and total RNA analysis. PLoS One. 2011;6(3):e17455.
[53] NARSINH KH, SUN N, SANCHEZ-FREIRE V, et al. Single cell transcriptional profiling reveals heterogeneity of human induced pluripotent stem cells. J Clin Invest. 2011;121(3):1217-1221.
[54] SANCHEZ-FREIRE V, EBERT AD, KALISKY T, et al. Microfluidic single-cell real-time PCR for comparative analysis of gene expression patterns. Nat Protoc. 2012;7(5):829-838.
[55] MORA-CASTILLA S, TO C, VAEZESLAMI S, et al. Miniaturization Technologies for Efficient Single-Cell Library Preparation for Next-Generation Sequencing. J Lab Autom. 2016;21(4):557-567.
[56] GUO G, HUSS M, TONG GQ, et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev Cell. 2010;18(4):675-685.
[57] LANG C, CAMPBELL KR, RYAN BJ, et al. Single-Cell Sequencing of iPSC-Dopamine Neurons Reconstructs Disease Progression and Identifies HDAC4 as a Regulator of Parkinson Cell Phenotypes. Cell Stem Cell. 2019;24(1):93-106.e6. |
| [1] | Fan Haixia, Tan Qingkun, Wang Hong, Cheng Huanzhi, Liu Xue, Ching-chang Ko, Geng Haixia. Rabbit skull defects repaired by the hydroxyapatite/geltin scaffold combined with bone marrow mesenchymal stem cells and umbilical vein endothelial cells [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(10): 1495-1499. |
| [2] | Jing Yucheng, Wang Le, Wang Xianyun, Wei Mei, Li Min, Ji Lishuang, Ma Fangfang, Liu Gang , Zheng Mingqi. Umbilical cord mesenchymal stem cell transplantation in the treatment of ischemic heart disease: a 3-year follow-up [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(1): 6-12. |
| [3] | Wei Yashu, Liu Hongjing, Wang Huifeng, Wang Junduo, Zhao Wenjing, Chen Weiping. Cardiomyocyte-like differentiation of bone marrow mesenchymal stem cells induced by myocardial tissue lysates from different parts of the myocardium [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(1): 32-37. |
| [4] | Gui Xianwei, Jiang Huijiao, Wu Jie, Liang Xueqi, Xu Xiaodan, Wang Erqiang, Zou Hailiang, Chen Hejie, Chen Xueling, Wu Xiangwei. Mesenchymal stem cell calcification induced by protoscolex of two species of Echinococcus: a differential analysis [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(1): 38-43. |
| [5] | Ye Dou, , Ma Xuexia , Guan Qian, , Luan Zuo , Yang Yinxiang , Wang Zhaoyan , Wang Qian , He Ying , Yao Ruiqin. Proportion and morphological characteristics of human oligodendrocyte precursor cells in different cell culture vessels [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(1): 44-49. |
| [6] | Gu Jingjing, Zhou Rui, Yang Tingting, Yang Xiaoping, Xu Fei, Zheng Bo. Supporting effect of human skeletal muscle-derived myoendothelial cells on hematopoietic stem/progenitor cells in vitro [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(1): 50-55. |
| [7] | Tao Guilu, Chu Tongbin, Zhang Lei. Proliferation and migration of endothelial progenitor cells promoted by bone marrow mesenchymal stem cells conditioned medium with rosiglitazone [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(1): 56-60. |
| [8] | Zhu Bingbing, He Haibin, Deng Jianghua, Wang Wenqiang, Mu Xiaoling. Human umbilical cord mesenchymal stem cells overexpressing interleukin 8 receptor inhibit inflammation and promote vascular repair [J]. Chinese Journal of Tissue Engineering Research, 2021, 25(1): 61-66. |
| [9] | Yu Xingge, Lin Kaili. Application of nanocomposite hydrogels in bone tissue engineering [J]. Chinese Journal of Tissue Engineering Research, 2020, 24(34): 5441-5446. |
| [10] | Zhang Huxiong, Li Wei, Yang Wupeng, New Suyaratu. 3D-printed icariin/decalcified bone matrix material promotes the repair of femoral condyle defects in rabbits [J]. Chinese Journal of Tissue Engineering Research, 2020, 24(34): 5461-5466. |
| [11] | Zhou Pengfei, Lin Jing, Chen Yuying, Lin Minkui. Canine dental pulp stem cells-polyglycolic acid scaffold complex for canine periodontal tissue defect [J]. Chinese Journal of Tissue Engineering Research, 2020, 24(34): 5526-5531. |
| [12] | Liu Xin, Du Bin, Sun Guangquan, Cao Jinxing, Jiang Xiaohong. Porous beta-tricalcium phosphate-polypyrrole-biotin-icariin composite scaffold promotes recruitment of bone marrow mesenchymal stem cells [J]. Chinese Journal of Tissue Engineering Research, 2020, 24(34): 5532-5537. |
| [13] | Zhang Shuang, Xu Xiaomei, Zeng Yang, Yuan Xiaoping, Lin Fuwei. Rev-erbα’s effect on osteoblastogenesis of mouse bone marrow mesenchymal stem cells [J]. Chinese Journal of Tissue Engineering Research, 2020, 24(31): 4921-4926. |
| [14] | Xun Chong, Wang Qiang, Li Changzhou, Liu Xiaofeng. Potential molecular targets and therapeutic mechanisms underlying transplantation of autologous bone marrow stem cells for the treatment of spinal cord injury based on bioinformatics [J]. Chinese Journal of Tissue Engineering Research, 2020, 24(31): 4927-4933. |
| [15] | Chen Jia, Yang Yiqiang, Hu Chen, Chen Qi, Zhao Tian, Yong Min, Ma Dongyang, Ren Liling. Fabrication of prevascularized osteogenic differentiated cell sheet based on human bone marrow mesenchymal stem cells and human umbilical vein endothelial cells [J]. Chinese Journal of Tissue Engineering Research, 2020, 24(31): 4934-4940. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||