Chinese Journal of Tissue Engineering Research ›› 2026, Vol. 30 ›› Issue (23): 5925-5933.doi: 10.12307/2026.787
Previous Articles Next Articles
Application and progress of transcriptomics and proteomics techniques in the study of intervertebral disc degeneration
Zhou Tianle1, Wang Wei2, 3, Zhang Zhiwen2, Liu Ximing3
- 1College of Acupuncture and Orthopedics, Hubei University of Chinese Medicine, Wuhan 430060, Hubei Province, China; 2Department of Orthopedic Surgery, Hubei Provincial Hospital of TCM, Affiliated Hospital of Hubei University of Chinese Medicine, Wuhan 430060, Hubei Province, China; 3Department of Orthopedic Surgery, General Hospital of Central Theater Command, Wuhan 430070, Hubei Province, China
-
Received:2025-08-04Accepted:2025-11-20Online:2026-08-18Published:2025-12-30 -
Contact:Wang Wei, MD, Associate chief physician, Master’s supervisor, Department of Orthopedic Surgery, Hubei Provincial Hospital of TCM, Affiliated Hospital of Hubei University of Chinese Medicine, Wuhan 430060, Hubei Province, China; Department of Orthopedic Surgery, General Hospital of Central Theater Command, Wuhan 430070, Hubei Province, China -
About author:Zhou Tianle, MS candidate, College of Acupuncture and Orthopedics, Hubei University of Chinese Medicine, Wuhan 430060, Hubei Province, China -
Supported by:2023-2024 Traditional Chinese Medicine Scientific Research Project of Hubei Provincial Administration of Traditional Chinese Medicine, No. ZY2023F006 (to WW); 2023-2024 Traditional Chinese Medicine Youth Talent Project of Hubei Provincial Administration of Traditional Chinese Medicine, No. ZY2023Q005 (to ZZW)
CLC Number:
Cite this article
Zhou Tianle, Wang Wei, Zhang Zhiwen, Liu Ximing. Application and progress of transcriptomics and proteomics techniques in the study of intervertebral disc degeneration[J]. Chinese Journal of Tissue Engineering Research, 2026, 30(23): 5925-5933.
share this article
Add to citation manager EndNote|Reference Manager|ProCite|BibTeX|RefWorks
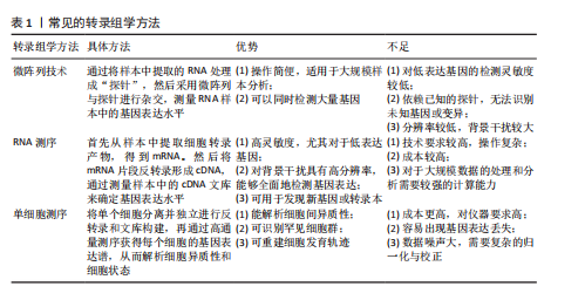
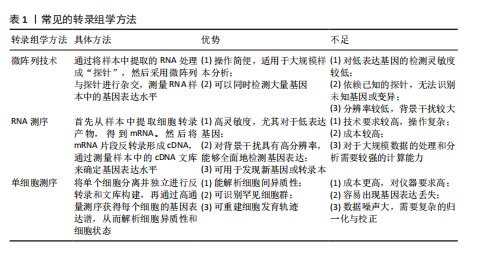
2.1 转录组学 转录组的概念最早由VELCULESCU 等[5]于1997年在研究酵母菌基因表达时提出。广义上是指在特定细胞类型、组织或生物体中针对特定条件转录出所有的完整基因或RNA,从RNA水平上研究基因表达、转录和调控。狭义上则是指细胞所能转录出的所有mRNA。转录组学则是从整体水平上研究细胞中基因转录的情况,也就是对转录组的研究。转录组学的研究流程一般分为实验操作和数据分析2个阶段(图3),实验操作阶段涉及从样本中提取RNA、构建cDNA文库,并通过高通量测序等技术获取转录组数据;数据分析阶段则利用生物信息学工具解析基因表达水平、调控网络及生物学功能。这些步骤依赖多种分子生物学技术,目前常见的转录组学方法(表1)包括:①微阵列技术:研究者通过将样本RNA转化为探针,与预设的微阵列芯片杂交,定量检测基因表达水平。尽管RNA测序技术逐渐成为主流,微阵列因成本效益和成熟的操作流程,仍广泛应用于转录组研究,尤其在特定基因表达分析中具有实用价值。②RNA测序:RNA测序是一种高分辨率技术,通过提取样本mRNA,反转录为cDNA后进行测序,以精确量化基因表达水平,优势在于对低丰度基因的高灵敏度以及对背景噪声的低干扰性,使它在解析复杂转录组动态时表现出色,成为当前转录组学研究的首选方法。③单细胞测序:用来获取细胞或组织的完整转录组数据,从而揭示基因表达变化。该技术主要是测定特定环境下单"

个细胞的基因水平,为细胞生理状态的描述提供了新的思路[6-7]。在此基础上发展出的空间转录组学技术,通过结合高通量测序和空间成像手段,能够在保持组织结构完整的前提下,同时获得基因表达信息及在组织中的具体空间位置。这一技术弥补了传统转录组技术在空间维度上的缺陷,使得研究人员不仅能观察基因的表达变化,还能解析基因在组织切片中的空间分布与细胞定位[8-9],对揭示细胞异质性、组织发育、疾病机制等具有重要价值。这些转录组学研究技术有助于研究者理解生物体在不同状态下基因表达的变化,并对基因调控、疾病机制等进行深入分析,有助于阐明疾病发病机制并为临床干预提供靶点。尽管如此,转录组学也存在一定的局限性,由于转录组数据主要反映mRNA水平,难以直接推断转录后修饰、翻译效率及蛋白质功能的变化,因此无法完全代表实际蛋白质表达情况。 2.1.1 转录组学在椎间盘退行性变发病机制中的研究 近年来,国内外学者持续深入探究椎间盘退行性变的分子机制,然而复杂的分子调控机制和病理生理过程仍未完全阐明,研究面临诸多挑战。转录组学技术为解析椎间盘退行性变的发病机制提供了重要工具,通过系统分析椎间盘退行性变患者椎间盘组织中的基因表达谱,可以识别关键的调控因子以及相关的生物学过程,为寻找新的治疗靶点和精准医疗提供重要参考,从而为椎间盘退行性变的临床诊断和个性化干预提供科学依据。在信号通路研究方面,ZHAO等[10]通过对椎间盘退行性变患者和脊髓损伤(对照组)患者的椎间盘样本进行RNA测序分析,然后进行全基因组范围内的长链非编码RNA鉴定,结果显示有1 854个长链非编码RNA在椎间盘退行性变和对照组之间差异表达。对目标基因进行京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路对比分析表明,这些长链非编码RNA参与多种通路调控,如溶酶体、焦点黏附和丝裂原活化蛋白激酶信号通路。HUANG等[11]通过RNA测序技术发现SOX4在退化的椎间盘组织中表达显著增加,而乙酰紫草素能通过激活磷脂酰肌醇3激酶/蛋白激酶B信号通路和抑制SOX4的表达来延缓椎间盘退行性变的进展。ZHANG等[12]通过对正常对照和椎间盘退行性变患者中分离的髓核组织进行单细胞测序分析,识别到7个软骨细胞亚群,其中有4个亚群(C1-C4)为首次发现,通过进一步基因本体(gene ontology,GO)富集和KEGG分析发现,铁死亡途径在这些亚群中富集。这些研究为进一步阐明椎间盘退行性变发展过程中的机制和病理变化提供了新的见解。 在关键基因调控研究方面,转录组测序可以反映出椎间盘中正在进行的基因表达程序,从而捕捉到基因表达的过程,有助于理解椎间盘退行性变的发病机制。国内学者何志伟[13]通过脂多糖诱导小鼠成软骨细胞建立退变软骨终板细胞模型,运用RNA测序技术得到差异表达基因,进一步实验发现通过促进脂多糖诱导的成软骨细胞中Becn2基因的表达,可缓解软骨终板退变,发挥延缓椎间盘退行性变的作用。李振聪[14]则采用单细胞测序技术对手术获取的正常和退变的髓核组织样本进行分析,发现与退变相关的基因表达水平发生变化,如Ⅱ型胶原α1链表达量降低,Ⅰ型胶原α1链和前列腺素E合成酶表达量升高。FERNANDES等[15]同样采用单细胞测序技术对从未退化椎间盘中分离的髓核和纤维环细胞进行转录组测序,通过调控网络分析识别出叉头框蛋白M1和赖氨酸去甲基化酶4E是纤维环和髓核的标志性转录因子,同时也是参与调控纤维环和髓核转录的核心基因。SHI等[16]通过对腰椎间盘突出和退行性脊柱侧凸患者的髓核样本进行转录组测序,比较由椎间盘退行性变导致的腰椎间盘突出和退行性脊柱侧凸的基因表达谱差异,发现了多个在退行性脊柱侧凸发病机制中起关键调控作用的非编码RNA,包括miRNA-766-3p、miRNA-326、novel_circ_0004327、XLOC_000209、GRK2。李志超等[17]通过转录组分析,发现U2AF同源基序激酶1(UHMK1)、锌指蛋白36样2(ZFP36L2)、CCHC型锌指含蛋白3(ZCCHC3)、锌指-BTB结构域含蛋白20(ZBTB20)等mRNA和LINC00641等长链非编码RNA可能是椎间盘退行性变的免疫相关关键靶点,并通过基因集富集分析确认LINC00641通过转化生长因子β通路调节椎间盘退行性变进程。这些发现为进一步研究参与调控椎间盘退行性变病理过程中的关键基因奠定了基础。 2.1.2 转录组学在椎间盘退行性变相关生物标志物研究中的应用 越来越多的研究表明,基因调控参与了椎间盘退行性变的病理进展,采用转录组学技术可以对疾病中的差异基因进行迅速筛选,有助于椎间盘退行性变早期诊断和揭示治疗新靶点。CHEN等[18]为进一步探究椎间盘退行性变的分子机制,通过单细胞测序分析人髓核细胞从而生成了一个全面的髓核细胞表达谱,并识别了晚期纤维化的髓核细胞,其特征是丝甘蛋白聚糖表达上调,因此认为晚期髓核细胞分泌的丝甘蛋白聚糖可作为早期诊断椎间盘退行性变的生物标志物。在miRNA研究方面,FAN等[19]对椎间盘损伤小鼠血清进行miRNA测序来表征与椎间盘退行性变相关的miRNAs,结果发现,血清中的miRNAs如miR-26a-5p、miR-122-5p和miR-215-5p在椎间盘退行性变过程中出现显著的变化,这些miRNAs可以作为诊断椎间盘退行性变的潜在生物标志物。此外,另一学者FAN等[20]对正常和退变的椎间盘进行研究,发现热休克蛋白90β家族成员1(HSP90B1)、核糖体蛋白S4Y链1(RPS4Y1)、核糖体蛋白L15 (RPL15)、泛转录四节肽重复含Y连锁蛋白(UTY)等作为枢纽基因,在椎间盘退行性变发展中起到关键作用。他们提出miR-124a和miR-506可作为椎间盘退行性变的生物标志物,能特异性识别椎间盘退行性变并反映病理进程。此外,在患者个性化诊断研究中,ZHAN等[21]系统地分析了椎间盘退行性变患者和健康人群之间的长链非编码RNA和miRNA表达谱,基于上调表达基因GRAM结构域含蛋白1B (GRAMD1B),下调表达基因淋巴细胞白血病缺失基因1(DLEU1)、ARMCX4、AC019205.1、DiGeorge综合征临界区基因5(DGCR5)和长链非编码RNA,建立了在训练数据集和验证数据集均具有100%诊断性能的遗传诊断模型和长链非编码RNA诊断模型。该研究为椎间盘退行性变提供了新的诊断特征,有助于今后设计椎间盘退行性变的个性化治疗方案。 2.1.3 转录组学在椎间盘退行性变药物研究及治疗中的应用 目前临床采用药物延缓或治疗椎间盘退行性变的效果尚不明确,转录组学技术在药物靶点筛查和疗效评价方面也发挥着重要作用。TAN等[22]通过研究花生四烯酸代谢在椎间盘退行性变中的作用,采用单细胞测序和RNA测序技术得到5个中心基因:醛-酮还原酶家族1C3成员(17β-羟基类固醇脱氢酶5型)(AKR1C3)、花生四烯酸5-脂氧合酶(ALOX5)、细胞色素P450 2B6(CYP2B6)、环氧化物水解酶2(EPHX2)和磷脂酶B1(PLB1)。对这些中心基因的功能深入探索研究发现,小分子药物AH6809可通过抑制醛-酮还原酶家族1C3成员(AKR1C3)表达从而延缓了椎间盘退行性变的进展。在中成药研究方面,国内学者何升华等[23]通过对腰突颗粒干预后的第3代髓核细胞进行RNA测序检测、差异基因表达及GO功能显著性富集发现,细胞外基质代谢途径关键基因表达下调,而多糖合成通路的相关基因上调,这为进一步研究该药物的疗效机制提供了分子生物学基础。在退变椎间盘修复方面,WANG等[24]利用单细胞测序技术解码小鼠纤维环细胞图谱,从而识别了一个新的纤维环细胞群体,称之为纤维软骨样纤维环细胞,在此基础上他们设计了一种新型复合水凝胶,发现该水凝胶对缺陷的纤维环具有重建作用,该研究进一步推进了椎间盘退行性变修复技术的发展。此外,CHEN等[18]通过对人类髓核细胞进行单细胞测序分析发现,天然香豆素衍生物瑞香素能够通过下调晚期髓核细胞中丝甘蛋白聚糖表达,减少炎症反应,从而减轻椎间盘退行性变进展。ZHAN等[21]通过结合药物-靶基因相互作用网络,也确定了对椎间盘退行性变有疗效的3种药物,即柠檬酸钙、磷酸钙和磷酸二水钙。这些研究为开发更有效的椎间盘退行性变治疗药物提供了有力的科学依据,为改善患者的生活质量提供了希望。 2.2 蛋白质组学 蛋白质组最早由澳大利亚科学家WILKINS和WILLIAMS于1994年提出[25]。蛋白质作为关键因素连接着基因型与表型,所以相较于转录组能更精确地反映出细胞的实际状态。蛋白质组学这一概念则最早诞生于1997年,是指以细胞、组织中的蛋白质为研究对象,运用高通量、高灵敏度的技术手段,对蛋白质进行全面的定性和定量分析。通过这些分析,可以揭示蛋白质的表达水平、修饰状态、相互作用网络以及它们在细胞中的定位等关键信息。在传统蛋白质组学基础上发展起来的空间蛋白质组学,进一步结合了成像质谱、质谱流式细胞术等先进技术,能够在保持组织原位结构的同时,实现蛋白质的空间定位和表达分析。该技术以单细胞或亚细胞分辨率检测数十至上百种蛋白,能够揭示蛋白质在组织中的精确分布与局部表达模式,这对于解析肿瘤微环境、细胞间相互作用、蛋白网络空间重构等具有重要意义[26]。依托人类蛋白质图谱等大型数据库的支持,空间蛋白质组学正成为理解疾病异质性和开发空间靶向疗法的重要手段。研究蛋白质组学的主要技术(表2)有双向电泳、液相色谱、生物质谱技术等。双向电泳是一种通过蛋白质的等电点和分子质量实现复杂蛋白质混合物分离的技术。通过对比不同样本的蛋白质图谱,可以鉴定差异表达蛋白,揭示在不同生理条件下的动态变化;液相色谱则用于蛋白质的分离和纯化,常与质谱技术结合,提升蛋白质组学研究的定量精度和鉴定效率。生物质谱技术是蛋白质组学中的核心工具,结合蛋白质分离技术和质谱分析,可用于鉴定未知蛋白质、定量蛋白质表达水平和检测蛋白质修饰,能够深入揭示与椎间盘退行性变相关的细胞外基质蛋白和细胞信号通路中的关键蛋 白[27]。蛋白质组学的研究过程一般分为以下几个步骤(图4):根据实验设计,准备样品并进行处理,以提取蛋白质;通过凝胶电泳等方法分离与富集蛋白质;利用胰蛋白酶将蛋白质酶解为肽段;对处理后的多肽样品进行质谱检测,获得质谱数据;通过数据库检索,将质谱数据转化为蛋白质的鉴定和定量信息;对获得的数据进行筛选,确定差异表达蛋白并进行生物信息学分析,从多个角度深入分析和挖掘蛋白质组学数据。通过深入研究蛋白质的变化和功能,研究者可以从蛋白质的角度理解生物体内的各种生物化学过程和疾病发生机制,通过识别特定的蛋白质表达模式和蛋白质相互作用机制,进而发现疾病的生物标志物和潜在的药物靶点。 2.2.1 蛋白质组学在椎间盘退行性变发病机制中的研究 通过分析椎间盘退行性变患者椎间盘组织中蛋白质的组成和功能,可以帮助研究者更好地了解病理过程和发现潜在的治疗靶点,推动针对椎间盘退行性变更精准、有效治疗方法的发展。WANGLER等[28]通过对健康、创伤性和退行性椎间盘"

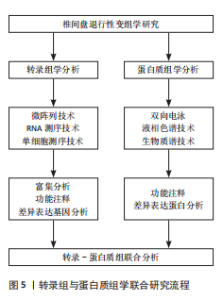
中间充质干细胞释放的蛋白质进行蛋白质组学研究,可以鉴定出与椎间盘退行性变密切相关的蛋白分子和信号通路。QIU等[29]运用蛋白质组学技术,针对胎儿和老年人这两个既相对独立又密切关联群体的研究,揭示了一系列与年龄相关性椎间盘退行性变潜在相关的差异表达蛋白:淋巴毒素α(LTA)、白细胞介素11(IL11)、热休克蛋白70家族成员8(HSPA8)、基质蛋白3(MATN3)、簇集蛋白(CLU)、乳脂球-表皮细胞生长因子8(MFGE8)、肿瘤坏死因子α诱导蛋白6(TNFAIP6)、基质重塑相关蛋白5(MXRA5)、细胞外基质蛋白1(ECM1)、血清胰蛋白酶抑制剂家族E1(SERPINE1)、血小板源生长因子C(PDGFC)和抑癌基因13(ST13),其中大部分差异表达蛋白为首次报道。该研究的富集分析表明,差异表达蛋白与各种类型的免疫反应、补体激活和丝氨酸蛋白酶相关活性密切相关。KUDELKO等[30]运用蛋白质组学技术,发现小鼠退变椎间盘中细胞间通信连接蛋白钙黏素2(CDH2)、胞间桥板蛋白(DSP)、间隙连接蛋白α1(GJA1)以及渗透调节蛋白溶质载体家族12成员2(SLC12A2)和WNK赖氨酸缺乏蛋白激酶1(WNK1)的富集。通过后续对这些蛋白的研究,进一步揭示椎间盘退行性变的发病机制。LIU等[31]对小鼠SM/J模型的髓核进行蛋白质组学分析发现,软骨寡聚基质蛋白(COMP)、富脯氨酸/精氨酸-亮氨酸重复蛋白(PROLARGIN)和纤维连接蛋白1(FN1)的表达均上调,这表明大量的基质重塑参与了髓核变性,并且与椎间盘退行性变人群的观察结果保持一致。因此,蛋白质组学在探索椎间盘退行性疾病发病机制方面具有重要的潜力。 2.2.2 蛋白质组学在椎间盘退行性变相关生物标志物研究中的应用 蛋白质组学技术运用于临床的目标之一是筛选疾病早期诊断和病理监测的生物标志物。采用蛋白质组学技术可以快速筛选出椎间盘退行性变的差异表达蛋白,有助于发现早期诊断椎间盘退行性变的新指标和治疗新靶点,这对于椎间盘退行性变的防治至关重要。TAM等[32]通过蛋白质组学在退变的髓核组织中筛选出能反映椎间盘退行性变发生、发展的标志性分子,结果发现在衰老的椎间盘中组蛋白1亚基(HIST1H1B/C/D/E、HIST1H2BL、HIST1H3A、HIST1H4A)和组蛋白2亚基(HIST2H2AC、HIST2H2BE、HIST2H3A)表达显著降低,认为这些因子可考虑作为诊断椎间盘退行性变发生的新型生物标志物。YANG等[33]研究发现,通过精准核内转录测序鉴定出α1-抗胰蛋白酶(血清胰蛋白酶抑制剂家族A1)(SERPINA1)、唾液酸糖蛋白2 (ORM2)、纤维蛋白原γ链(FGG)和Ⅰ型胶原α1链(COL1A1)是参与调节椎间盘退行性变的枢纽蛋白;进一步研究表明,SERPINA1因子在椎间盘退行性变模型大鼠及椎间盘退行性变患者椎间盘中均存在差异性表达,因此,认为SERPINA1可以作为调节或预测椎间盘退行性变进展的生物标志物。此外,国内学者王华聪[34]通过蛋白质组学技术比较热休克蛋白70家族成员8在正常与不同退变程度的腰椎间盘髓核组织中的表达情况,结果发现,随着腰椎间盘退行性变程度的加重,髓核细胞的数量和密度显著减少,热休克蛋白70家族成员8的表达量也逐渐减少,他认为热休克蛋白70家族成员8参与了人腰椎间盘退行性变的过程,可以作为腰椎间盘退行性变严重程度的生物学检测指标之一。ZHANG等[35]采用基于串联质谱标记蛋白定量分析的蛋白质组学来研究正常和衰老的髓核组织,通过筛选差异表达蛋白,发现椎间盘退行性变期间髓核细胞的衰老速度显著增加,并且它们能分泌改变细胞外基质的代谢因子;进一步采用GO分析显示,部分差异表达蛋白在细胞外基质、细胞蛋白质代谢过程和翻译后蛋白质的修饰过程中显著富集,其中包括基质金属蛋白酶3(MMP3)、纤溶酶原(PLG)、肌动蛋白α1(骨骼肌型)(ACTA1)、透光素(LUM)、组织金属蛋白酶抑制因子1(TIMP1)、纤溶酶原激活物抑制剂2(HEL-S-72P)、间型纤维蛋白(HEL113)、细胞分裂周期蛋白42(CDC42)、肽基脯氨酰顺反异构酶A(PPIA)和过氧化氢酶(CAT)等。另外,陈志光等[36]对正常和退变的椎间盘进行蛋白质组学分析发现,退变的椎间盘中胶原蛋白、纤维黏蛋白、基质金属蛋白酶、弹性蛋白等含量显著下降,这些差异表达蛋白也可作为椎间盘退行性变的分子标志物,这为椎间盘退行性变的后续研究提供了多个方向。值得一提的是,在基于实验室验证的生物标志物在临床实际应用中仍会面临诸多挑战,包括复杂的验证方法、接受个性化治疗的患者分层标准的不同、蛋白质丰度差异和异构体的存在等,因此这些标志物临床应用仍有待进一步验证和优化。 2.2.3 蛋白质组学在椎间盘退行性变药物研究及治疗中的应用 在通过蛋白质组学分析寻找治疗椎间盘退行性变有效药物的研究中,LIAO等[37]运用蛋白质组学的方法,发现双氢青蒿素通过抑制肿瘤坏死因子α诱导的磷脂酰肌醇3激酶/蛋白激酶B和核因子κB信号通路的激活,改善细胞外基质合成代谢和分解代谢失衡,减轻肿瘤坏死因子α诱导的髓核细胞衰老,参与延缓椎间盘退行性变进展。此外,目前已有多项研究发现,在椎间盘退行性变模型中移植骨髓来源间充质干细胞,激活了一组天然的、未表征的细胞,它们同时表达Ⅱ型胶原α1链和SOX9,从而发挥延缓椎间盘退行性变的作用[38-39]。在中医治法研究方面,邰志洪等[40]利用液相色谱-串联质谱联用技术分析腰椎间盘突出患者在接受两种非手术治疗前后血清中蛋白质表达差异,旨在鉴定活血化瘀疗法的关键血清标志物,研究发现,碳酸酐酶1与转铁蛋白变体是活血化瘀法治疗腰椎间盘突出血瘀证的目标蛋白质,为活血化瘀法治疗腰椎间盘突出提供了科学依据,并通过蛋白质组学技术揭示了潜在的作用机制。该研究还发现诸如基质金属蛋白酶和组织金属蛋白酶抑制因子等与细胞外基质降解相关的酶类的表达差异,这为疾病的生物标志物筛选提供了理论依据。这些研究为开发新的治疗策略提供了重要的理论基础和临床前数据。 2.3 转录组与蛋白质组学联合应用 近年来,虽已有研究试图通过转录组学或蛋白质组学分析来揭示椎间盘退行性变的调控网络,然而,两者均未能完全阐明退变过程的确切机制。虽然转录组学为研究椎间盘退行性变提供了基因表达层面的信息,但所揭示的结果并不足以全面反映细胞的生物学过程,因为基因的转录并不完全等同于蛋白质的表达。基因在转录、剪接、翻译成蛋白质的过程中由于转录后调控可能通过调节mRNA到蛋白产物的翻译过程来影响基因表达,这个过程是生物体中基因信息表达的关键环节,决定了细胞如何合成蛋白质以执行相关功能[41]。通过在蛋白质组学数据分析中整合表达的转录本信息,可以获得新的编码基因、交替翻译起始位点、剪接变异体、单氨基酸多态性等。转录组的敏感性高,但转录表达不足以提供真实的生物学状态,因为mRNA表达谱不能捕捉到可能影响活性蛋白数量的调控过程或转录后修饰过程。同样,蛋白质组学缺乏检测低丰度蛋白的敏感性,并且在识别新型蛋白形式方面能力有限。在转录组学和蛋白质组学的研究中,虽然已有许多研究者鉴定出潜在的靶点和生物标志物,但如何将这些发现有效推向临床仍是一个挑战。为了满足不同患者群体的需求,必须通过进一步的临床验证,确保这些标志物具有足够的特异性和敏感性。转化医学研究也需整合临床样本数据,进行大规模验证和临床试验,证明这些标志物在早期诊断、疾病监测和个性化治疗中的实际应用价值。基于现有的组学研究,开发与靶点相对应的药物或干预措施,并进行药物筛选和疗效评估,也将推动这些靶点和标志物在椎间盘退行性变临床管理中的应用,进一步支持精准医学和个性化治疗的发展。 整合转录组与蛋白质组学分析,揭示了RNA转录本与蛋白质表达水平之间的复杂关联,凸显了两者在调控机制中的互补性[42],这种联合研究为解析椎间盘退行性变的核心调控网络提供了多维度见解。整合转录组-蛋白质组学分析的步骤包括(图5):①采集高质量的组织或细胞样本;②分别通过RNA测序和蛋白质质谱分析获取基因转录本和蛋白质的信息;③进行数据整合,建立基因表达和蛋白质水平之间的关联;④借助生物信息学工具进行深入分析[43]。这一联合研究提供了多层次信息,从转录本到蛋白质水平,为全面理解生物系统提供了视角。通过免疫印迹法或实时荧光定量聚合酶链反应等实验验证关键发现,再整合这些数据,创建可视化的整合图谱,有助于系统性地理解不同分子水平之间的相互关系。"
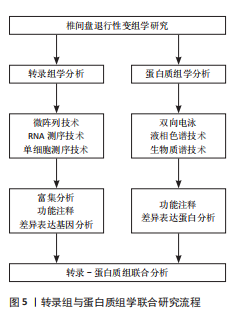
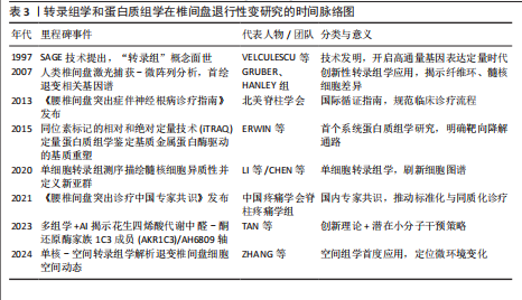
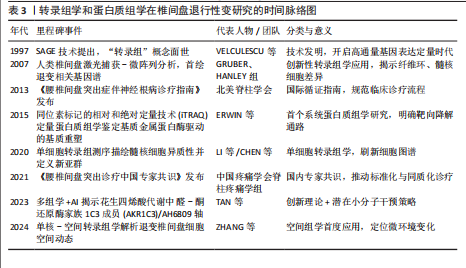
联合应用转录组学和蛋白质组学为研究复杂生物系统提供了独特优势,能够从基因转录到蛋白质表达提供多层次的生物学信息,深化对调控机制的理解,这种整合策略通过揭示mRNA与蛋白质之间的动态关联,有助于全面解析生物系统的功能网络。XU等[44]通过对新鲜分离的髓核细胞进行非标记蛋白质组学分析,同时从基因表达数据库中获得转录组分析数据进行生物信息学分析并构建RNA-蛋白质相互作用网络。研究进一步通过体外实验和免疫化学分析验证候选基因的功能,发现壳多糖酶3样蛋白1(CHI3L1)、角蛋白19(KRT19)、Ⅵ型胶原α2链(COL6A2)、皮肤桥蛋白(DPT)、肿瘤坏死因子α诱导蛋白6(TNFAIP6)和Ⅺ型胶原α2链(COL11A2)等6个基因在mRNA和蛋白质水平上表现出高度一致的变化。功能分析表明,当改变这些候选基因的表达时,会对髓核细胞有显著的影响,加速椎间盘退行性变。此外,联合分析能够识别mRNA与蛋白质表达水平的不一致现象,为研究转录后修饰和翻译调控提供了新视角。这种多组学整合方法不仅揭示了椎间盘退行性变的分子机制,还为筛选潜在生物标志物和治疗靶点提供丰富数据,为新型药物开发和临床干预策略的制定提供坚实的理论支持和实验依据。文章总结了转录组学和蛋白质组学在椎间盘退行性变研究的时间脉络图,见表3。"
| [1] TANG X, JING L, RICHARDSON WJ, et al. Identifying molecular phenotype of nucleus pulposus cells in human intervertebral disc with aging and degeneration. J Orthop Res. 2016;34(8):1316-1326. [2] WU PH, KIM HS, JANG IT. Intervertebral Disc Diseases PART 2: A Review of the Current Diagnostic and Treatment Strategies for Intervertebral Disc Disease. Int J Mol Sci. 2020;21(6):2135. [3] GBD 2019 DISEASES AND INJURIES COLLABORATORS. Global burden of 369 diseases and injuries in 204 countries and territories, 1990-2019: a systematic analysis for the Global Burden of Disease Study 2019. Lancet. 2020;396(10258):1204-1222. [4] SAEIDIAN AH, YOUSSEFIAN L, VAHIDNEZHAD H, et al. Research Techniques Made Simple: Whole-Transcriptome Sequencing by RNA-Seq for Diagnosis of Monogenic Disorders. J Invest Dermatol. 2020;140(6):1117-1126.e1. [5] VELCULESCU VE, ZHANG L, ZHOU W, et al. Characterization of the yeast transcriptome. Cell. 1997;88(2):243-251. [6] TEVES JM, WON KJ. Mapping Cellular Coordinates through Advances in Spatial Transcriptomics Technology. Mol Cells. 2020;43(7):591-599. [7] RAO A, BARKLEY D, FRANÇA GS, et al. Exploring tissue architecture using spatial transcriptomics. Nature. 2021;596(7871): 211-220. [8] BARECHE Y, BUISSERET L, GRUOSSO T, et al. Unraveling Triple-Negative Breast Cancer Tumor Microenvironment Heterogeneity: Towards an Optimized Treatment Approach. J Natl Cancer Inst. 2020;112(7):708-719. [9] LIU G, HU Q, PENG S, et al. The spatial and single-cell analysis reveals remodeled immune microenvironment induced by synthetic oncolytic adenovirus treatment. Cancer Lett. 2024;581:216485. [10] ZHAO B, LU M, WANG D, et al. Corrigendum to “Genome-Wide Identification of Long Noncoding RNAs in Human Intervertebral Disc Degeneration by RNA Sequencing”. Biomed Res Int. 2019;2019:3132626. [11] HUANG Y, LEI L, ZHAO Z, et al. Acetylshikonin promoting PI3K/Akt pathway and inhibiting SOX4 expression to delay intervertebral disc degeneration and low back pain. J Orthop Res. 2024;42(1):172-182. [12] ZHANG Y, HAN S, KONG M, et al. Single-cell RNA-seq analysis identifies unique chondrocyte subsets and reveals involvement of ferroptosis in human intervertebral disc degeneration. Osteoarthritis Cartilage. 2021; 29(9):1324-1334. [13] 何志伟.基于RNA-seq技术探究芝麻素影响椎间盘退变的机制研究[D].呼和浩特:内蒙古医科大学,2023. [14] 李振聪.基于单细胞测序的人正常和退变髓核细胞差异性分析[D].广州:暨南大学,2022. [15] FERNANDES LM, KHAN NM, TROCHEZ CM, et al. Single-cell RNA-seq identifies unique transcriptional landscapes of human nucleus pulposus and annulus fibrosus cells. Sci Rep. 2020;10(1):15263. [16] SHI X, LI P, WU X, et al. Whole-transcriptome sequencing identifies key differentially expressed circRNAs/lncRNAs/miRNAs/mRNAs and linked ceRNA networks in adult degenerative scoliosis. Front Mol Neurosci. 2023;16:1038816. [17] 李志超,王磊,薛景才,等.转录组分析鉴定腰椎椎间盘退行性变中潜在免疫相关生物标志物[J].脊柱外科杂志,2023, 21(5):316-325. [18] CHEN F, LEI L, CHEN S, et al. Serglycin secreted by late-stage nucleus pulposus cells is a biomarker of intervertebral disc degeneration. Nat Commun. 2024;15(1):47. [19] FAN Y, ZHAO L, XIE W, et al. Serum miRNAs are potential biomarkers for the detection of disc degeneration, among which miR-26a-5p suppresses Smad1 to regulate disc homeostasis. J Cell Mol Med. 2019;23(10):6679-6689. [20] FAN Z, ZHAO W, FAN S, et al. Identification of Potential Biomarkers for Intervertebral Disc Degeneration Using the Genome-Wide Expression Analysis. J Comput Biol. 2020;27(9):1341-1349. [21] ZHAN J, WANG S, WEI X, et al. Systematic analysis of Long non-coding RNAs reveals diagnostic biomarkers and potential therapeutic drugs for intervertebral disc degeneration. Bioengineered. 2021;12(1): 5069-5084. [22] TAN J, SHI M, LI B, et al. Role of arachidonic acid metabolism in intervertebral disc degeneration: identification of potential biomarkers and therapeutic targets via multi-omics analysis and artificial intelligence strategies. Lipids Health Dis. 2023;22(1):204. [23] 何升华,赖居易,孙志涛,等.基于RNA-seq技术分析腰突颗粒防治腰椎间盘退变的转录组学特征[J].中国组织工程研究,2017,21(24):3778-3783. [24] WANG H, WANG D, LUO B, et al. Decoding the annulus fibrosus cell atlas by scRNA-seq to develop an inducible composite hydrogel: A novel strategy for disc reconstruction. Bioact Mater. 2022;14:350-363. [25] WASINGER VC, CORDWELL SJ, CERPA-POLJAK A, et al. Progress with gene-product mapping of the Mollicutes: Mycoplasma genitalium. Electrophoresis. 1995;16(7):1090-1094. [26] THUL PJ, ÅKESSON L, WIKING M, et al. A subcellular map of the human proteome. Science. 2017;356(6340):eaal3321. [27] RAJASEKARAN S, TANGAVEL C, K S SVA, et al. Inflammaging determines health and disease in lumbar discs-evidence from differing proteomic signatures of healthy, aging, and degenerating discs. Spine J. 2020;20(1):48-59. [28] WANGLER S, KAMALI A, WAPP C, et al. Uncovering the secretome of mesenchymal stromal cells exposed to healthy, traumatic, and degenerative intervertebral discs: a proteomic analysis. Stem Cell Res Ther. 2021;12(1):11. [29] QIU C, WU X, BIAN J, et al. Differential proteomic analysis of fetal and geriatric lumbar nucleus pulposus: immunoinflammation and age-related intervertebral disc degeneration. BMC Musculoskelet Disord. 2020;21(1):339. [30] KUDELKO M, CHEN P, TAM V, et al. PRIMUS: Comprehensive proteomics of mouse intervertebral discs that inform novel biology and relevance to human disease modelling. Matrix Biol Plus. 2021;12: 100082. [31] LIU C, YANG M, LIU L, et al. Molecular basis of degenerative spinal disorders from a proteomic perspective (Review). Mol Med Rep. 2020;21(1):9-19. [32] TAM V, CHEN P, YEE A, et al. DIPPER, a spatiotemporal proteomics atlas of human intervertebral discs for exploring ageing and degeneration dynamics. Elife. 2020;9:e64940. [33] YANG X, LU Y, ZHOU H, et al. Integrated proteome sequencing, bulk RNA sequencing and single-cell RNA sequencing to identify potential biomarkers in different grades of intervertebral disc degeneration. Front Cell Dev Biol. 2023;11:1136777. [34] 王华聪.蛋白质组学70kDa热休克蛋白8变化对人椎间盘退变影响的实验研究[D].青岛:青岛大学,2019. [35] ZHANG G, LI L, YANG Z, et al. TMT-Based Proteomics Analysis of Senescent Nucleus Pulposus from Patients with Intervertebral Disc Degeneration. Int J Mol Sci. 2023; 24(17):13236. [36] 陈志光,叶东平.正常与退变人椎间盘的蛋白质组学差异比较[J].世界最新医学信息文摘(连续型电子期刊),2020, 20(72):119-120. [37] LIAO Z, SU D, LIU H, et al. Dihydroartemisinin Attenuated Intervertebral Disc Degeneration via Inhibiting PI3K/AKT and NF-κB Signaling Pathways. Oxid Med Cell Longev. 2022;2022:8672969. [38] SHAO J, YU M, JIANG L, et al. Sequencing and bioinformatics analysis of the differentially expressed genes in herniated discs with or without calcification. Int J Mol Med. 2017;39(1):81-90. [39] ZIGOURIS A, ALEXIOU GA, BATISTATOU A, et al. The role of matrix metalloproteinase 9 in intervertebral disc degeneration. J Clin Neurosci. 2011;18(10):1424-1425. [40] 邰志洪,桂裕昌,许建文,等.基于iTRAQ结合LC-MS/MS技术筛选腰椎间盘突出症活血化瘀治疗的目标蛋白质[J].时珍国医国药,2017,28(5):1256-1260. [41] MANZONI C, KIA DA, VANDROVCOVA J, et al. Genome, transcriptome and proteome: the rise of omics data and their integration in biomedical sciences. Brief Bioinform. 2018;19(2):286-302. [42] BEN-CHETRIT N, NIU X, SWETT AD, et al. Integration of whole transcriptome spatial profiling with protein markers. Nat Biotechnol. 2023;41(6):788-793. [43] KUMAR D, BANSAL G, NARANG A, et al. Integrating transcriptome and proteome profiling: Strategies and applications. Proteomics. 2016;16(19):2533-2544. [44] XU C, LUO S, WEI L, et al. Integrated transcriptome and proteome analyses identify novel regulatory network of nucleus pulposus cells in intervertebral disc degeneration. BMC Med Genomics. 2021;14(1):40. |
| [1] | Liu Jiafu, Ren Ruxia, Liao Zhouwei, Zhou Xiali, Wu Yihong, Zhang Shaoqun. Three-dimensional finite element analysis of cervical spine biomechanical characteristics in a rat model of cervical vertigo [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(9): 2182-2190. |
| [2] | Zhu Xiaolong, Zhang Wei, Yang Yang. Visualization analysis of research hotspots and cutting-edge information in the field of intervertebral disc regeneration and repair [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(9): 2391-2402. |
| [3] | Cai Qirui, Dai Xiaowei, Zheng Xiaobin, Jian Sili, Lu Shaoping, Liu Texi, Liu Guoke, Lin Yuanfang. Mechanical effects of Long’s traction orthopedic method on cervical functional units: quantitative analysis of biomechanical model of head and neck [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(9): 2208-2216. |
| [4] | Cai Ziming, Yu Qinghe, Ma Pengfei, Zhang Xin, Zhou Longqian, Zhang Chongyang, Lin Wenping. Heme oxygenase-1 alleviates lipopolysaccharide-induced inflammatory response in nucleus pulposus mesenchymal stem cells [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(7): 1624-1631. |
| [5] | Wang Peng, Li Zhijun, Zhang Shaojie, Wu Yimin. Intervertebral disc rehydration after posterior lumbar dynamic internal fixation [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(3): 711-720. |
| [6] | Jiang Chao, Che Yanjun. Biological mechanisms and future research trends of cartilaginous endplate degeneration [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(23): 5915-5924. |
| [7] | Xie Ziying, Li Songbo, Li Jianwen, Yin Yuchao, Zheng Baichuan, Hu Chengshang. Animal experimental study on the treatment of lumbar intervertebral disc degeneration with Chinese herbal compound: species selection, modeling method and drug administration [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(23): 5934-5942. |
| [8] | Li Chunchao, Julaiti·Maitirouzi, Xie Xuechen, Zhang Le, Wang Yixi, Paerhati·Rexiti. Finite element analysis of effect of passive support of deep intrinsic lumbar muscle groups on the lumbar spine [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(15): 3820-3831. |
| [9] | Chen Yuanjun, Lin Sixing, Ji Lichun, Li Dongxiao, Liao Guangzhi, Lin Xingdong. Anti-inflammatory activity and mechanism of Lonicera japonica Thunb.-derived extracellular vesicle-like particles [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(13): 3308-3320. |
| [10] | Liang Zhou, Pan Chengzhen, Chen Feng, Zhang Chi, Yang Bo, Wei Zongbo, Meng Jianhua, Zhou Zhu. Identification of diagnostic biomarkers related to osteoporosis exosomes and preliminary drug screening [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(13): 3458-3473. |
| [11] | Lu Yuchun, Zhu Zimo, Li Chaomeng, Liu Ju, Jiang Zixian, Li Xiufang, Wang Tao, Wang Wenjing. Effect and mechanism of dichloromethane extract of fresh Sambucus adnata Wall. in rat osteoarthritis models [J]. Chinese Journal of Tissue Engineering Research, 2026, 30(12): 3014-3028. |
| [12] | Qian Kun, Li Ziqing, Sun Shui . Endoplasmic reticulum stress in the occurrence and development of common degenerative bone diseases [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(6): 1285-1295. |
| [13] | Xiang Pan, Che Yanjun, Luo Zongping. Compressive stress induces degeneration of cartilaginous endplate cells through the SOST/Wnt/beta-catenin pathway [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(5): 951-957. |
| [14] | Ding Zhili, Huang Jie, Jiang Qiang, Li Tusheng, Liu Jiang, Ding Yu. Constructing rabbit intervertebral disc degeneration models by different methods under X-ray guidance: a comparative study [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(5): 995-1002. |
| [15] | Zhang Yixuan, Li Dongna, Liu Chunyan. Pathological processes, inflammatory responses, and related biomarkers of periodontitis: a multi-omics analysis [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(35): 7601-7610. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||