Chinese Journal of Tissue Engineering Research ›› 2025, Vol. 29 ›› Issue (20): 4351-4360.doi: 10.12307/2025.677
Previous Articles Next Articles
Targeted biomarkers of single-cell RNA sequencing in intervertebral disc degeneration
Liu Zhaofeng1, Yang Xuejun2
- 1Inner Mongolia Medical University, Hohhot 010030, Inner Mongolia Autonomous Region, China; 2Spine Surgery Centre A, The Second Affiliated Hospital of Inner Mongolia Medical University, Hohhot 010000, Inner Mongolia Autonomous Region, China
-
Received:2024-07-06Accepted:2024-08-28Online:2025-07-18Published:2024-12-25 -
Contact:Yang Xuejun, Professor, Chief physician, Master’s supervisor, Spine Surgery Centre A, The Second Affiliated Hospital of Inner Mongolia Medical University, Hohhot 010030, Inner Mongolia Autonomous Region, China -
About author:Liu Zhaofeng, Master candidate, Physician, Inner Mongolia Medical University, Hohhot 010030, Inner Mongolia Autonomous Region, China
CLC Number:
Cite this article
Liu Zhaofeng, Yang Xuejun. Targeted biomarkers of single-cell RNA sequencing in intervertebral disc degeneration[J]. Chinese Journal of Tissue Engineering Research, 2025, 29(20): 4351-4360.
share this article
Add to citation manager EndNote|Reference Manager|ProCite|BibTeX|RefWorks

2.1 单细胞转录组测序技术 单细胞转录组测序是从单个细胞水平上对单细胞中mRNA进行基因表达定量、功能富集、代谢通路等分析的一项新技术。与传统的RNA测序相比,单细胞转录组测序的优势在于它可以检测个体细胞中细微的转录组变化,阐明不同细胞间mRNA表达的异质性,并通过高效扩增和高通量测序收集广泛的样本数据。在椎间盘退变的研究中,单细胞转录组测序技术展现了其独特的价值。通过该技术,研究人员能够鉴定出椎间盘中不同的功能细胞亚群,揭示细胞来源和治疗靶点[5]。在椎间盘退变的背景下,单细胞转录组测序的快速发展确定了不同的分子和细胞特征和相互作用[6],最近的研究揭示了椎间盘退变中细胞的异质性,如髓核、纤维环、软骨终板、免疫细胞以及它们生物组成的变化[6-7], 这提高了对椎间盘退变病理过程的理解,并为治疗策略提供了新的见解。目前单细胞转录组测序在椎间盘退变研究中的主要流程包括椎间盘组织的采集与处理、单细胞分离和标记、mRNA反转录和cDNA扩增、高通量测序以及数据分析[8]。 2.1.1 椎间盘组织的采集与处理 椎间盘组织的获取来源包括大鼠、小鼠、牛尾及人类椎间盘组织等。大鼠、小鼠因易获取且通过诱导双足行走,进而可模拟椎间盘退变过程[9]。同时,牛尾椎间盘因其结构、生物力学特性以及易获取性等方面的优势,被广泛用作椎间盘退变研究的模型[10]。人类椎间盘组织的采集则主要来源于手术治疗中摘取的组织标本。研究人员常依据MRI的检查结果,采用Pfirrmann评级系统来评估椎间盘的退变程度[11]。为了确保样本的代表性和完整性,在采集椎间盘组织时必须严格操作。一旦采集完成,应立即将椎间盘组织放置于无菌且无RNA酶的冻存管中,并迅速冷冻于液氮中,以保持组织的RNA稳定性。采集后椎间盘组织的处理也不容忽视,需在实验室条件下迅速进行解冻,并立即清洗去杂质处理,旨在最大程度减少外部污染对实验结果的潜在影响[12]。送测时需与测序机构充分沟通,确保椎间盘组织样本包装与运输的安全稳定。之后采用单细胞分离技术将椎间盘组织解离成单个细胞悬液,随后进行mRNA反转录与cDNA扩增与高通量测序。 2.1.2 单细胞分离和标记 单细胞分离是单细胞转录组测序的第一步,是从复杂的细胞群体中将单个细胞分离出来的过程。单细胞分离方法包括有限稀释法、显微操作法、激光捕获显微切割、荧光激活流式细胞分选和微流控分选。其中荧光激活流式细胞分选具有细胞活性保持、高效且高质量的细胞分选和多参数分析等优势,现已在椎间盘退变研究中广泛应用[13-14]。分离得到的椎间盘单细胞进行特定标记后,形成具有特异标记的单细胞文库,以便于后续高通量测序和数据分析。 2.1.3 mRNA反转录和cDNA扩增 单细胞中的RNA含量通常为pg级,而高通量测序需要至少200 ng的DNA样品量,单细胞中的DNA含量不足以满足此要求,因此需将mRNA反转录为cDNA进行扩增[2]。轻微的降解、丢失和污染会显著影响测序分析,过度扩增也会传播误差,导致覆盖不均、噪声和量化不准确等问题,因此核酸扩增技术是单细胞测序的难点和关键[15]。常用的扩增原理分为3种:传统指数PCR扩增、体外反转录线性扩增和模板置换法。以此为原理,开发出许多转录组扩增技术,如模板转换法扩增术(Smart-seq、Smart-seq2、SCRB-seq)、体外线性扩增术(CEL-seq、CEL-seq2)、大规模平行扩增术(MARS-seq)、微流体扩增术(Drop-seq)[16]。在椎间盘退变研究中,应用最多的单细胞转录组测序方法为Smart-seq技术,Smart-seq在建库时能获取全长转录本,可以检测到更多的基因,凭借其高覆盖率和高灵敏度,适用于需要全长转录组数据和高分辨率的小规模研究[17-19]。在椎间盘退变中常用单细胞转录组扩增技术方法及优缺点见表1。 2.1.4 高通量测序及平台 细胞测序技术使用的高通量测序平台有很多,如illumina系列、PacBio系列、ONT平台、10x Genomics平台、BGI/MGI系列和Ion Torrent系列[20-26]。目前在椎间盘退变中应用最为广泛和成熟的高通量测序平台是illumina系列和10x Genomics平台。其中illumina系列和10x Genomics平台常用型号及特点见表2。这些平台各有其独特的优势和限制,研究人员可以根据具体的实验需求、预算和技术要求选择最合适的平台进行单细胞测序。最终,通过生物信息学工具分析测序数据,揭示椎间盘组织中单细胞的基因表达状态和细胞异质性,为相关研究提供新线索。"

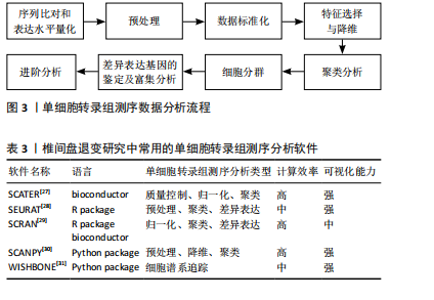
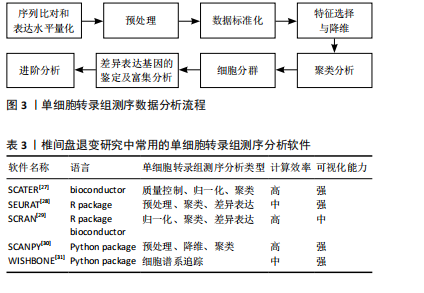
2.1.5 数据分析及处理 单细胞转录组测序的数据分析流程包括从序列比对到预处理、数据标准化、特征选择和降维、细胞分群以及差异表达基因的鉴定及富集分析等步骤,见图3。其中所用到的主流工具主要是由R和Python编写,常用于椎间盘细胞的单细胞转录组测序分析软件包括scater、Seurat、scran、Scanpy和Wishbone等[27-31],见表3。高通量测序后对RNA数据进行初步处理,比对后进行定量获得基因表达谱矩阵。该原始矩阵不能直接用于下游分析,还需要进行质量控制,包括去除低质量椎间盘细胞和缺失值填充进行预处理,提高数据分析的精度。在椎间盘退变研究中,特别需要关注与退变过程相关的细胞亚群,确保这些细胞的数据质量达到分析要求。Seurat等先进方法被广泛应用于单细胞转录组测序数据的标准化处理,它们能够更好地保留生物变异,可以消除各单细胞文库大小和测序深度的差异,提高数据的可信度[32], 之后通过特征选择筛选出与退变过程密切相关的基因,利用主成分分析、t-SNE等降维方法将高维数据映射到低维空间,有助于直观地展示细胞亚群的分布和异质性。通过聚类及细胞类群注释识别椎间盘不同发育阶段或病理状态下的细胞亚群,随后鉴定各细胞亚群的marker基因,对其进行差异表达基因分析和基因富集分析。通过比较退变组织与正常组织的基因表达谱,筛选出与退变过程密切相关的差异表达基因。进一步地,利用基因富集分析揭示这些基因参与的生物过程和信号通路,为深入理解椎间盘退变的分子机制提供线索。"
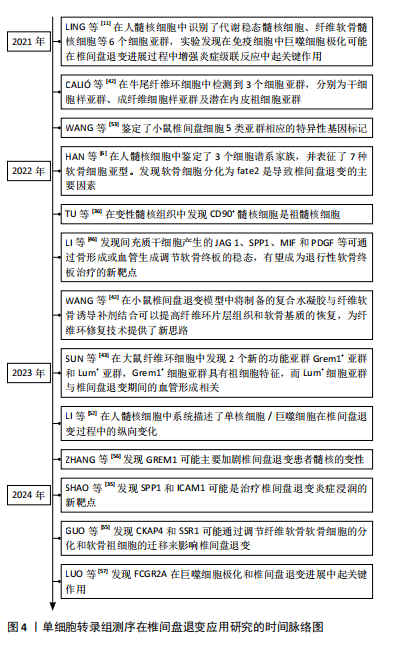
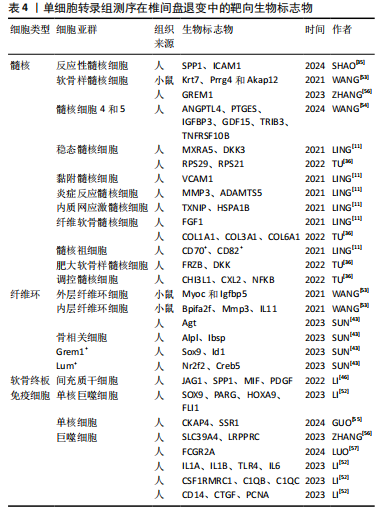
2.2 单细胞转录组测序在椎间盘退变中的应用 2.2.1 髓核组织的异质性 在健康的椎间盘中,髓核由一种高度水合的胶状物质组成,这种物质是由髓核细胞产生的细胞外基质成分构成的。这些细胞起源于胚胎的脊索,并在人体承受轴向和脊柱压力的能力中发挥着关键作用[33]。髓核由Ⅱ型胶原蛋白、蛋白多糖和髓核细胞组成,其中髓核细胞可以产生聚集蛋白多糖和细胞外基质成分,在维持椎间盘完整性方面起关键作用。越来越多的证据表明,细胞增殖、凋亡、细胞外基质生成与降解和mRNA水平分泌变化等髓核细胞的功能异常,是椎间盘退变发病机制的关键[34]。因此,髓核细胞的变性是椎间盘退变进程中不可或缺的一部分。 SHAO等[35]对8例退变的髓核组织进行了单细胞转录组测序研究,发现了4个髓核细胞亚群,亚群1为肥大软骨样髓核细胞,主要负责蛋白质合成;亚群2为反应型髓核细胞,主要负责应激反应;亚群3为支持型髓核细胞,主要负责细胞骨架的合成和调节;亚群4为储备型髓核细胞,缺乏特征性功能基因,主要负责RNA剪接。KEGG通路富集分析在时间序列中分为4组,分别主导椎间盘退变的健康、轻、中和重度退化过程。集群2代表健康阶段,主要由亚群4主导,富集通路主要集中在核糖体;集群1对应椎间盘退变前中期,由亚群1和2主导,主要富集在磷脂酰肌醇3激酶/蛋白激酶B(phosphoinositide3-kinase/protein kinase B,PI3K/Akt)通路和脂质与动脉粥样硬化通路;集群3对应椎间盘退变中后期,主要由亚群2主导,其富集通路包括矿物质吸收和化学致癌反应性氧物种通路;最后集群4对应椎间盘退变晚期,主要由亚群3主导,该集群中富集通路包括焦点黏附、Ras信号和丝裂原活化蛋白激酶信号通路。通过系统分析椎间盘退变的分化有助于了解椎间盘退变的整体发展过程。 HAN等[6]对5例不同程度椎间盘退变患者的6个髓核样本进行单细胞转录组测序分析,确定了内皮细胞、巨噬细胞和软骨细胞3类细胞谱系群。后来的研究主要集中在软骨细胞谱系群,共发现7个亚群,包括软骨祖细胞、纤维软骨细胞祖细胞、稳态软骨细胞和C1-C4四个新亚群。C1是退变疾病样本中最常见的亚型,随着椎间盘退变的不断进展,软骨祖细胞的比例显著下降;C3在重度退变组中的比例高于中度退变组,且仅在退变样本中检测到;进一步富集分析确定C1和C3是炎症反应亚群,C2高表达蛋白聚糖和Ⅱ型胶原,有助于维持细胞外基质的正常结构,C4具有类似成纤维细胞的表型,可能有助于细胞外基质重塑。然而,该研究并未明确这些软骨细胞类型的分化方向,这有待于未来的进一步探索。ZHANG等[1]使用单细胞转录组测序鉴定了7个软骨细胞亚群,与HAN等[6]的临床发现一致。其中,C1亚群可能参与血管生成;C2亚群主要表达细胞外基质分泌基因;C3亚群表现出参与细胞外基质炎症免疫反应和重塑基因的高表达水平;C4亚群表达维持胶原异位骨化平衡所必需的基因,可能有助于免疫反应;纤维软骨细胞祖细胞具有多分化能力,以保持纤维软骨细胞发育的稳态;稳态软骨细胞有助于调节软骨细胞代谢活性的稳态平衡,以响应环境信号,可能在蛋白质代谢中发挥重要作用。 为了进一步了解髓核的异质性,LING等[11]分离了3例椎间盘退变患者的髓核组织(Pfirrmann Ⅱ、Ⅲ和Ⅳ级),对其进行单细胞转录组测序,发现了11个不同的亚群。其中髓核细胞有6个亚群:①代谢稳态髓核细胞,高表达MXRA5和DKK3;②黏附髓核细胞,高表达VCAM1;③炎症反应髓核细胞,高表达MMP3和ADAMTS5;④内质网应激髓核细胞,高表达TXNIP和HSPA1B;⑤纤维软骨髓核细胞,高表达FGF1;⑥CD70+和CD82+髓核祖细胞。其中代谢稳态髓核细胞(32.37%)的比例在椎间盘退变早期(Ⅱ级)最高,其次是纤维软骨髓核细胞的比例在椎间盘退变中期(Ⅲ级,40.64%)和晚期(Ⅳ级,31.11%)增加。此外,纤维软骨髓核的比例在椎间盘退变的中后期增加,而代谢稳态髓核在早期最丰富。 TU等[36]分离了8例不同程度退变的椎间盘退变患者的髓核组织,并进行单细胞转录组测序,确定了6个亚群,分别为:①肥大软骨样髓核细胞,表达FRZB、DKK;②效应髓核细胞,表达编码参与基因细胞代谢蛋白质的mRNA;③稳态髓核细胞,表达RPS29、RPS21;④调控髓核细胞,表达CHI3L1、CXCL2、NFKB;⑤纤维髓核细胞,表达与纤维化、COL1A1、COL3A1、COL6A1相关的mRNA;⑥黏附髓核细胞,表达与细胞黏附和迁移相关的mRNA。进一步分析退变等级和亚群之间关系,Ⅱ级椎间盘的主要亚群为肥大软骨样髓核细胞和调控髓核细胞;Ⅲ级和Ⅳ级椎间盘的主要亚群为效应髓核细胞和肥大软骨样髓核细胞;对于Ⅴ级椎间盘,纤维髓核细胞和黏附髓核细胞是主要的髓核细胞。此外,随着椎间盘退变的严重程度进展(从Ⅱ级到Ⅴ级),黏附性髓核细胞和纤溶性髓核细胞的比例增加,而稳态髓核细胞的比例呈现相反的趋势。通过富集分析进一步分析亚群之间的功能差异:效应髓核细胞富含甾醇生物合成和糖胺聚糖代谢过程相关基因和细胞外基质组装的正调控;调控髓核细胞负责细胞对炎症和内源性刺激反应,可能潜在调节免疫功能;稳态髓核细胞富集于翻译调节和蛋白质、RNA代谢等细胞稳态相关的过程;黏附髓核细胞富集于细胞迁移和细胞基质黏附;纤维髓核细胞对大分子损伤有高度活性;肥大软骨样髓核细胞对细胞周期阻滞和衰老相关分泌的表观遗传学改变具有活性。 2.2.2 纤维环的异质性 纤维环环绕在髓核外围,通过限制髓核的膨胀压力和抵抗髓核施加的内部压力产生的剪切和拉应力来保持髓核。纤维环在微观结构下具有高度异质性,显微镜下,纤维环由15-25层交错排列的倾斜胶原纤维组成,在髓核外呈同心圆排列[37]。纤维环的外区纤维性更强,主要由胶原蛋白Ⅰ组成,胶原蛋白Ⅰ的含量从纤维环的内区向外区增加,内区软骨性更强,主要含有胶原蛋白Ⅱ和蛋白聚糖,而胶原Ⅱ和蛋白聚糖的含量则相反。除了微观结构的异质性外,纤维环的生物力学方面也具有典型的径向梯度特征,力学性能也会沿径向由内层向外层逐渐变化,从而产生区域特异性的力学性能和生物功能[38]。椎间盘退变的主要病理改变涉及纤维环缺陷,直接导致髓核疝和神经压迫。因此,椎间盘退变治疗的主要挑战在于修复纤维环缺陷[39]。然而,由于纤维环的异质性,使用单一子细胞类型和单一诱导环境恢复各种纤维环类型较为困难[40]。 为了进一步了解纤维环的异质性,CALIó等[41]对8例源自牛尾的椎间盘纤维环样本进行单细胞转录组测序,通过鉴定24个纤维环组织特异性基因对纤维环中的异质细胞群进行反卷积,共检测到3个亚群:1个干细胞样亚群,1个主要具有成纤维细胞样表型的亚群和1个潜在的内皮祖细胞亚群,说明了牛椎间盘中纤维环细胞表型的复杂性。WANG等[42]对小鼠纤维环组织进行单细胞转录组测序,确定了10个主要的细胞群,可大致分为3大类:祖细胞、成纤维细胞样细胞和软骨细胞样细胞。单细胞测序解码的纤维环细胞图谱,可为纤维环生物学研究提供细胞学基础。基于这一发现,WANG等[42]设计了一种纤维软骨诱导补剂,将其加入丝素蛋白和透明质酸形成的复合水凝胶中,在体外诱导纤维环干细胞生成了纤维软骨样纤维环细胞,其能够同时表达和分泌Ⅰ型胶原蛋白和Ⅱ型胶原蛋白,是纤维环重建过程中诱导的理想靶点。进一步在体内测试该复合水凝胶在纤维环缺损重建中的作用,将其植入小鼠椎间盘退变模型中可以促进纤维环片层组织和软骨基质的恢复,为纤维环修复技术提供了新思路。 SUN等[43]对8只大鼠的健康椎间盘纤维环细胞进行单细胞转录组测序,利用Seurat进一步将纤维环细胞分为7个亚群:表达Agt的内层纤维环,可能通过肾素-血管紧张素系统在纤维环中发挥调节作用;表达细胞外基质基因的外层纤维环;炎症相关细胞和氧化还原相关细胞,参与氧化还原过程,可能与维持组织稳态相关;骨相关细胞表达编码碱性磷酸酶α和编码骨唾液蛋白Ⅱ的Ibsp。除了上述5个亚群外,还有2个新发现的功能亚群,Grem1+亚群和Lum+亚群。Grem1+是纤维环的常驻祖亚群,Sox9和Id1是Grem1+亚群的关键调控因子,与组织发育和细胞分化有关;Lum+亚群与椎间盘退变期间的血管形成有关,Nr2f2和Creb5可能负责Lum+亚群的血管化功能。这些发现补充了对纤维环的认识,为椎间盘退变治疗和组织修复提供了潜在的细胞来源和调控靶点。 2.2.3 软骨终板的异质性 软骨终板位于椎间盘的上下侧,与椎体形成连接,是平衡椎间盘和椎体之间的压力,促进营养代谢通过椎血管运输的关键组成部分[44]。RADE等[45]以人群为基础的大型研究表明,软骨终板缺损和每个椎间盘水平的椎间盘退变有显著的相关性。因此,软骨终板退化可能是椎间盘退变发生的关键起始因素。LI等[46]对退行性软骨终板样本进行单细胞转录组测序,仅提取软骨终板组织进行后续分析。研究发现软骨终板组织由13个细胞亚群组成,表现出明显的细胞间异质性,这些细胞亚群可大致分为血细胞、血液相关细胞、巨噬细胞和间充质软骨细胞。其中血细胞参与抗原加工、递呈和各种免疫细胞相关过程,包括T细胞、B细胞和巨噬细胞的活化、细胞因子介导的信号传导、核因子κB信号通路等;血液相关细胞包括平滑肌细胞、血细胞和内皮细胞,平滑肌细胞通过PI3K-PKB信号通路参与肌肉收缩、血管生成调控和血管发育调控,这一发现证明组织中存在少量血管,可以促进营养物质扩散到髓核和纤维环组织;间充质软骨细胞进一步细分为稳态、效应和调节间充质软骨细胞,其中稳态软骨细胞主要参与骨化和胶原结合过程,调节软骨细胞主要参与刺激和控制反应,效应软骨细胞主要参与代谢过程;巨噬细胞方面,健康和退行性软骨终板组织主要由肥大细胞和M0巨噬细胞组成,M1和M2巨噬细胞仅在软骨终板组织中偶有表达,退行性软骨终板组织的免疫环境具有高度活化的树突状细胞、自然杀伤细胞和记忆性CD4+ T细胞,而M0巨噬细胞无明显变化。 2.2.4 免疫细胞的异质性 椎间盘突出是神经根性疼痛的主要原因之一。然而,疼痛并不总是与结构性椎间盘退变有关,疼痛可能是由于髓核物质通过纤维环裂缝渗漏或损伤,免疫细胞迁移到结构缺陷部位所产生,也可能与免疫细胞侵入血管和痛觉神经纤维有关[47]。椎间盘退变的特征包括CD68巨噬细胞、CD4和CD8 T细胞以及中性粒细胞的浸润[48]。RISBUD等[49]研究发现髓核、纤维环和免疫细胞如巨噬细胞、T细胞和中性粒细胞释放肿瘤坏死因子α、白细胞介素和干扰素等神经源性因子和细胞因子,导致椎间盘源性疼痛和椎间盘细胞的病理过程。与对照组相比,退行性椎间盘中调节性T细胞和巨噬细胞的浸润更高,随着椎间盘退变的进展,巨噬细胞的积累明显增加。LING等[11]研究表明,髓核组织还包括巨噬细胞、T细胞、骨髓祖细胞和中性粒细胞[14]。实验发现M2巨噬细胞的比例随着椎间盘退变的进展而下降,随后M1巨噬细胞呈上升趋势,这表明在椎间盘退变过程中巨噬细胞极化可能在增强炎症级联反应中起关键作用。 氧化还原稳态是许多细胞活动生理维持的关键过程,氧化应激被认为是氧化还原稳态失衡的一种状态,具有较高的活性氧生成,在椎间盘退变进展中也起着重要作用[50]。FENG等[51]研究发现活性氧的过度积累会引起氧化应激反应,损害髓核细胞的正常功能和完整性,从而引发炎症反应,促进椎间盘退变进展。LI等[52]对8例退行性变的髓核组织样本进行单细胞转录组测序,发现6种免疫细胞和其余类型的髓核相关细胞。晚期退行性椎间盘组织中氧化应激相关功能明显高于早期,单核/巨噬细胞在退行性组织中具有最高的氧化应激相关功能。基于不同的基因特征,免疫细胞中的单核/巨噬细胞进一步富集分析分为6个不同的亚群:氧化应激相关巨噬细胞在急性炎症反应、氧化应激反应等功能上富集,主要表达M1的标记物IL1A、IL1B、TLR4和IL6,可能具有与M2巨噬细胞类似的作用;活化组织巨噬细胞主要参与脂多糖的反应和炎症反应的调节;稳态巨噬细胞和效应单核/巨噬细胞主要具有组织重塑和胶原结合等保护功能,同时均高表达M2标记物CSF1R、MRC1、C1QB和C1QC,可能具有与M2巨噬细胞类似的作用;效应单核/巨噬细胞高表达单核细胞标记物CD14和增殖标记物CTGF和PCNA;调节性巨噬细胞主要参与细胞质翻译和体液免疫反应调节作用。此外,椎间盘退变期间单核/巨噬细胞各亚群比例发生了明显的变化,效应单核/巨噬细胞和调节性巨噬细胞亚群在退变Ⅱ级时占主导地位,但从退变Ⅲ级开始急剧下降;随着退行性分级的进展,氧化应激相关巨噬细胞、活化组织巨噬细胞和稳态巨噬细胞的比例增加,而效应单核/巨噬细胞和调节性巨噬细胞几乎不存在。这些研究进一步加深了对单核/巨噬细胞亚群在椎间盘退变进展过程中不同功能的理解,从而提供基于特定单核/巨噬细胞亚群的个性化治疗。 2.2.5 单细胞转录组测序在椎间盘退变中的靶向生物标志物 单细胞转录组测序在椎间盘退变的研究中展现出巨大潜力,通过单细胞水平的基因表达分析,能够详细解析椎间盘细胞的异质性和复杂的分子机制。该技术不仅揭示了不同细胞亚群的特征和功能,还可以识别出退变进程中起关键作用的潜在分子靶点,提高了对椎间盘退变病理的认识。 SHAO等[35]通过评估髓核细胞的特征模块,富集分析显示M1在反应性髓核细胞的炎症反应中不可或缺,在与应答性髓核细胞特征标记基因和免疫相关基因相交后鉴定出7个共表达基因——GBP2、ICAM1、NFKB1、PTX3、SAA1、SPP1和TNFAIP3,通过单细胞转录组测序进一步验证SPP1和ICAM1为髓核免疫浸润枢纽基因。巨噬细胞和髓核细胞分别通过SPP1-CD44途径和ICAM1-ITGB2配体受体途径在SPP1-ICAM双激活髓核细胞组中发挥促炎作用;同时,髓核细胞通过ANXA1-FPR1途径寻求中性粒细胞的自助炎症缓解。实验表明SPP1和ICAM1可能是治疗椎间盘退变炎症浸润的新靶点,极大地促进了对髓核细胞免疫微环境的了解。 WANG等[53]对小鼠椎间盘细胞进行单细胞转录组测序,在单细胞水平上将椎间盘细胞分为髓核、内外层纤维环、干细胞样和其他细胞5类亚群,并鉴定了相应的特异性基因标记,包括髓核细胞中的Krt7、Prrg4和Akap12基因、外层纤维环细胞的Myoc和Igfbp5基因和内层纤维环细胞的Bpifa2f、Mmp3和IL11基因,发现其中一个异质细胞组最有可能是干细胞组,为进一步研究单个椎间盘细胞对疾病状态的反应奠定了基础,并为今后的治疗提供更准确的靶细胞亚群和靶基因。 WANG等[54]对退变的髓核组织进行研究,将其分为软骨细胞1-5共5个亚群,其中,软骨细胞4和5主要出现在退行性髓核组织中。单细胞转录组测序显示Wnt/Ca2+信号在人正常髓核细胞和变性髓核细胞中存在差异表达,这种差异表达可能与变性髓核细胞中软骨细胞4和5的丰度密切相关。在退行性髓核细胞中,LRP5激活Wnt5B,通过调节Wnt5B的表达,促进髓核细胞凋亡和炎症反应。广泛存在于软骨细胞4和5中的ANGPTL4、PTGES、IGFBP3、GDF15、TRIB3和TNFRSF10B这6个与细胞凋亡和炎症反应相关的基因可能与髓核细胞的退行性变化密切相关。 LI等[52]发现特异性转录因子在不同的单核/巨噬细胞亚型中具有不同的功能。SOX9在软骨细胞分化中起重要作用,SOX9缺失可导致以细胞凋亡和基质重塑为特征的严重椎间盘退变;PPARG调节破骨细胞和成骨细胞的分化;HOXA9在fas介导的髓核细胞凋亡中起关键作用,可作为治疗椎间盘退变的潜在治疗靶点;FLI1在活化的免疫细胞中高表达,通过c端转录激活域调节单核/巨噬细胞的发育,此外还通过调节细胞因子和趋化因子参与影响免疫细胞的功能。通过转录因子可以在退行性椎间盘退变微环境中将其与特定单核/巨噬细胞亚型的下游治疗靶点结合起来,明确地促进髓核细胞稳态的发展。 LI等[46]分析各间充质干细胞群的潜在功能差异及与不同细胞类型可能的相互作用,发现不同细胞产生的JAG1、SPP1、MIF、PDGF可以通过成骨或血管生成调节软骨终板稳态,可作为退行性软骨终板的新治疗靶点。 GUO等[55]从椎间盘突出相关数据集中获得基因表达谱,并鉴定出1 015个相关基因;通过加权基因共表达网络分析进一步分析鉴定出5个关键基因,其中CKAP4和SSR1已被验证且免疫浸润也与单核细胞高度相关,显示出对椎间盘退变的显著诊断价值。CKAP4和SSR1主要表达于纤维软骨软骨细胞,推测可能通过调节纤维软骨软骨细胞的分化和软骨祖细胞的迁移来影响椎间盘退变,后通过对30例椎间盘样本进行体外验证表明CKAP4和SSR1在退变的人椎间盘组织中的表达增加。因此,CKAP4和SSR1已被确定为治疗椎间盘退变的有希望的治疗靶点。 ZHANG等[56]通过芯片数据的差异分析鉴定出6个椎间盘退变基因,并通过相关数据集验证筛选出3个关键基因GREM1、SLC39A4、LRPPRC,免疫浸润结果显示关键基因与树突状细胞、巨噬细胞等多种免疫细胞的浸润丰度相关。利用伪时间序列分析表明,GREM1的表达水平与髓核软骨细胞的分化轨迹基本一致,GREM1可能主要加剧椎间盘退变患者髓核的变性。 LUO等[57]对与椎间盘退变相关的转录组数据集进行单细胞转录组测序分析,显示485个差异表达基因与椎间盘退变相关,表明这些基因可能通过免疫调节影响椎间盘退变的发生。通过加权基因共表达网络分析和蛋白质互作用网络分析发现ITGAX、MMP9和FCGR2A是枢纽基因,主要在巨噬细胞中表达。通过体外和体内实验的功能验证证明了FCGR2A在巨噬细胞极化和椎间盘退变进展中起关键作用。机制上FCGR2A敲低可抑制M1巨噬细胞极化和核因子κB磷酸化,同时增强M2极化和STAT3激活,从而改善椎间盘退变。该研究揭示了FCGR2A在巨噬细胞极化中的调控功能,为椎间盘退变干预策略提供了新的见解。 综上,各种细胞类型的靶向生物标志物的鉴定提高了椎间盘的诊断和治疗。文章总结了单细胞转录组测序在椎间盘退变应用研究的时间脉络图及靶向生物标志物,见图4及表4。"

随着单细胞转录组测序技术的日益成熟及其在生物医学研究领域的广泛应用,该技术为椎间盘退变的早期诊断与治疗提供了新的可能性,特别是在靶向生物标志物的发现上展示出巨大潜力。椎间盘退变作为临床上引起下腰痛的主要原因,其发病机制尚未完全明确,缺乏有效延缓疾病进展的手段。单细胞转录组测序技术能够在单个细胞水平对mRNA进行高通量测序,精确揭示细胞间的基因表达差异及异质性,为深入理解腰椎间盘退变的分子机制提供了前所未有的分辨率。 在诊断方面,单细胞转录组测序技术能够识别椎间盘中不同类型细胞亚群的基因表达谱,如反应性髓核细胞、软骨样髓核细胞、稳态髓核细胞等,从而识别出与椎间盘退变发生发展密切相关的特异性基因。这些基因作为潜在的靶向生物标志物,加深了对椎间盘退变病理生"

理过程的理解,为疾病进展的检测提供了可靠指标。未来,通过结合机器学习算法和大数据分析,单细胞转录组测序技术有望实现椎间盘退变的早期、精准诊断,为个体化治疗方案的制定奠定基础。在治疗方面,靶向生物标志物的发现为开发个性化治疗方案提供了重要依据。单细胞转录组测序技术揭示的细胞异质性及其功能变化,使得针对特定细胞亚群或分子通路的靶向治疗成为可能。例如通过诱导具有再生潜力的祖细胞或干细胞特性细胞亚群的分化与增殖,可以促进椎间盘组织的修复与再生,为从再生医学和组织工程学角度探索椎间盘退变的治疗手段提供了可能。此外,基于单细胞转录组测序技术的分子机制研究成果,如免疫细胞在椎间盘退变中的作用及其调控机制,为开发靶向治疗药物提供了新思路。例如,通过调控特定免疫细胞如巨噬细胞极化,从而延缓椎间盘退变进程。 综上所述,单细胞转录组测序技术在椎间盘退变的诊断与治疗中具有重要价值,特别是在靶向生物标志物的发现上展现出广阔前景。随着技术的不断进步和研究的深入,有理由相信,这一技术将在椎间盘退变的临床诊疗中发挥越来越重要的作用,为改善患者生活质量作出积极贡献。"

| [1] ZHANG Y, HAN S, KONG M, et al. Single-cell RNA-seq analysis identifies unique chondrocyte subsets and reveals involvement of ferroptosis in human intervertebral disc degeneration. Osteoarthritis Cartilage. 2021;29(9):1324-1334. [2] 程浩天, 赵晓峰, 陆向东, 等. 单细胞转录组测序技术与椎间盘退变的发病机制[J]. 中国组织工程研究,2024,28(1):93-99. [3] MA JF, ZANG LN, XI YM, et al. MiR-125a Rs12976445 Polymorphism is Associated with the Apoptosis Status of Nucleus Pulposus Cells and the Risk of Intervertebral Disc Degeneration. Cell Physiol Biochem. 2016;38(1):295-305. [4] 吴昊, 叶冬平. 单细胞转录组测序在椎间盘退行性变中的应用[J]. 中国组织工程研究,2023,27(33):5370-5376. [5] HU X, WANG Z, ZHANG H, et al. Single-cell sequencing: New insights for intervertebral disc degeneration. Biomed Pharmacother. 2023; 165:115224. [6] HAN S, ZHANG Y, ZHANG X, et al. Single-Cell RNA Sequencing of the Nucleus Pulposus Reveals Chondrocyte Differentiation and Regulation in Intervertebral Disc Degeneration. Front Cell Dev Biol. 2022;10:824771. [7] KOROTH J, CHITWOOD C, KUMAR R, et al. Identification of a novel, MSC-induced macrophage subtype via single-cell sequencing: implications for intervertebral disc degeneration therapy. Front Cell Dev Biol. 2023;11:1286011. [8] SLOVIN S, CARISSIMO A, PANARIELLO F, et al. Single-Cell RNA Sequencing Analysis: A Step-by-Step Overview. Methods Mol Biol. 2021;2284:343-365. [9] AO X, WANG L, SHAO Y, et al. Development and Characterization of a Novel Bipedal Standing Mouse Model of Intervertebral Disc and Facet Joint Degeneration. Clin Orthop Relat Res. 2019;477(6):1492-1504. [10] ZHOU M, LIM S, O’CONNELL GD. A Robust Multiscale and Multiphasic Structure-Based Modeling Framework for the Intervertebral Disc. Front Bioeng Biotechnol. 2021;9:685799. [11] LING Z, LIU Y, WANG Z, et al. Single-Cell RNA-Seq Analysis Reveals Macrophage Involved in the Progression of Human Intervertebral Disc Degeneration. Front Cell Dev Biol. 2021;9:833420. [12] CAPREZ S, MENZEL U, LI Z, et al. Isolation of high-quality RNA from intervertebral disc tissue via pronase predigestion and tissue pulverization. JOR Spine. 2018;1(2):e1017. [13] WANG X, WANG Q, LI G, et al. Identifying the protective effects of miR-874-3p/ATF3 axis in intervertebral disc degeneration by single-cell RNA sequencing and validation. J Cell Mol Med. 2024;28(12):e18492. [14] ROBINSON JP, OSTAFE R, IYENGAR SN, et al. Flow Cytometry: The Next Revolution. Cells. 2023;12(14):1875. [15] 张永卓, 傅博强, 牛春艳, 等. 单细胞测序技术的发展[J]. 计量学报, 2023,44(1):149-156. [16] ZIEGENHAIN C, VIETH B, PAREKH S, et al. Comparative Analysis of Single-Cell RNA Sequencing Methods. Mol Cell. 2017;65(4):631-643.e4. [17] PICELLI S, FARIDANI OR, BJÖRKLUND AK, et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc. 2014;9(1):171-181. [18] CHERIF H, MANNARINO M, PACIS AS, et al. Single-Cell RNA-Seq Analysis of Cells from Degenerating and Non-Degenerating Intervertebral Discs from the Same Individual Reveals New Biomarkers for Intervertebral Disc Degeneration. Int J Mol Sci. 2022;23(7):3993. [19] HASHIMSHONY T, WAGNER F, SHER N, et al. CEL-Seq: single-cell RNA-Seq by multiplexed linear amplification. Cell Reports. 2012;2(3): 666-673. [20] Natarajan KN, Miao Z, Jiang M, et al. Comparative analysis of sequencing technologies for single-cell transcriptomics. Genome Biol. 2019;20(1):70. [21] Kucuk E, van der Sanden BPGH, O’Gorman L, et al. Comprehensive de novo mutation discovery with HiFi long-read sequencing. Genome Med. 2023;15(1):34. [22] GOUSSAROV G, MYSARA M, CLEENWERCK I, et al. Benchmarking short-, long- and hybrid-read assemblers for metagenome sequencing of complex microbial communities. Microbiology (Reading). 2024; 170(6):001469. [23] WANG X, HE Y, ZHANG Q, et al. Direct Comparative Analyses of 10X Genomics Chromium and Smart-seq2. Genomics Proteomics Bioinformatics. 2021;19(2):253-266. [24] KIM HM, JEON S, CHUNG O, et al. Comparative analysis of 7 short-read sequencing platforms using the Korean Reference Genome: MGI and Illumina sequencing benchmark for whole-genome sequencing. Gigascience. 2021;10(3):giab014. [25] CARBO EC, MOURIK K, BOERS SA, et al. A comparison of five Illumina, Ion Torrent, and nanopore sequencing technology-based approaches for whole genome sequencing of SARS-CoV-2. Eur J Clin Microbiol Infect Dis. 2023;42(6):701-713. [26] YANG X, LU Y, ZHOU H, et al. Integrated proteome sequencing, bulk RNA sequencing and single-cell RNA sequencing to identify potential biomarkers in different grades of intervertebral disc degeneration. Front Cell Dev Biol. 2023;11:1136777. [27] MCCARTHY DJ, CAMPBELL KR, LUN ATL, et al. Scater: pre-processing, quality control, normalization and visualization of single-cell RNA-seq data in R. Bioinformatics. 2017;33(8):1179-1186. [28] HAO Y, HAO S, ANDERSEN-NISSEN E, et al. Integrated analysis of multimodal single-cell data. Cell. 2021;184(13):3573-3587.e29. [29] HAGHVERDI L, LUN ATL, MORGAN MD, et al. Batch effects in single-cell RNA-sequencing data are corrected by matching mutual nearest neighbors. Nat Biotechnol. 2018;36(5):421-427. [30] WOLF FA, ANGERER P, THEIS FJ. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 2018;19(1):15. [31] SU M, PAN T, CHEN QZ, et al. Data analysis guidelines for single-cell RNA-seq in biomedical studies and clinical applications. Mil Med Res. 2022;9(1):68. [32] 李琳,钱四化,吕天琦,等. 新一代测序技术的文库制备方法研究进展[J]. 应用化学,2021,38(1):11-23. [33] DOU Y, SUN X, MA X, et al. Intervertebral Disk Degeneration: The Microenvironment and Tissue Engineering Strategies. Front Bioeng Biotechnol. 2021;9:592118. [34] LI Z, YU X, SHEN J, et al. MicroRNA in intervertebral disc degeneration. Cell Prolif. 2015;48(3):278-283.
[35] SHAO T, GAO Q, TANG W, et al. The Role of Immunocyte Infiltration Regulatory Network Based on hdWGCNA and Single-Cell Bioinformatics Analysis in Intervertebral Disc Degeneration. Inflammation. 2024.doi: 10.1007/s10753-024-02020-7. [36] TU J, LI W, YANG S, et al. Single-Cell Transcriptome Profiling Reveals Multicellular Ecosystem of Nucleus Pulposus during Degeneration Progression. Adv Sci (Weinh). 2022;9(3):e2103631. [37] LI J, LIU C, GUO Q, et al. Regional variations in the cellular, biochemical, and biomechanical characteristics of rabbit annulus fibrosus. PloS One. 2014;9(3):e91799. [38] CHU G, SHI C, WANG H, et al. Strategies for Annulus Fibrosus Regeneration: From Biological Therapies to Tissue Engineering. Front Bioeng Biotechnol. 2018;6:90. [39] ITO K, CREEMERS L. Mechanisms of intervertebral disk degeneration/injury and pain: a review. Global Spine J. 2013;3(3):145-152. [40] TORRE OM, MROZ V, BARTELSTEIN MK, et al. Annulus fibrosus cell phenotypes in homeostasis and injury: implications for regenerative strategies. Ann N Y Acad Sci. 2019;1442(1):61-78. [41] CALIÓ M, GANTENBEIN B, EGLI M, et al. The Cellular Composition of Bovine Coccygeal Intervertebral Discs: A Comprehensive Single-Cell RNAseq Analysis. Int J Mol Sci. 2021;22(9):4917. [42] WANG H, WANG D, LUO B, et al. Decoding the annulus fibrosus cell atlas by scRNA-seq to develop an inducible composite hydrogel: A novel strategy for disc reconstruction. Bioact Mater. 2022;14:350-363. [43] SUN H, WANG H, ZHANG W, et al. Single-cell RNA sequencing reveals resident progenitor and vascularization-associated cell subpopulations in rat annulus fibrosus. J Orthop Translat. 2023;38:256-267. [44] GRUNHAGEN T, SHIRAZI-ADL A, FAIRBANK JCT, et al. Intervertebral disk nutrition: a review of factors influencing concentrations of nutrients and metabolites. Orthop Clin North Am. 2011;42(4):465-477, vii. [45] RADE M, MÄÄTTÄ JH, FREIDIN MB, et al. Vertebral Endplate Defect as Initiating Factor in Intervertebral Disc Degeneration: Strong Association Between Endplate Defect and Disc Degeneration in the General Population. Spine (Phila Pa 1976). 2018;43(6):412-419. [46] LI W, ZHANG S, ZHAO Y, et al. Revealing the Key MSCs Niches and Pathogenic Genes in Influencing CEP Homeostasis: A Conjoint Analysis of Single-Cell and WGCNA. Front Immunol. 2022;13:933721. [47] NORBERTCZAK HT, INGHAM E, FERMOR HL, et al. Decellularized Intervertebral Discs: A Potential Replacement for Degenerate Human Discs. Tissue Eng Part C Methods. 2020;26(11):565-576. [48] SHAMJI MF, SETTON LA, JARVIS W, et al. Proinflammatory cytokine expression profile in degenerated and herniated human intervertebral disc tissues. Arthritis Rheum. 2010;62(7):1974-1982. [49] RISBUD MV, SHAPIRO IM. Role of cytokines in intervertebral disc degeneration: pain and disc content. Nat Rev Rheumatol. 2014;10(1): 44-56. [50] XIANG Q, ZHAO Y, LIN J, et al. The Nrf2 antioxidant defense system in intervertebral disc degeneration: Molecular insights. Exp Mol Med. 2022;54(8):1067-1075. [51] FENG C, YANG M, LAN M, et al. ROS: Crucial Intermediators in the Pathogenesis of Intervertebral Disc Degeneration. Oxid Med Cell Longev. 2017;2017:5601593. [52] LI W, ZHAO Y, WANG Y, et al. Deciphering the sequential changes of monocytes/macrophages in the progression of IDD with longitudinal approach using single-cell transcriptome. Front Immunol. 2023;14:1090637. [53] WANG J, HUANG Y, HUANG L, et al. Novel biomarkers of intervertebral disc cells and evidence of stem cells in the intervertebral disc. Osteoarthritis Cartilage. 2021;29(3):389-401. [54] WANG P, LI Z, YE D. Single-cell RNA-seq analysis reveals the Wnt/Ca2+ signaling pathway with inflammation, apoptosis in nucleus pulposus degeneration. BMC Musculoskelet Disord. 2024;25(1):321. [55] GUO D, ZENG M, YU M, et al. SSR1 and CKAP4 as potential biomarkers for intervertebral disc degeneration based on integrated bioinformatics analysis. JOR Spine. 2024;7(1):e1309. [56] ZHANG Z, HUO J, JI X, et al. GREM1, LRPPRC and SLC39A4 as potential biomarkers of intervertebral disc degeneration: a bioinformatics analysis based on multiple microarray and single-cell sequencing data. BMC Musculoskelet Disord. 2023;24(1):729. [57] LUO J, JIN G, CUI S, et al. Regulatory mechanism of FCGR2A in macrophage polarization and its effects on intervertebral disc degeneration. J Physiol. 2024;602(7):1341-1369. |
| [1] | Qian Kun, Li Ziqing, Sun Shui . Endoplasmic reticulum stress in the occurrence and development of common degenerative bone diseases [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(6): 1285-1295. |
| [2] | Xiang Pan, Che Yanjun, Luo Zongping. Compressive stress induces degeneration of cartilaginous endplate cells through the SOST/Wnt/beta-catenin pathway [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(5): 951-957. |
| [3] | Ding Zhili, Huang Jie, Jiang Qiang, Li Tusheng, Liu Jiang, Ding Yu. Constructing rabbit intervertebral disc degeneration models by different methods under X-ray guidance: a comparative study [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(5): 995-1002. |
| [4] | Zhang Xiaoyu, Wei Shanwen, Fang Jiawei, Ni Li. Prussian blue nanoparticles restore mitochondrial function in nucleus pulposus cells through antioxidation [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(34): 7318-7325. |
| [5] | Su Yongkun, Sun Hong, Liu Miao, Yang Hua, Li Qingsong. Development of novel antioxidants and antioxidant combination carried by nano-hydrogel systems in treatment of intervertebral disc degeneration [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(34): 7376-7384. |
| [6] | Yu Qinghe, Cai Ziming, Wu Jintao, Ma Pengfei, Zhang Xin, Zhou Longqian, Wang Yakun, Lin Xiaoqin, Lin Wenping. Vanillic acid inhibits inflammatory response and extracellular matrix degradation of endplate chondrocytes [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(30): 6391-9397. |
| [7] | Gao Xilin, Wu Si Zhang Chao Zhu Liguo, Fu Bifeng, Wang Ping. Mechanotransduction proteins in intervertebral disc degeneration [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(3): 579-589. |
| [8] | Xi Haixiang, Duan Jie, Xu Ping, Fei Xi, Li Xiaoping, Cao Lei, Tang Guangping, Zhang Lei. Syringin-chitosan hydrogel suppresses intervertebral disc degeneration [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(28): 5968-5976. |
| [9] | Pan Hongyu, Li Hongtao, Xiao Changming, Li Sen. Biomechanical analysis on treatment of different types of osteoporotic vertebral compression fractures with individualized precise puncture vertebral augmentation [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(27): 5773-5784. |
| [10] | Bai Liang, Fu Su, Yan Xu, Zhang Chunlin, Li Ying . Measurement of intervertebral disc height and analysis of strength after induced resorption of herniated nucleus pulpous [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(27): 5785-5794. |
| [11] | Ma Jikun, Wang Jianru, Qi Junjie, Liu Haifei. Mechanism by which diabetes exacerbates intervertebral disc degeneration [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(27): 5907-5913. |
| [12] | Sun Yahui, Wang Yufeng, Guo Chao, Yao Junjie, Ji Yuanyuan, Li Zhongxu, Lou Huijuan, Jiang Jinglei, Sun Yiping, Xu Jing, Cong Deyu. Effect of massage on extracellular matrix collagen deposition in skeletal muscle of type 2 diabetic rats [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(26): 5549-5555. |
| [13] | Guo Sunlin, Hong Enda, Dai Xinhua, Lin Xi, Peng Zhiyi, Cheng Yingxiong, Fan Linyan. Effect of Bushen Jianpi Formula on bone metabolism and bone microarchitecture in ovariectomized rats [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(26): 5588-5594. |
| [14] | Chen Yixin, Lu Yan, Zhang Xuan, Chen Xiaoli, Tan Liangyuan, Xu Zhangjie, Chen Wanglong, Su Shaoting, Liang Jiyao, Zhou Honghai. Mechanism by which Tongan Decoction regulates synovial macrophage polarization in rats with knee osteoarthritis [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(26): 5621-5631. |
| [15] | Yu Qinghe, Cai Ziming, Tian He, Li Pian, Ruan Ye, Liang Jinzhu, Lin Shuhui, Lin Wenping. Formononetin inhibits lipopolysaccharide-induced inflammation in nucleus pulposus mesenchymal stem cells [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(25): 5328-5334. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||