Chinese Journal of Tissue Engineering Research ›› 2025, Vol. 29 ›› Issue (2): 419-429.doi: 10.12307/2025.235
Previous Articles Next Articles
Epigenetic changes and exercise regulation: mechanisms underlying skeletal muscle aging and improvement
Fan Rao, Kong Jianda, Li lin, Zhai teng, Yang zirou, Zhu Lei
- Institute of Physical Education, Qufu Normal University, Jining 272000, Shandong Province, China
-
Received:2023-12-19Accepted:2024-02-04Online:2025-01-18Published:2024-05-27 -
Contact:Zhu Lei, PhD, Doctoral supervisor, Institute of Physical Education, Qufu Normal University, Jining 272000, Shandong Province, China -
About author:Fan Rao, Master candidate, Institute of Physical Education, Qufu Normal University, Jining 272000, Shandong Province, China
CLC Number:
Cite this article
Fan Rao, Kong Jianda, Li lin, Zhai teng, Yang zirou, Zhu Lei . Epigenetic changes and exercise regulation: mechanisms underlying skeletal muscle aging and improvement [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(2): 419-429.
share this article
Add to citation manager EndNote|Reference Manager|ProCite|BibTeX|RefWorks
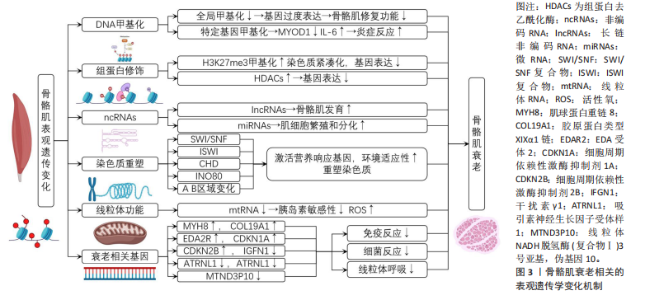
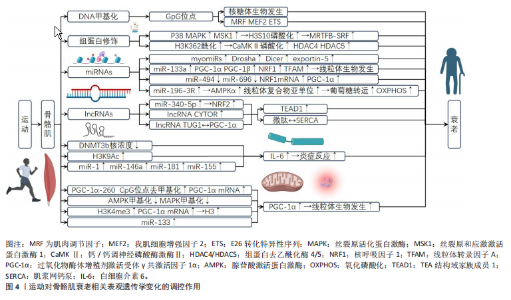
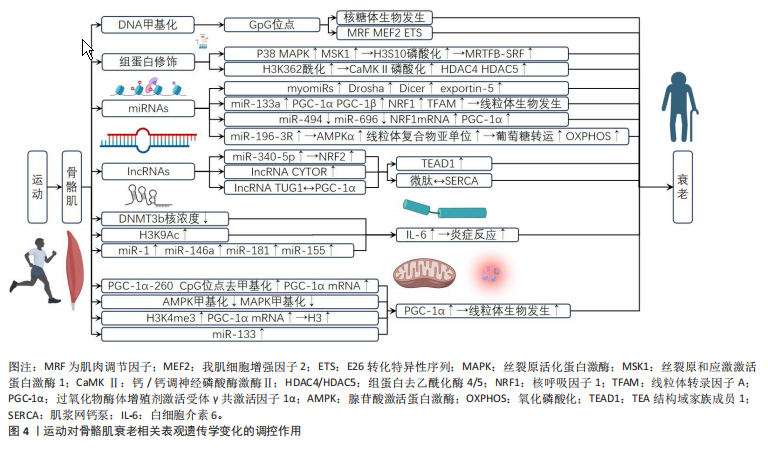
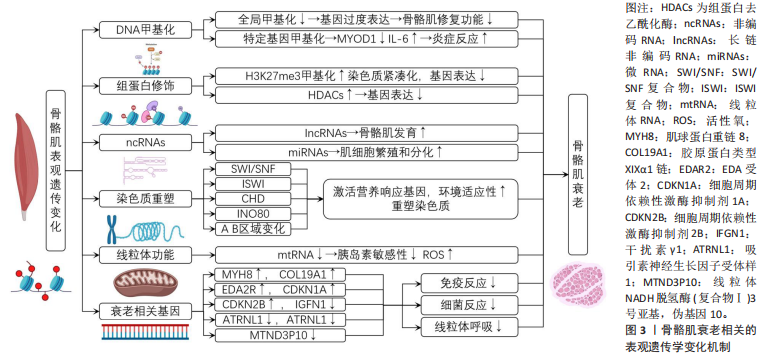
2.1 表观遗传学变化概述 表观遗传学是指基因序列不发生改变的情况下,基因表达和功能受到调控的现象,这一领域的研究揭示了环境因素、生活方式和遗传因素之间如何相互作用,影响生物体的生长发育、行为和疾病风险[1]。染色质重塑是表观遗传学中一个重要的方面,它是指染色质结构发生改变,从而影响基因的可接近性和表达,染色质重塑因子可以促进或抑制某些基因的表达,从而影响细胞的状态和功能[2-6]。此外,甲基化和乙酰化也是表观遗传学的重要变化[7-8],它们通过影响DNA和染色质的修饰来调控基因表达。除了DNA甲基化和组蛋白修饰外,非编码RNA和非编码核糖核酸也起着关键作用,这些非编码RNA分子可以与目标mRNA结合,影响其稳定性和翻译效率,从而调控基因表达[9]。此外,蛋白质修饰也是表观遗传学变化的一个重要方面,它在蛋白质分子上添加化学基团,从而改变蛋白质的结构和功能[10]。 总的来说,表观遗传学变化涉及许多复杂的调控机制,包括染色质重塑、DNA甲基化、组蛋白修饰、非编码RNA和非编码核糖核酸等,这些机制在生物体的生长发育、行为和疾病风险中发挥着重要作用。通过深入了解表观遗传学变化,可以更好地理解环境、生活方式和遗传因素如何相互作用,并为预防和治疗疾病提供新的思路。 2.2 骨骼肌衰老相关的表观遗传学变化机制 骨骼肌衰老过程中的表观遗传学变化包括DNA甲基化、组蛋白修饰、非编码RNA调控、染色质重塑以及衰老相关基因的表达改变。DNA甲基化的变化可能导致基因过度表达,影响骨骼肌的维持和修复能力。组蛋白修饰的改变影响基因表达和细胞功能,可能与肌肉细胞的增殖和分化能力降低有关。非编码RNA通过调节基因的表达和蛋白的功能影响肌肉的再生和衰老。染色质重塑通过改变染色质结构和基因表达模式影响肌肉细胞的功能和衰老过程。线粒体功能的改变可能是由于线粒体DNA的表观遗传学变化,这可能导致能量代谢的下降和细胞死亡过程的改变。此外,衰老过程中特定基因的表达水平发生变化,这些基因与细胞周期抑制、能量代谢、细胞衰老和肌肉发育等方面有关。骨骼肌衰老相关的表观遗传学变化机制见图3。 2.2.1 DNA甲基化 在骨骼肌衰老的过程中,DNA甲基化作为一种关键的表观遗传学机制,发挥着重要作用。DNA甲基化主要涉及在胞嘧啶环的第5位加入甲基基团,这种修饰通常发生在CpG岛(富含胞嘧啶和鸟嘌呤碱基对的DNA区域)上。随着年龄的增长,骨骼肌细胞中的全局DNA甲基化水平出现变化,这直接影响基因表达和细胞功能[7]。研究发现,骨骼肌组织中的全局DNA甲基化水平随年龄增长而下降,这种全局性的甲基化水平下降可能导致某些基因过度表达,从而影响骨骼肌维持和修复的能力[11]。例如,与年轻人的骨骼肌相比,老年人的骨骼肌中某些与细胞增殖和分化相关的基因表现出低甲基化状态,这可能是肌肉功能下降的原因之一[12]。 除了全局甲基化水平的变化,特定基因的甲基化状态也在衰老过程中发生改变。MYOD1基因作为肌肉细胞分化的关键调控因子,其启动子区域的甲基化水平在衰老肌肉中有所增加,导致该基因表达下降,影响肌肉再生能力。此外,与细胞老化和炎症反应相关的基因,如白细胞介素6和肿瘤坏死因子α,也表现出甲基化水平的变化,这可能与衰老过程中的慢性低度炎症状态有关[13]。DNA甲基化在调节骨骼肌衰老中的具体机制是多方面的:一方面,甲基化状态的改变影响基因的直接表达;另一方面,它还可能通过改变染色质的开放程度间接影响基因表达。此外,DNA甲基化与其他表观遗传学调控机制(如组蛋白修饰)的相互作用也在衰老过程中发挥作用[14]。 然而,目前关于DNA甲基化在骨骼肌衰老中的作用仍然是一个活跃的研究领域。未来的研究需要更深入地探究甲基化变化与肌肉功能下降之间的具体联系,并考察这些变化在不同个体和不同肌肉类型中的异同。此外,探索调节这些甲基化变化的潜在干预策略,可能为延缓或逆转肌肉衰老提供新的途径。 2.2.2 组蛋白修饰 组蛋白修饰是骨骼肌衰老过程中的一个关键表观遗传学机制,这些修饰主要包括组蛋白甲基化和乙酰化,它们在调节基因表达和细胞功能方面发挥着重要作用[4-10]。在衰老过程中,组蛋白的甲基化和乙酰化水平发生变化,这些变化与基因表达调控和染色质结构改变密切相关[10]。研究表明,在老年骨骼肌中,特定组蛋白位点(如H3K27me3)的甲基化水平增加,这可能与染色质的紧凑化和基因表达降低有关[10-14]。组蛋白修饰的变化不仅影响肌肉细胞的基因表达,还可能影响肌肉细胞的功能。例如,某些组蛋白修饰的改变可能与肌肉细胞的增殖和分化能力降低有关。此外,这些修饰可能也与肌肉细胞对应激反应的调整有关[15]。组蛋白修饰不仅受遗传因素的影响,还受环境因素的调控。例如,运动可能通过抑制组蛋白去乙酰化酶的功能,改变肌肉中关键基因的DNA甲基化和组蛋白修饰,从而影响基因表达模式[2-8]。 2.2.3 非编码RNA的调控 非编码RNA在骨骼肌衰老过程中扮演着重要角色,这类RNA包括长链非编码RNAs(long noncoding RNAs,lncRNAs)、微小RNA(microRNA,miRNA)等,它们通过多种方式调控肌肉的生长、修复和疾病发 展,lncRNA和miRNA通过调节基因的表达和蛋白的功能来影响肌肉的再生和衰老[16]。例如,某些lncRNA可以调控与肌肉发育相关的基因,而miRNA则可以通过调节靶标基因来影响肌肉细胞的增殖和分化[17]。在肌肉损伤修复过程中,非编码RNA也发挥着关键作用,其可以调节肌肉干细胞的活性和分化能力,从而影响肌肉的修复和重建[18]。非编码RNA的表达和功能受到多种因素的影响,包括年龄、运动、饮食和其他环境因素[19]。这些因素可以通过改变非编码RNA的表达模式,进而影响肌肉的衰老和功能。 2.2.4 染色质重塑 染色质重塑在骨骼肌衰老的表观遗传学变化中扮演着核心角色,它涉及到染色质结构的改变,从而影响基因的表达和细胞功能。染色质重塑涉及多种类型,包括SWI/SNF、ISWI、CHD和INO80家族的ATP依赖性染色质重塑复合体,其中,SWI/SNF复合体对于激活营养响应基因和维持细胞对环境适应能力至关重要,SWI/SNF还通过重塑染色质并促进更开放的染色质配置来调节转录[2]。在衰老过程中,染色质区域的可及性发生变化,这与衰老相关基因的表达程序正相关。例如,增加可及性区域(IARs)主要存在于与衰老发生和发展相关的基因中,而减少可及性区域(DARs)主要存在于与衰老引起的功能下降相关的基因中[2]。人类细胞中的染色质被空间分隔为A和B两个区域,同一区域的染色质之间有更频繁的相互作用。随着细胞衰老,染色质的高级结构和异染色质与核膜的分离被观察到。例如,核纤层蛋白功能的丧失导致在抑制性LAD结构中标记的H3K9甲基化组成性异染色质的去抑制[20]。 此外,染色质的特定变化与衰老过程紧密相关。例如,Hutchinson-Gilford早衰综合征和Werner综合征等衰老模型中观察到异染色质的损失,这与H3K9甲基化和HP1蛋白水平的降低有关,这些改变导致DNA损伤的积累和异常转录本的表达[20]。与染色质重塑相关的另一个重要方面是DNA甲基化。其中,“Horvath时钟”描述了DNA甲基化改变的模式,这是目前监测衰老和预测人类预期寿命一个有希望的分子标记,全基因组CpG甲基化的全球性下降被认为是衰老中的主要事件,这与异染色质损失期间的变化可能存在因果关联[21]。 总体来说,染色质重塑在骨骼肌衰老的表观遗传学变化中起着关键作用,它通过改变染色质结构和基因表达模式影响肌肉细胞的功能和衰老过程。 2.2.5 线粒体功能改变 线粒体在细胞的能量代谢和死亡过程中扮演着至关重要的角色。随着年龄的增长,线粒体DNA可能会发生表观遗传学变化,从而影响其功能,这些变化与骨骼肌的衰老密切相关。研究发现,在老年人中,线粒体功能的下降与身体活动量的减少有关,这可能导致运动效率降低。例如,老年人在进行次最大运动测试时,无论是在静息状态还是运动状态,呼吸交换比与年轻人相比没有显著差异,但他们的运动效率显著降低[22]。此外,年龄增长还会导致全身胰岛素敏感性降低,这可能部分归因于瘦体质量的差异[22]。在更详细的层面上,线粒体在衰老过程中会出现功能性改变。在不同类型肌肉纤维中,老化会影响线粒体的含量、呼吸和活性氧的释放,这种影响可以通过线粒体对钙挑战的敏感性来观察,其中快肌纤维丰富的肌肉在老化过程中对线粒体的渗透转换更为敏感[23]。 总的来说,随着年龄的增长,线粒体功能的改变可能是由于线粒体DNA的表观遗传学变化,这些变化可能导致能量代谢下降和细胞死亡过程的改变,进而影响骨骼肌的健康和功能。 2.2.6 衰老相关基因的表达改变 在衰老过程中,骨骼肌中的基因表达发生显著变化,这些变化与衰老相关的表观遗传学改变紧密相关。研究发现,随着年龄的增长,特定基因的表达水平会发生变化,这些基因通常与细胞周期抑制、能量代谢、细胞衰老和肌肉发育等方面有关。一项研究发现,在衰老过程中,人类骨骼肌中多达1 442个基因表现出差异表达,其中包括MYH8、COL19A1、EDA2R、CDKN1A和CDKN2B等基因的上调,以及IGFN1、MTND3P10、ATRNL1和PVALB等基因的下调[24]。这些基因的变化与免疫反应、细菌反应的上调以及线粒体呼吸的下调相关。另一项研究则关注了与衰老密切相关的前20个RNA(包括编码蛋白和非编码RNA),这些RNA包括与细胞周期抑制相关的CDKN2B、与胰岛素信号和脂代谢有关的IRS2、与肌肉发育重要的NR2F2,以及与平滑肌分化相关的LPP等[25]。此外,SHAVLAKADZE等[26]发现,在小鼠和大鼠中,与年龄相关的基因表达变化包括线性或逻辑模式的变化。这些研究揭示了在衰老过程中涉及的多种通路,例如代谢和线粒体功能的降低。 2.3 运动对骨骼肌衰老相关表观遗传学变化的调控作用 运动在缓解与衰老相关的生物过程中的基本作用至关重要,定期进行运动一直与延长寿命和降低与年龄相关的疾病风险密切相关。运动以一种延缓衰老过程中典型衰退的方式影响多个生理系统,这些影响包括改善心血管功能、增强肌肉力量和质量、改善代谢调节以及增强免疫反应。运动对衰老影响的基石为深入探讨这些益处如何在分子层面上延伸,特别是通过表观遗传修饰提供了理论框架。研究运动如何通过表观遗传改变来产生这些效应的具体机制,包括DNA甲基化和组蛋白修饰,阐明了物理活动与衰老过程之间的复杂关系。运动对骨骼肌衰老相关表观遗传学变化的调控作用见图4。 2.3.1 运动改变骨骼肌的DNA甲基化 运动对骨骼肌的DNA甲基化具有显著影响。运动能够使组蛋白修饰和DNA甲基化发生变化,这与DNA甲基化在基因组中的CpG位点上发生的胞嘧啶残基的5’端融合密切相关。DNA甲基化在顺式作用调节区域通常与基因沉默有关,但POU和NFAT家族的转录因子等某些转录因子更倾向于与甲基化的CpG序列相关[27]。 运动训练能够在肌肉中诱导DNA的低甲基化和高甲基化,主要发生在基因体、基因间区域和增强子区域。FIGUEIREDO等[28]的研究发现,抗阻训练可以导致核糖体生物发生,这与核糖体DNA基因剂量、增强体和非正则相关,但与启动子无关。这表明运动响应下骨骼肌遗传和核糖体生物发生调控的信息,同时揭示了核糖体DNA剂"
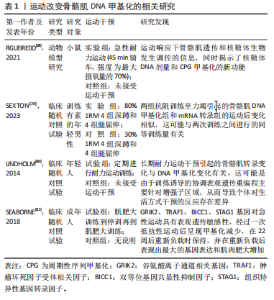
量和CPG甲基化的新功能。此外,SEXTON等[29]的研究表明,在抗阻运动后的3 h内,CpG位点甲基化变化的数量最多,mRNA转录本倒转DNA甲基化模式的百分比亦最高,这些变化在6 h内开始恢复到基线甲基化水平,表明骨骼肌基因组是动态的,在恢复运动中受到紧密调控。此外,骨骼肌在进行长期耐力运动干预时发生的转录改变与DNA甲基化变化有关,主要取决于MRF、MEF2和ETS蛋白的转录调节因子结合基序[30]。然而,SEABORNE等[31]则发现,在抗阻运动后,选择性的DNA低甲基化和高甲基化仍然存在,表明不同运动方式具有特异性的DNA甲基化特征,可能在肌肉中引导特定的运动后转录程序。 综上所述,运动可以显著影响骨骼肌的DNA甲基化,而不同运动方式具有特异性的DNA甲基化特征。然而,这一领域的研究尚处于初步阶段,急性运动和不同类型运动对DNA甲基化的具体影响仍有待深入探讨。因此,后续研究可以关注运动诱导的DNA甲基化变化在不同运动方式中的差异,以及这些变化在肌肉适应过程中的作用。运动改变骨骼肌DNA甲基化的相关研究见表1。 2.3.2 运动促进骨骼肌的组蛋白修饰 运动对骨骼肌的作用是通过改变肌肉内的代谢环境和上游信号传导来改变染色质的状态,从而影响基因表达[32]。组蛋白修饰是指在组蛋白尾部或核心结构中的氨基酸上发生的表观遗传"

学修饰,它可以在染色质的开放或关闭状态之间转换,具体取决于修饰的类型、目标残基和组蛋白蛋白质[32]。研究表明,一次急性耐力运动可以增加染色质上H3K36的乙酰化[33],而H3K36是一种开放染色质的标记,能促进钙/钙调素依赖性蛋白激酶Ⅱ的磷酸化以及转录抑制类 IIHDAC4和HDAC5的核外定位[34]。然而,NAGHAVI MOGHADAM等[35]的研究发现,在非运动员男性进行高强度运动并补充人参后,骨骼肌中的H3K36甲基化并没有显著增加,这说明其他机制可能参与了高强度运动和人参对骨骼肌功能的影响,而不仅仅是H3K36乙酰化的变化。此外,高强度抗阻运动还可以通过p38 MAPK和丝裂原活化蛋白激酶1在人体肌肉中短暂刺激肌核H3S10的磷酸化[36],该信号轴对小鼠体内收缩后细胞核MRTFB-SRF靶基因的上调和蛋白质合成亦具有重要作用。 综上所述,运动通过上游信号传导和改变肌肉内的代谢环境来影响骨骼肌的组蛋白修饰状态,从而影响基因表达。然而,对于不同的修饰类型和目标残基,在运动后的修饰效果可能存在差异。因此,后续的研究可以关注不同运动类型和不同修饰效应之间的关系,进一步揭示运动对骨骼肌表观遗传学的影响机制。运动促进骨骼肌的组蛋白修饰的相关研究见表2。 2.3.3 运动调控骨骼肌的miRNA miRNA作为表观遗传学调控分子,通过转录后调节改变基因表达并影响蛋白质翻译过程。其中,肌肉特异性miRNAs来源于骨骼肌或心肌,与运动调控密切相关。当前,已知与骨骼肌相关的肌肉特异性miRNAs共有7种,包括miR-1、miR-133a、 miR-133b、miR-206、miR-208b、miR-486及miR-499,其表现水平通常与运动强度相关[37]。研究证实,运动不仅能上调myomiRs(肌肉特异性miRNAs)的表达,还能增加与miRNA成熟相关的蛋白质(如Drosha、Dicer和exportin-5) 的表达[38]。此外,耐力运动还能提高miR-133a的水平,"


并增强线粒体生物发生调节因子过氧化物酶体增殖物活化受体γ共激活因子1α(peroxisome proliferator-activated receptor γ coactivator-1α,PGC-1α)、PGC-1β、NRF1和TFAM在野生型小鼠骨骼肌中的表达,进而促进线粒体生物发生[39]。此外,陈圣菊等[40]发现,中强度运动下小鼠骨骼肌中miR-1a和miR-133a的表达显著下降,而高强度运动下则显著上升,这说明不同强度的运动导致miR-1a和miR-133a的表达情况具有差异性。此外,运动训练能够使老年鼠骨骼肌miR-1和miR-133a的表达明显提升[41]。 除肌肉特异性miRNAs外,运动还能通过调节其他类型miRNA的表达来调控线粒体生物发生,进而调控骨骼肌的表观遗传学。研究发现,跑步运动可降低小鼠腓肠肌中miR-494和miR-696的表达,同时增加NRF1 mRNA和PGC-1α蛋白的表达[42],这表明运动可能通过调节miRNA表达来调节线粒体生物发生。另有研究发现,耐力运动能够使miR-19b-3p在骨骼肌中的表达上升,且 miR-19b-3p过表达可以增加AMPKα和线粒体复合物亚单位的表达,促进葡萄糖转运和氧化磷酸化呼吸[43]。此外,研究发现,在不同性别中,运动后唾液中miR-4675、 miR-6745和miR-6746-3p的表达具有明显差异[44]。这说明运动对miRNA的影响存在性别上的差异,但当前证据仅存在于唾液相关研究中,尚缺乏关于运动引起不同性别间骨骼肌miRNA表达差异性的直接证据。 总之,运动对miRNA的表达具有显著调控作用,不同强度和类型的运动对miRNA的表达影响各异,说明运动调控骨骼肌miRNA的表达具有复杂的机制。然而,尚需进一步研究不同强度和性别运动对骨骼肌miRNA表达的影响,以深入了解运动调控骨骼肌表观遗传学的生物学机制。运动调控骨骼肌miRNA的相关研究见表3。 2.3.4 运动调控骨骼肌的lncRNAs lncRNAs被认为是重要的表观遗传学调节因子,通过海绵活性可以中和miRNAs的作用,同时还能够引导DNA甲基化和组蛋白修饰,从"


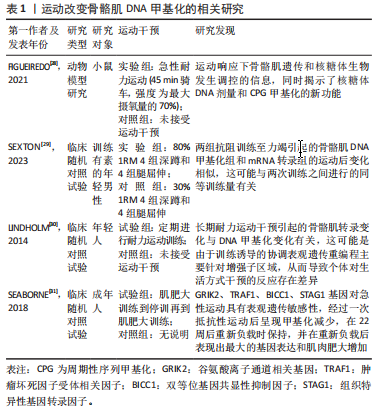
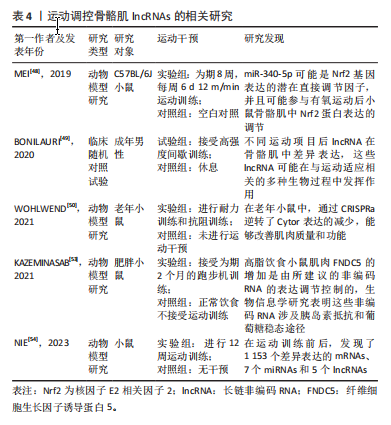
而调节基因表达[45]。此外,lncRNAs可以作为竞争性内源RNA吸附并抑制结合的miRNAs的活性,有效解除这些miRNAs对其他靶标的抑制作用[46]。在运动调控骨骼肌的过程中,lncRNAs主要通过表观遗传学、转录和转录后水平等不同机制,调节基因表达,进而影响运动的效应。 研究证明,有氧运动可以调控某些lncRNAs的表达[47]。MEI等[48]研究发现,在小鼠骨骼肌中,miR-340-5p在有氧运动后对NRF2表达的转录后调控中发挥作用,这证实了有氧运动调控miR-340-5p在骨骼肌中对NRF2的调控中起到了重要的作用,但尚不清楚该miRNA在其他生物体或不同细胞类型中的功能和作用机制。此外,Bonilauri等[49]的研究了不同运动训练方案下lncRNA的表达及其与运动适应过程的关联模式,其发现不同的训练方案会导致非常特异的lncRNA表达模式,不同训练方案之间的表达差异明显,但在不同运动类型之间共有的lncRNA较少。然而,这些lncRNA在运动适应过程中的具体功能和相互作用机制尚不明确。已知研究表明,lncRNA对耐力训练、抗阻训练和高强度间歇训练具有不同的表达谱。 WOHLWEND等[50]研究发现,lncRNA CYTOR在骨骼肌中受耐力和抗阻训练诱导,通过抑制TEA结构域家族成员1的转录活性,调控快速收缩纤维的形成;CYTOR mRNA在衰老过程中减少,过表达该lncRNA可以增加ⅡA型和ⅡB型纤维的数量,从而改善老年小鼠的握力和上坡跑步表现。此外,lncRNA还可能包含短开放读框,编码微肽,这些肌肉特异性的微肽和Myoregulin通过竞争性相互作用与肌浆/内质网钙离子转运ATP酶相互影响,进而调节肌浆/内质网钙离子动力学和肌肉收缩能力的正负方面[51-52]。 然而,目前对运动调控骨骼肌的lncRNAs研究仍处于初步阶段,尚不清楚所有调控机制和具体作用。 KAZEMINASAB等[53]对肥胖小鼠进行有氧运动,发现其腓肠肌中与FNDC5相关的非编码RNA的表达影响。此外,NIE等[54]构建了一个与运动训练反应的骨骼肌新型内源竞争RNA网络,确定了运动前后样本中不同表达的lncRNA、miRNA和mRNA。TREWIN等[55]发现了lncRNA TUG1在骨骼肌对运动的转录反应中与PGC-1α的相互作用。这些研究结果揭示了lncRNA在运动适应和骨骼肌生物学方面的重要性,但具体的调控机制和作用仍需进一步研究。 综上,lncRNAs在运动调控骨骼肌过程中发挥着重要作用,其通过不同的机制如表观遗传学、转录和转录后水平调节基因表达,进而影响运动的效应。然而,目前对运动调控骨骼肌的lncRNAs研究仍处于初步阶段,尚不清楚所有调控机制和具体作用。运动调控骨骼肌lncRNAs的相关研究见表4。 "

2.3.5 运动调控骨骼肌表观遗传学变化 运动可能通过改变表观遗传学修饰来上调白细胞介素6等细胞因子的表达。其中运动可能直接调节肌纤维分泌基因的DNA甲基化、组蛋白修饰和miRNA表达,并可以调节基因表达[56]。研究报道,运动诱导的白细胞介素6水平升高伴随着外周血单核细胞中DNA甲基转移酶3b核浓度减少,并且在运动后DNMT3b核浓度减少与白细胞介素6血浆浓度增加之间存在显著相关性[56-57],这说明运动可能通过减少DNMT3b的核浓度和减少DNA甲基化来增加白细胞介素6的表达。另外,白细胞介素6的表达也受到组蛋白乙酰化的调节。骨骼肌中白细胞介素6合成和释放的负表观遗传学调节剂HDAC5通过下调白细胞介素6启动子的H3K9Ac修饰来抑制白细胞介素6的表达。白细胞介素6的表达也受到miRNA的调节[58]。运动后血浆中高表达的miR-1、miR-146a、miR-181和miR-155与白细胞介素6水平呈正相关[59],这表明miRNA可能参与到白细胞介素6表达的调控中。YUAN等[60]的研究表明,miR-223和白细胞介素6靶向相关,能够直接与白细胞介素6的3个非翻译区结合并抑制白细胞介素6的表达。以上证据表明,白细胞介素6的表达受到多种表观遗传学修饰的调节。此外,DE GONZALO-CALVO等[61]的观察性研究发现,马拉松比赛后,miR-223-3p和miR-223-5p的水平立即升高,并在24 h 后恢复到基础水平,这一发现揭示了包括miR-223在内的c-inflammamiRs模式可能与炎症过程的调节密切相关。 运动不仅通过表观遗传学变化调节白细胞介素6等细胞因子的表达,还影响其他多种由骨骼肌分泌的细胞因子。研究表明,骨骼肌在运动过程中还能分泌如白细胞介素15、肌肉生成蛋白(Myostatin)、白细胞介素8和骨形态生成蛋白7等细胞因子,这些因子在代谢调节、肌肉生长、炎症反应以及其他重要生理过程中发挥作用。白细胞介素15长期以来被认为与肌肉代谢和生长密切相关。一项研究表明,白细胞介素15在促进脂肪氧化和减少脂肪储存方面起着关键作用[62]。另外,Myostatin是一种负调节肌肉生长的因子,运动能降低其表达水平,从而促进肌肉生长[63]。白细胞介素8在运动诱导的血管新生中起到了关键作用。一项研究发现,白细胞介素8的表达在运动后增加,这与血管新生和肌肉血流增加有关[64]。 骨形态生成蛋白7也在运动后的肌肉修复和重建中发挥作用,研究显示骨形态生成蛋白7有助于促进肌肉再生和修复[65]。 总之,运动可以通过改变表观遗传学修饰来调节细胞因子白细胞介素6的表达,这种调节可能通过直接影响肌纤维分泌基因的DNA甲基化、组蛋白修饰和miRNA表达来实现,并且可以调节基因表达。运动还可以影响其他多种由骨骼肌分泌的细胞因子,如白细胞介素15、Myostatin、白细胞介素8和骨形态生成蛋白7,这些因子在代谢调节、肌肉生长、炎症反应和其他生理过程中发挥重要作用。运动通过细胞因子调控表观遗传学变化的相关研究见表5。 2.3.6 运动调控骨骼肌的线粒体功能:PGC-1α的作用 运动能够调控骨骼肌的线粒体相关的受体蛋白和信号通路,"


在这些受体蛋白和信号通路中,PGC-1α起着关键作用[66],且对骨骼肌的表观遗传学变化起到潜在的影响。运动诱导的线粒体相关DNA甲基化在运动的健康益处中起着重要作用。BAJPEYI等[67]的研究发现,在进行急性运动后,出现骨骼肌PGC-1α-260 CpG位点的去甲基化、PGC-1α mRNA表达上调以及线粒体产生的增加。此外,MAASAR等[68]发现,年轻男性运动员在30 min运动后,与腺苷酸5’单磷酸激活蛋白激酶、丝裂原激活蛋白激酶、蛋白质结合、胰岛素和轴突导向通路相关的基因甲基化降低,PGC-1α表达增加,这说明运动可以通过改变骨骼肌中线粒体生物发生相关基因的DNA甲基化来影响线粒体生物发生。 除了DNA甲基化,运动也会影响其他表观遗传学修饰,如组蛋白甲基化、乙酰化和泛素化等,这些都可以调控运动诱导的PGC-1α基因表达。LOCHMANN等[69]的研究表明,在运动中PGC-1α启动子转录活性标记物H3K4me3水平会增加2-4倍,并伴随着PGC-1α mRNA水平的增加。这表明运动可能通过增加PGC-1α启动子中的H3K4me3修饰,上调PGC-1α蛋白表达,并增加线粒体生物合成。此外,研究发现大鼠在急性运动后2 h,PGC-1α近端启动子上的组蛋白3乙酰化水平会随着PGC-1α mRNA水平的增加而增加,且这种变化还具有肌纤维类型的依赖性[70]。这些研究将运动与线粒体相关基因组蛋白的翻译后修饰联系起来,表明运动诱导的线粒体相关基因翻译后修饰的调控可能是运动诱导的骨骼肌适应性反应的重要环节。NIE等[39]证实了6周耐力运动可增加miR-133a水平,并促进野生型小鼠骨骼肌中线粒体生物生成调节剂PGC-1α、PGC-1β、NRF1和TFAM的表达,从而促进线粒体生物生成。 综上所述,运动通过调控PGC-1α的表达和线粒体生物合成,可能对骨骼肌的表观遗传学变化产生影响。然而,这些发现仍需进一步的研究来证实和扩展。运动通过PGC-1α调控骨骼肌线粒体功能的相关研究见表6。"

| [1] PEIXOTO P, CARTRON PF, SERANDOUR AA, et al. From 1957 to Nowadays: A Brief History of Epigenetics. Int J Mol Sci. 2020;21(20): 7571. [2] WANG K, LIU H, HU Q, et al. Epigenetic regulation of aging: implications for interventions of aging and diseases. Signal Transduct Target Ther. 2022;7(1):374. [3] SCHIAFFINO S, REGGIANI C. Fiber types in mammalian skeletal muscles. Physiol Rev. 2011;91(4):1447-1531. [4] 杨园,叶啟发,杨翼.运动调节衰老的表观遗传机制研究进展[J]. 中国运动医学杂志,2019,38(8):712-716. [5] BARRèS R, ZIERATH JR. The role of diet and exercise in the transgenerational epigenetic landscape of T2DM. Nat Rev Endocrinol. 2016;12(8):441-451. [6] 曹红, 吴迪, 魏瑜,等. 染色质结构蛋白变异与人类疾病[J]. 中国科学:生命科学,2023,53(11):1575-1594. [7] TURNER DC, GORSKI PP, MAASAR MF, et al. DNA methylation across the genome in aged human skeletal muscle tissue and muscle-derived cells: the role of HOX genes and physical activity. Sci Rep. 2020;10(1):15360. [8] 尹苗苗,牛燕媚,傅力.组蛋白去乙酰化酶在骨骼肌能量代谢调控中的潜在作用[J]. 中国糖尿病杂志,2013,21(2):189-192. [9] MBADHI MN, TANG JM, ZHANG JX. Histone Lysine Methylation and Long Non-Coding RNA: The New Target Players in Skeletal Muscle Cell Regeneration. Front Cell Dev Biol. 2021;9:759237. [10] 贾梅,浦敏铁.组蛋白修饰调控衰老过程的机制研究进展[J]. 中国科学:生命科学,2019,49(7):806-813. [11] VOISIN S, HARVEY NR, HAUPT LM, et al. An epigenetic clock for human skeletal muscle. J Cachexia Sarcopenia Muscle. 2020;11(4):887-898. [12] MASSENET J, GARDNER E, CHAZAUD B, et al. Epigenetic regulation of satellite cell fate during skeletal muscle regeneration. Skelet Muscle. 2021;11(1):4. [13] PONSUKSILI S, TRAKOOLJUL N, BASAVARAJ S, et al. Epigenome-wide skeletal muscle DNA methylation profiles at the background of distinct metabolic types and ryanodine receptor variation in pigs. BMC Genomics. 2019;20(1):492. [14] SAILANI MR, HALLING JF, MøLLER HD, et al. Lifelong physical activity is associated with promoter hypomethylation of genes involved in metabolism, myogenesis, contractile properties and oxidative stress resistance in aged human skeletal muscle. Sci Rep. 2019; 9(1):3272. [15] WANG Y, YUAN Q, XIE L. Histone Modifications in Aging: The Underlying Mechanisms and Implications. Curr Stem Cell Res Ther. 2018;13(2): 125-135. [16] 陈晓彤,赵文龙,孙林玉,等.非编码RNA来源的小肽:“微不足道”却“功能强大”[J]. 中山大学学报(自然科学版)(中英文), 2023,62(3):1-13. [17] ZHAO Y, CHEN M, LIAN D, et al. Non-Coding RNA Regulates the Myogenesis of Skeletal Muscle Satellite Cells, Injury Repair and Diseases. Cells. 2019;8(9):988. [18] 孔健达, 穆玉晶, 朱磊,等. 骨骼肌再生过程中卫星细胞调控机制及其生态位信号的作用[J]. 中国组织工程研究,2024,28(7): 1105-1111. [19] LIU Q, DENG J, QIU Y, et al. Non-coding RNA basis of muscle atrophy. Mol Ther Nucleic Acids. 2021;26:1066-1078. [20] LEE JH, KIM EW, CROTEAU DL, et al. Heterochromatin: an epigenetic point of view in aging. Exp Mol Med. 2020;52(9):1466-1474. [21] BELL CG, LOWE R, ADAMS PD, et al. DNA methylation aging clocks: challenges and recommendations. Genome Biol. 2019;20(1):249. [22] GREVENDONK L, CONNELL NJ, MCCRUM C, et al. Impact of aging and exercise on skeletal muscle mitochondrial capacity, energy metabolism, and physical function. Nat Commun. 2021;12(1):4773. [23] HEPPLE RT. Mitochondrial involvement and impact in aging skeletal muscle. Front Aging Neurosci. 2014;6:211. [24] PEREZ K, CIOTLOS S, MCGIRR J, et al. Single nuclei profiling identifies cell specific markers of skeletal muscle aging, frailty, and senescence. Aging (Albany NY). 2022;14(23):9393-9422. [25] TUMASIAN RA, 3RD, HARISH A, KUNDU G, et al. Skeletal muscle transcriptome in healthy aging. Nat Commun. 2021;12(1):2014. [26] SHAVLAKADZE T, XIONG K, MISHRA S, et al. Age-related gene expression signatures from limb skeletal muscles and the diaphragm in mice and rats reveal common and species-specific changes. Skelet Muscle. 2023;13(1):11. [27] YIN Y, MORGUNOVA E, JOLMA A, et al. Impact of cytosine methylation on DNA binding specificities of human transcription factors. Science. 2017;356(6337):eaaj2239. [28] FIGUEIREDO VC, WEN Y, ALKNER B, et al. Genetic and epigenetic regulation of skeletal muscle ribosome biogenesis with exercise. J Physiol. 2021;599(13):3363-3384. [29] SEXTON CL, GODWIN JS, MCINTOSH MC, et al. Skeletal Muscle DNA Methylation and mRNA Responses to a Bout of Higher versus Lower Load Resistance Exercise in Previously Trained Men. Cells. 2023; 12(2):263. [30] LINDHOLM ME, MARABITA F, GOMEZ-CABRERO D, et al. An integrative analysis reveals coordinated reprogramming of the epigenome and the transcriptome in human skeletal muscle after training. Epigenetics. 2014;9(12):1557-1569. [31] SEABORNE RA, STRAUSS J, COCKS M, et al. Human Skeletal Muscle Possesses an Epigenetic Memory of Hypertrophy. Sci Rep. 2018;8(1): 1898. [32] SEABORNE RA, SHARPLES AP. The Interplay Between Exercise Metabolism, Epigenetics, and Skeletal Muscle Remodeling. Exerc Sport Sci Rev. 2020;48(4):188-200. [33] ŚWIATOWY WJ, ZIELIŃSKI J, OSIELSKA MA, et al. No dynamic changes in the expression of genes related to the epigenetic mechanism during acute exercise. J Appl Genet. 2023;64(1):81-87. [34] 张艺蓉,陈力方,王维蓉. HDACs去乙酰化酶非依赖性作用在疾病中的研究进展[J]. 生命科学,2023,35(5):583-591. [35] NAGHAVI MOGHADAM AA, SHIRAVAND M, REZAPOUR S, et al. Effect of a session of intensive exercise with ginseng supplementation on histone H3 protein methylation of skeletal muscle of nonathlete men. Mol Genet Genomic Med. 2019;7(5):e651. [36] SOLAGNA F, NOGARA L, DYAR KA, et al. Exercise-dependent increases in protein synthesis are accompanied by chromatin modifications and increased MRTF-SRF signalling. Acta Physiol (Oxf). 2020;230(1):e13496. [37] JACQUES M, HIAM D, CRAIG J, et al. Epigenetic changes in healthy human skeletal muscle following exercise- a systematic review. Epigenetics. 2019;14(7):633-648. [38] RUSSELL AP, LAMON S, BOON H, et al. Regulation of miRNAs in human skeletal muscle following acute endurance exercise and short-term endurance training. J Physiol. 2013;591(18):4637-4653. [39] NIE Y, SATO Y, WANG C, et al. Impaired exercise tolerance, mitochondrial biogenesis, and muscle fiber maintenance in miR-133a-deficient mice. Faseb J. 2016;30(11):3745-3758. [40] 陈圣菊,王飞,李文炯,等.小鼠运动敏感型血清miRNA的筛选[J]. 航天医学与医学工程,2018,31(2):224-228. [41] 陈彩珍,李德深,邱守涛,等.肌组织特异性microRNA介导的骨骼肌衰减机制及运动的干预研究[J]. 西安体育学院学报,2017, 34(6):705-713. [42] SUN Y, CUI D, ZHANG Z, et al. Voluntary wheel exercise alters the levels of miR-494 and miR-696 in the skeletal muscle of C57BL/6 mice. Comp Biochem Physiol B Biochem Mol Biol. 2016;202:16-22. [43] MASSART J, SJöGREN RJO, EGAN B, et al. Endurance exercise training-responsive miR-19b-3p improves skeletal muscle glucose metabolism. Nat Commun. 2021;12(1):5948. [44] HICKS SD, JACOB P, MIDDLETON FA, et al. Distance running alters peripheral microRNAs implicated in metabolism, fluid balance, and myosin regulation in a sex-specific manner. Physiol Genomics. 2018; 50(8):658-667. [45] KOPP F, MENDELL JT. Functional Classification and Experimental Dissection of Long Noncoding RNAs. Cell. 2018;172(3):393-407. [46] THOMSON DW, DINGER ME. Endogenous microRNA sponges: evidence and controversy. Nat Rev Genet. 2016;17(5):272-283. [47] WU YL, LIN ZJ, LI CC, et al. Epigenetic regulation in metabolic diseases: mechanisms and advances in clinical study. Signal Transduct Target Ther. 2023;8(1):98. [48] MEI T, LIU Y, WANG J, et al. miR‑340‑5p: A potential direct regulator of Nrf2 expression in the post‑exercise skeletal muscle of mice. Mol Med Rep. 2019;19(2):1340-1348. [49] BONILAURI B, DALLAGIOVANNA B. Long Non-coding RNAs Are Differentially Expressed After Different Exercise Training Programs. Front Physiol. 2020;11:567614. [50] WOHLWEND M, LAURILA PP, WILLIAMS K, et al. The exercise-induced long noncoding RNA CYTOR promotes fast-twitch myogenesis in aging. Sci Transl Med. 2021;13(623):eabc7367. [51] 陈雪飞,张靓. 肌浆网钙泵调节肽的研究进展[J]. 生命的化学, 2017,37(6):1005-1011. [52] 潘剑锋,尚方正,马荣,等.长非编码RNA编码微肽的研究进展[J]. 生物工程学报,2022,38(9):3194-3214. [53] KAZEMINASAB F, MARANDI SM, BAHARLOOIE M, et al. Aerobic exercise modulates noncoding RNA network upstream of FNDC5 in the Gastrocnemius muscle of high-fat-diet-induced obese mice. J Physiol Biochem. 2021;77(4):589-600. [54] NIE M, LIU Q, YAN C. Construction of a novel lncRNA-miRNA-mRNA competing endogenous RNA network in muscle in response to exercise training. Gen Physiol Biophys. 2023;42(2):123-133. [55] TREWIN AJ, SILVER J, DILLON HT, et al. Long non-coding RNA Tug1 modulates mitochondrial and myogenic responses to exercise in skeletal muscle. BMC Biol. 2022;20(1):164. [56] HUNTER DJ, JAMES L, HUSSEY B, et al. Impact of aerobic exercise and fatty acid supplementation on global and gene-specific DNA methylation. Epigenetics. 2019;14(3):294-309. [57] COCO M, PERCIAVALLE V, CAVALLARI P, et al. Effects of age and sex on epigenetic modification induced by an acute physical exercise. Medicine (Baltimore). 2017;96(44):e8325. [58] SANDONà M, CAVIOLI G, RENZINI A, et al. Histone Deacetylases: Molecular Mechanisms and Therapeutic Implications for Muscular Dystrophies. Int J Mol Sci. 2023;24(5):4306. [59] LI D, WANG P, WEI W, et al. Serum MicroRNA Expression Patterns in Subjects After the 5-km Exercise Are Strongly Associated With Cardiovascular Adaptation. Front Physiol. 2021;12:755656. [60] YUAN X, BERG N, LEE JW, et al. MicroRNA miR-223 as regulator of innate immunity. J Leukoc Biol. 2018;104(3):515-524. [61] DE GONZALO-CALVO D, DáVALOS A, MONTERO A, et al. Circulating inflammatory miRNA signature in response to different doses of aerobic exercise. J Appl Physiol (1985). 2015;119(2):124-134. [62] PEDERSEN BK, AKERSTRöM TC, NIELSEN AR, et al. Role of myokines in exercise and metabolism. J Appl Physiol (1985). 2007;103(3): 1093-1098. [63] HULSTON CJ, VENABLES MC, MANN CH, et al. Training with low muscle glycogen enhances fat metabolism in well-trained cyclists. Med Sci Sports Exerc. 2010;42(11):2046-2055. [64] PALMER MJ. Functional segregation of synaptic GABAA and GABAC receptors in goldfish bipolar cell terminals. J Physiol. 2006;577(Pt 1): 45-53. [65] WINBANKS CE, WEEKS KL, THOMSON RE, et al. Follistatin-mediated skeletal muscle hypertrophy is regulated by Smad3 and mTOR independently of myostatin. J Cell Biol. 2012;197(7):997-1008. [66] 李兆进, 郑鹏程, 孔健达,等. 基于不同组织和器官角度回顾PGC-1α在运动抗衰老中的作用[J]. 中国组织工程研究,2024,28(29): 4717-4725. [67] BAJPEYI S, COVINGTON JD, TAYLOR EM, et al. Skeletal Muscle PGC1α -1 Nucleosome Position and -260 nt DNA Methylation Determine Exercise Response and Prevent Ectopic Lipid Accumulation in Men. Endocrinology. 2017;158(7):2190-2199. [68] MAASAR MF, TURNER DC, GORSKI PP, et al. The Comparative Methylome and Transcriptome After Change of Direction Compared to Straight Line Running Exercise in Human Skeletal Muscle. Front Physiol. 2021;12:619447. [69] LOCHMANN TL, THOMAS RR, BENNETT JP, JR, et al. Epigenetic Modifications of the PGC-1α Promoter during Exercise Induced Expression in Mice. PLoS One. 2015;10(6):e0129647. [70] MASUZAWA R, KONNO R, OHSAWA I, et al. Muscle type-specific RNA polymerase II recruitment during PGC-1α gene transcription after acute exercise in adult rats. J Appl Physiol (1985). 2018;125(4):1238-1245. |
| [1] | Miao Jiahang, Ma Sheng, Li Qupeng, Yu Huilin, Hu Tianyu, Gao Xiao, Feng Hu. Cervical lordosis ratio can be used as a decision-making indicator for selection of posterior surgical approach for multi-level cervical spondylotic myelopathy [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(9): 1796-1802. |
| [2] | Wang Qiuyue, Jin Pan, Pu Rui . Exercise intervention and the role of pyroptosis in osteoarthritis [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(8): 1667-1675. |
| [3] | Lyu Liting, Yu Xia, Zhang Jinmei, Gao Qiaojing, Liu Renfan, Li Meng, Wang Lu. Bibliometric analysis of research process and current situation of brain aging and exosomes [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(7): 1457-1465. |
| [4] | Chen Yilin, Jiang Xiaobo, Qu Honglin, Liu Ruilian. General pattern of GSK3/Nrf2-regulated biological rhythms in organismal aging [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(6): 1257-1264. |
| [5] | He Yang, Tang Buyuan, Lu Changhuai. Molecular mechanisms of ligament flavum hypertrophy: analysis based on methylation sequencing and transcriptome integration [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(5): 1013-1020. |
| [6] | Wen Zixing, Xu Xin, Zhu Shengqun. Correlations between gastrocnemius morphology parameters and physical activity capacity in elderly females under high-frequency ultrasound [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(5): 1058-1063. |
| [7] | Zhang Xiongjinfu, Chen Yida, Cheng Xinyi, Liu Daihui, Shi Qin . Exosomes derived from bone marrow mesenchymal stem cells of young rats to reverse senescence in aged rat bone marrow mesenchymal stem cells [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(36): 7709-7718. |
| [8] | Sima Xinli, Liu Danping, Qi Hui. Effect and mechanism of metformin-modified bone marrow mesenchymal stem cell exosomes on regulating chondrocytes [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(36): 7728-7734. |
| [9] | Wang Simin, Zhang Dezhou, Zhao Jing, Wang Chaoqun, Li Kun, Chen Jie, Bai Xue, Zhao Hailong, Zhang Shaojie, Ma Yuan, Hao Yunteng, Yang Yang, Li Zhijun, Shi Jun, Wang Xing. Artificial intelligence and cervical spine image recognition: application prospects and challenges [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(33): 7231-7240. |
| [10] | Lin Shuqian, Zhao Xilong, Gao Jing, Pan Xinghua, Li Zian, Ruan Guangping. Comparison of biological characteristics of mouse bone marrow mesenchymal stem cells after interference and overexpression of telomere Cajal body protein-1 [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(31): 6616-6624. |
| [11] | Wang Jianxu, Dong Zihao, Huang Zishuai, Li Siying, Yang Guang. Interaction between immune microenvironment and bone aging and treatment strategies [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(30): 6509-6519. |
| [12] | Zhang Bochun, Li Wei, Li Guangzheng, Ding Haoqin, Li Gang, Liang Xuezhen, . Association between neuroimaging changes and osteonecrosis: a large sample analysis from UK Biobank and FinnGen databases [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(30): 6574-6582. |
| [13] | Song Haoran, Zhang Yuqiang, Gu Na, Zhi Xiaodong, Wang Wei. Visualization analysis of artificial intelligence in bone trauma research based on Citespace [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(3): 493-502. |
| [14] | Chen Senlin, Zhu Zhou, Wan Qianbing. Application of Janus micro/nanoparticles in biomedicine [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(28): 6101-6109. |
| [15] | Wu Yonghao, Zhu Shuaiqi, Li Yuqiao, Zhang Chenfei, Xia Weiwei, Zhu Zhenqi, Wang Kaifeng. Correction effect of local kyphosis of the spine after percutaneous kyphoplasty in super-aging patients with vertebral compression fractures [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(27): 5854-5861. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||