Chinese Journal of Tissue Engineering Research ›› 2025, Vol. 29 ›› Issue (13): 2812-2821.doi: 10.12307/2025.034
Previous Articles Next Articles
Applications and advances of single-cell transcriptome sequencing technology in osteoporosis research
Sun Chengtao1, 2 , Sun Guangjiang3 , Qi Xiaonan3 , Cheng Ming4 , Yao Xiaosheng3
- 1Liaoning University of Traditional Chinese Medicine, Shenyang 110847, Liaoning Province, China; 2First Department of Orthopedics and Traumatology, Second Affiliated Hospital of Liaoning University of Traditional Chinese Medicine, Shenyang 110034, Liaoning Province, China; 3First Department of Orthopedics and Traumatology, Affiliated Hospital of Liaoning University of Traditional Chinese Medicine, Shenyang 110032, Liaoning Province, China; 4Fourth People’s Hospital of Shenyang, China Medical University, Shenyang 110031, Liaoning Province, China
-
Received:2024-01-02Accepted:2024-02-24Online:2025-05-08Published:2024-09-12 -
Contact:Yao Xiaosheng, PhD, Doctoral supervisor, Chief physician, First Department of Orthopedics and Traumatology, Affiliated Hospital of Liaoning University of Traditional Chinese Medicine, Shenyang 110032, Liaoning Province, China -
About author:Sun Chengtao, Doctoral candidate, Attending physician, Liaoning University of Traditional Chinese Medicine, Shenyang 110847, Liaoning Province, China; First Department of Orthopedics and Traumatology, Second Affiliated Hospital of Liaoning University of Traditional Chinese Medicine, Shenyang 110034, Liaoning Province, China -
Supported by:National Natural Science Foundation of China for Young Scientists, No. 82305275 (to QXN); General Project of Natural Science Foundation of Liaoning Province, No. 2022-MS-284 (to SGJ); Science and Technology Plan Medical Industry Cross Joint Fund of Liaoning Province, No. 2022-YGJC-79 (to QXN)
CLC Number:
Cite this article
Sun Chengtao, Sun Guangjiang , Qi Xiaonan , Cheng Ming , Yao Xiaosheng. Applications and advances of single-cell transcriptome sequencing technology in osteoporosis research[J]. Chinese Journal of Tissue Engineering Research, 2025, 29(13): 2812-2821.
share this article
Add to citation manager EndNote|Reference Manager|ProCite|BibTeX|RefWorks

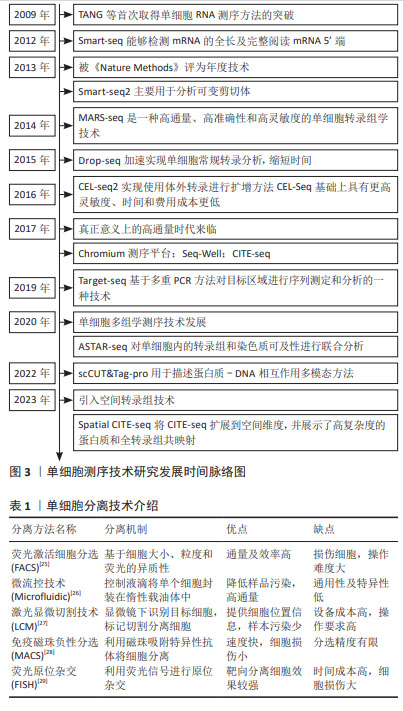
2.1 scRNA-seq技术的发展 scRNA-seq是在单个细胞水平对mRNA进行高通量测序的一项新技术,可以用来分析群体平均测量中被掩盖的转录组异质性。 标准的scRNA-seq步骤包括:样品获取、单个细胞分离、裂解、反转录、DNA扩增、构建文库、测序和分析数据[10]。自2009年TANG等[11]首次取得scRNA-seq的突破,各研究领域研究人员相继开展应用此项技术并不断改进。2012年smart-seq技术可以完整阅读mRNA的5’端[12],在2013年被《Nature Methods》评为年度技术。自此以后scRNA-seq技术飞速发展,在减少测序成本、缩短时间及开发新技术上投入大量人力、物力并取得一定成果[13-16],但直到2017年,才真正意义上实现了高通量测序,使该项技术趋于成熟[17-20]。2020年以来,已经向单细胞基因组、蛋白组和代谢组等多组学方向发展,并融合诸如空间转录组等技术,将单细胞测序技术发展推向高潮,从不同角度揭示细胞的有效信息[21-24],具体时间脉络见图3。单细胞的研究依赖于将数千到数百万个细胞分离获得每个细胞中的转录组数据,这依赖于细胞分离技术的实现,其中单细胞转录组测序中关键一步就是单个细胞分离,从最初的人工挑选到低通量分离再到如今的高通量靶向分离技术[25-29],助力单细胞转录组测序技术高速发展。具体单细胞分离技术见表1。"

2.2 骨髓间充质干细胞的异质性 骨髓间充质干细胞在骨稳态中具有核心作用,但其分化的异质性特征、分化途径及潜在机制尚未明确。近些年单细胞转录组技术的发展为此提供了技术支持,并有学者探讨了此方面问题。骨髓间充质干细胞是具有成纤维细胞集落形成单位和多向分化能力的非造血骨髓基质细胞,具有分化为成骨/骨细胞和软骨细胞的能力[30],其异质性和可塑性取决于所处的微环境。骨髓间充质干细胞大部分位于血窦或远端动脉周围区域,至少包含两个亚群:骨骼干细胞群和基质细胞群[31-32]。近些年,人原代骨髓间充质干细胞标志物鉴定方面取得了长足的进展,已经鉴定出C-X-C基序趋化因子12(C-X-C motif chemokine 12,CXCL12)、瘦素受体(leptin receptor,LepR)及干细胞因子(stem cell factor,SCF)等[33-34], 其中CXCL12+LepR+网状细胞被定义为总骨髓间充质干细胞。TIKHONOVA等[35]完成了稳定状态下骨髓微环境的单细胞转录组分析,鉴定出2个内皮细胞亚群、4个血管周围细胞亚群及3个骨谱系细胞亚群,并且通过成纤维细胞集落形成单位(fibroblastic colony-forming units,CFU-F)发现,LEPR+亚群具有多向分化潜能和骨骼干细胞活性。 骨髓间充质干细胞标志物的鉴定取得进展的同时,更加深入精准鉴定处于不同生命阶段骨骼干细胞亚群变化也有新的发现。有研究表明骨骼干细胞的谱系分化潜能随时间逐渐发生转变,衰老的骨骼干细胞分化为成骨细胞和脂肪细胞比例倒置是导致骨骼退化的根本原 因[36-37]。有研究团队利用scRNA-seq技术探究中年小鼠(10-14月龄)参与骨骼分化的进展因素,共鉴定出2个基质/骨谱系亚群,有22个基因簇表达LepR+骨骼干细胞的特征基因,包括LepR,CXCL12和SCF在内[38]。进一步基因集富集分析(Gene Set Enrichment Analysis,GSEA)发现Notch信号通路在基质/骨谱细胞中随着年龄的增长而显著增加,同时发现转录因子早期B细胞因子3(EBF3)在Notch信号缺陷的骨骼干细胞中下调,且EBF3位于Notch信号通路的下游,表明EBF3是一个很有前途的治疗靶点,可以预防或挽救老年人骨骼完整性的年龄相关性丧失。 ZHU等[39]取股骨转子间骨折患者第2代骨髓间充质干细胞进行单细胞转录组测序,同时鉴定出3个具有不同功能的亚群,可以证明在骨髓微环境内细胞分化并非同步。ZHANG等[40]对2例样本(骨质疏松症患者和骨关节炎患者)进行单细胞亚群研究,共鉴定出12个细胞亚群,发现其中6个亚群为CD45+骨髓间充质干细胞,6个亚群为CD45- 骨髓间充质干细胞,筛选出7个差异表达的基因(HSD17B2,ACHE,CCR4,F9,ADRA1D,MC5R和GRM2),基于此作者构建了能够准确预测骨质疏松症患者的7基因诊断模型和治疗剂,但作者并未给出治疗剂的具体名称。可以确定骨髓间充质干细胞在不同微环境下具有不同亚群,那么这些亚群的功能如何?是否可以直接分离出研究者们所需要的功能亚群?XIE等[41]利用不同供者鉴定骨髓间充质干细胞的亚群特征,均鉴定到干性亚群、功能亚群和增殖亚群3个亚群,并且确定了干性亚群可以分别分化为功能亚群或增殖亚群,其中功能亚群具有更强的免疫调节和成骨分化能力,但是不同供者之间存在个体差异,这可能导致不同临床应用效果。但尚未有涉及大样本供者的相关研究。研究表明随着骨髓间充质干细胞传代增加,细胞逐渐衰老,表现出各种能力的下降,包括分化和迁移能力,标志基因主要富集于氧化应激、内质网应激和自噬等与衰老相关的代谢过程[42-43]。现在研究者们可以明确不同供体同种细胞在不同条件下具有不同亚群分布且有着不同的转录特征,执行不同的功能,其生物标志物鉴定及治疗方面现阶段尚处于起步阶段,未发现有系统的、明确的、公认的亚群分类及新发现潜在生物标志物和治疗方面的更新。 目前已经有多项研究将scRNA-seq应用于不同来源的骨髓基质成分或针对骨髓间充质干细胞。通过对股骨头组织单细胞分析[44],共鉴定到间充质干细胞(骨髓间充质干细胞)、血管周细胞和血管内皮细胞。其中骨髓间充质干细胞随着细胞衰老偏向分化为脂肪细胞特性,这与既往认知中由于增龄,骨髓腔中形成更多脂肪组织相一致,但是当抗脂肪形成时这种成脂分化是可逆的。除此发现外,研究者们还鉴定了3种新的生物标志物:早期生长反应因子(early growth response,EGR1)、软脂酰化磷蛋白(sprouty,SPRY1)和RNA结合锌指蛋白36(zinc finger protein 36,ZFP36),而且这3种标志物均可能处于关键通路的中心位置,这些关键标志物可调节骨髓间充质干细胞成脂分化的命运,以改善骨骼强度。强调了骨髓间充质干细胞在衰老环境中的表型和功能状态,有助于寻找解决由于衰老而导致的骨代谢变化。已知骨髓间充质干细胞的基因表达具有高度异质性,但缺乏系统的剖析其细胞异质性的程度及在维持骨稳态中的作用。与上文作者研究思路相同,有学者对新鲜分离人的CD271+骨髓间充质干细胞单细胞进行RNA测序并鉴定到骨髓间充质干细胞中与多种分化轨迹相对应的亚型[45],与此不同的是,通过进一步考察各亚群中高表达的基因、生物学过程和信号通路之间的关系,提示成骨细胞前体细胞具有诱发血管发育的能力,软骨细胞前体细胞具有成肌潜能。对表达谱的进一步分析揭示了一个连续的基因表达模式,表明成骨只发生在骨髓间充质干细胞发育的早期阶段,而成脂和静止期细胞在终末阶段占据主导地位。这些发现也证明衰老是骨髓间充质干细胞成骨和成脂分化之间平衡的重要因素。 由于人类样本的缺乏,模式动物研究也较深入,有益于深入探讨骨质疏松症的具体分子机制。失用性骨质疏松症是由于机械刺激减少导致的骨量减少和骨脆性增加,探讨力学加载如何调控骨重建非常有意义。多种力学刺激可以调节骨髓间充质干细胞的增殖和分化[46]。HU等[47]通过scRNA-seq分析后肢去负荷4周小鼠骨髓微环境的变化,共鉴定了4组免疫细胞(粒细胞、嗜碱性粒细胞、淋巴细胞和单核-巨噬细胞)、2组成骨谱系细胞(骨髓间充质干细胞和成骨细胞)、破骨细胞、成脂谱系细胞和造血谱系细胞,与正常组(9.6%)相比,微重力组小鼠(4.1%)骨髓间充质干细胞比例显著降低,同时,Piezo1在正常组中高表达,4周后在微重力组下调明显。Piezo1蛋白是机械敏感性离子通道,它可以将机械刺激(包括静压力、剪切力和膜拉伸)转化为生物电信号,从而调节细胞的生物学功能[48], 提示骨髓间充质干细胞数量减少和Piezo1表达下降与微重力导致的失用性骨质疏松密切相关。在机制研究上,Piezo1激活β-catenin及其靶基因ATF4 促进Gli1+骨髓间充质干细胞的增殖和成骨分化。总之,骨髓间充质干细胞中Piezo1功能受损导致骨形成不足,特别是异常力学信号刺激引起的骨形成不足是骨质疏松症的潜在治疗靶点。 骨密度作为骨质疏松症的诊断指标,是一个受遗传效应影响较大的复杂性状,因此,遗传效应对峰值骨量和年龄相关的骨丢失一直是该领域的研究热点。来自大型个体队列研究已经确定了可能调节骨密度的候选基因,其变异可能参与骨质疏松症的发生[49]。一旦确定某个或者几个候选基因在骨代谢中的潜在作用,后续转化研究可以将这些遗传因素转化为有效治疗的靶点,例如罗莫珠单抗和地诺单抗[50-51]。然而很多技术阻碍人们了解候选基因的运作机制及功能验证,scRNA-seq技术的出现为探究候选基因打开了新思路。有研究首先比较同年龄小鼠骨组织中候选基因表达差异及亚群分布情况发现:首先,相比于骨髓,候选基因(骨形态发生蛋白4,Tmem119,col1α2)主要在骨组织中上调,在单细胞水平上,这些候选基因主要在骨髓间充质干细胞中富集[52];其次,富集在软骨细胞,骨谱系细胞和矿化骨细胞;此外,免疫细胞亚群中未显示候选基因的富集。这些结果均表明,候选基因在骨髓间充质干细胞谱系中发挥作用,表达最高的候选基因是Sparc,它负责编码细胞外基质蛋白——骨连接蛋白,对骨量维持至关重要。进一步功能验证明确了性别决定区Y框蛋白9(sex determining region Y box protein 9,Sox9)是骨谱系细胞群中的关键调控转录因子,证实Sox9对于成骨细胞分泌的Sparc蛋白表达的促进作用。该结果可能有助于弥合骨质疏松症遗传和转化研究之间的鸿沟。 2.3 成骨细胞的异质性 成骨细胞是骨重塑的主要细胞,占骨骼细胞群含量的4%-6%,其主要功能有合成蛋白、分泌基质及形成新骨[53]。在新骨形成中会有一半以上的成骨细胞发生凋亡,而剩余的则被包裹在骨基质中成为骨细胞或作为骨衬里细胞[54]。成骨细胞分化阶段主要有3个时期:增殖期、细胞外基质产生期、细胞外基质矿化期[55]。这些处于不同分化阶段的前成骨细胞内转录组的表达一定是不同的,scRNA-seq技术可以在看似均一的细胞群体中发现新的细胞间基因表达水平的异质性,可以解答一些重要的生物学问题。 有学者利用单细胞测序技术定义到2种成骨细胞亚群(OLC-1和OLC-2)均可以表达骨钙素(bone gla protein,BGLAP)[56],但是差异表达Runx相关转录因子2(runt-related transcription factor,Runx2)控制骨髓间充质干细胞向成骨细胞方向分化,形成不同功能的细胞亚群。基于此,对成骨细胞异质性得到了初步的认识,但是不同亚群之间分化轨迹如何呢?ZHANG等[57]的单细胞分析揭示了3种成骨细胞亚群和3个新的独特的细胞亚群,在谱系分支中,所有细胞都保留在一个细胞谱系轨迹中,成骨细胞祖细胞主要富集在分化轨迹的初始阶段,前成骨细胞主要富集在分化轨迹早期阶段,未确定的成骨细胞亚群集中在分化轨迹的中期阶段,成熟的成骨细胞主要富集在谱系轨迹的末期阶段。细胞转录谱的连续性验证发现,成骨细胞特异性标志基因的表达模式与骨髓间充质干细胞分化时间轨迹是吻合的。差异基因进行富集主要富集到“调控细胞周期相变、组蛋白修饰、组蛋白去乙酰化”等条目;代谢通路分析显示2条代谢通路高度富集,包括“表皮生长因子受体ErbB信号通路”和“表皮生长因子受体EGFR酪氨酸激酶抑制剂通路”,这些发现有助于更好地理解成骨细胞的异质性、分化轨迹和代谢过程。 特立帕肽(teriparatide)是第一个用于治疗骨质疏松症的合成代谢类药物,通过刺激成骨细胞活性,减少临床上新椎体压缩骨折的风险并增加腰椎的骨密度[58]。但成骨细胞作用靶点尚不完全明确,单细胞组学技术的出现为此提供了技术支持。研究人员首先通过scRNA-seq技术揭示了小鼠出生后成骨细胞的主要来源:Gli1+干骺端的间充质干细胞的异质性[59],并且揭示了一个软骨细胞样骨祖细胞亚群,在所有细胞亚群中,骨祖细胞亚群中Hedgehog(Hh)靶基因和胰岛素样生长因子1受体。经过特立帕肽处理后进一步上调胰岛素样生长因子1受体,抑制Hh信号或从Gli1+干骺端的间充质干细胞中删除胰岛素样生长因子1受体可减弱特立帕肽的增殖和成骨作用。所以研究人员认为:骨祖细胞亚群是特立帕肽的靶细胞,Gli1+干骺端的间充质干细胞中的Hh和胰岛素样生长因子相关信号介导了特立帕肽的合成代谢效应。为特立帕肽提供了更为准确的靶细胞和介导合成代谢途径。 2.4 骨细胞的异质性 骨细胞起源于终末分化的成骨细胞,随着时间的进展,成骨细胞嵌入骨基质深处并矿化,骨细胞之间通过缝隙连接和液体流动相互连接和作用。骨骼中骨细胞占比达到90% [60],最近,新的证据强调了骨细胞在骨重建中的关键作用[61]。HANAI等[62]对C57BL/6J雄性小鼠股骨组织进行单细胞测序表征骨细胞异质性,验证了牙本质基质蛋白(dental matrix protein 1,Dmp1),细胞外基质磷酸化糖蛋白(matrix extracellular phosphoglycoprotein,MEPE)和X染色体上与内肽酶同源的磷调节基因(posphate regulating gene with homology to endopeotidases on X,phex)作为骨细胞的标记蛋白,定义骨细胞。在注射骨化三醇的骨细胞亚群中观察到表达成纤维细胞生长因子23的细胞,并证实表达成纤维细胞活性因子23的骨细胞表现出更高的Dmp1,MEPE和phex蛋白表达水平,已知1,25 -二羟维生素D3可以诱导骨细胞中成纤维细胞活性因子23的表达,并介导对机械负荷和激素刺激的适应性反应以维持骨稳态。 骨细胞在骨微环境中具有独特的多个类似神经元树突状的突起穿行于骨中的腔隙小管系统,树突状突起作为物质基础允许骨细胞之间和骨细胞与其他细胞之间建立“神经网络”进行信息交流[63]。骨细胞树突网络的缺失可能在衰老环境下导致骨骼脆性增加[64]。研究人员在成熟的成骨细胞和骨细胞中敲除Osterix并观察骨骼表型的变化,观察到皮质空隙、骨细胞凋亡增加和骨细胞树突严重缺陷[65]。随后对两种细胞进行单细胞测序,共捕获了8个不同的“Ob/Ocy骨谱系亚群”从增殖的前成骨细胞到成熟的表达Sost的骨细胞。骨硬化蛋白(Sclerostin,Sost)是由成熟骨细胞分泌调节骨矿化的重要调节剂之一[66]。拟时序分析证实存在两种潜在的分化途径,同时发现主要富集到“神经元”相关术语如细胞投射组织和神经元分化;突出了骨细胞和神经元连接之间的共同特征,并显示出骨细胞生成的步骤,可以为骨质疏症患者提高骨强度提供治疗办法。骨细胞分化如图4,成骨系亚群鉴定研究进展见表2。"

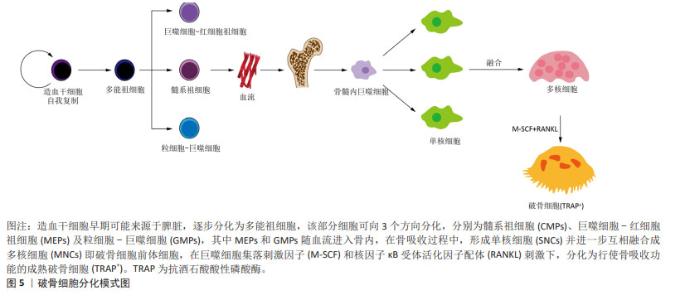
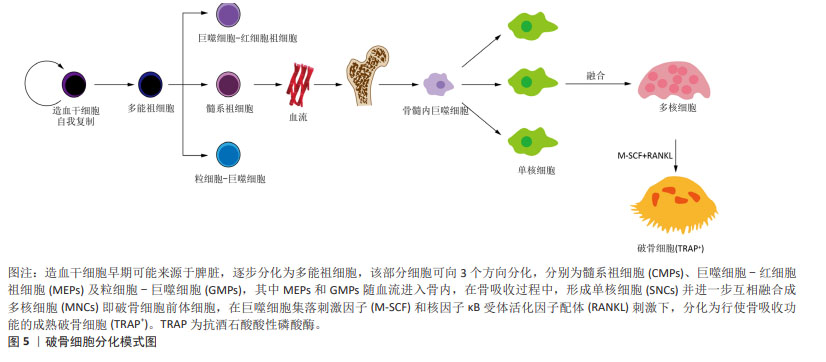
2.5 破骨细胞的异质性 骨吸收不仅影响骨量,而且对骨髓微环境稳态也至关重要。破骨细胞是唯一的骨吸收细胞,过度生成的破骨细胞参与包括骨质疏松在内的病理状态 [67-68]。因此,对破骨细胞分化的详细分子表征对于理解骨骼和免疫系统的生理和病理都是至关重要的。 破骨细胞体外培养体系应用广泛,也为骨生物学提供了相当大贡献,但因为破骨细胞培养物中含有异质性的细胞群,只有一部分能够分化为成熟的破骨细胞,因此其应用具有一定的局限性。研究人员对小鼠破骨细胞体外培养体系进行3次不同时间点亚群鉴定揭示:亚群7第3天表现出破骨细胞标志基因[69],表明其为成熟的破骨细胞;相反,亚群1在第0天大量存在,第3天消失并且具有破骨细胞前体细胞的标志基因,以及破骨细胞生成的负调控因子,表明其由破骨细胞前体细胞组成。分化轨迹表明,亚群1逐步分化为亚群7,沿途经过亚群9,3,2和4,这一过程破骨细胞基因表达水平逐渐升高,而破骨细胞生成的负调控因子表达水平逐渐下调。文章提供了一份详细的破骨细胞分化轨迹图,完善和扩展了研究者们对破骨细胞生成的分子机制的理解。有学者利用同样方法分析识别人和小鼠破骨细胞形成过程物种间差异的关键因子发现:鸟苷三磷酸酶(GTPase)家族成员RAB38在两个物种间均高表达,RAB38与组蛋白修饰和转录调控动态变化有关[70],这一发现有助于确定骨质疏松症的治疗靶点。 YAHARA等[71]研究出生后破骨细胞的起源问题时发现,新生骨骼中的破骨细胞大部分来源于红髓系祖细胞(erythromyeloid progenitors,EMPs)来源的卵黄囊巨噬细胞,Cx3cr1+卵黄囊巨噬细胞的后代产生持久的破骨细胞前体,并在成人骨骼中提供破骨细胞。scRNA-seq表明Cx3cr1+卵黄囊巨噬细胞的后代独立于造血干细胞而Csf1r+破骨细胞前体不能产生持续的破骨细胞。进一步研究发现脾脏可以作为红髓系祖细胞来源的破骨细胞前体的来源,但是其在脾脏中建立的生物学机制仍不清楚。在骨发育进程中,破骨细胞前体在骨基质周围的间充质中迁移,它们分化为TRAP+细胞,并转变为成熟的破骨细胞,使基质变形,产生造血空间。上述结果为调节破骨细胞功能的最佳方法提供信息,并增加证据,也使研究者们更清楚地认识到红髓系祖细胞来源的破骨细胞是一个独特的前体群体,在骨稳态中具有特定的作用。 单核细胞属于单核吞噬细胞系统,起源于造血前体细胞[72],具有多种功能,包括支持组织稳态、介导宿主防御、启动炎症和分化为破骨细胞[73]。有研究以健康3月龄SD大鼠为研究对象,探索大鼠骨髓中主要谱系亚群分布中识别了19个聚类[74],通过CD43的表达揭示了单核细胞的2个亚群:CD34low和CD34hi,对亚群进行了KEGG和GO分析,发现KEGG和GO分析主要集中在破骨细胞分化、类风湿关节炎、吞噬体和溶酶体等与破骨细胞分化高度相关的通路,进一步的特异性表达特征包括Mitf,Spi1,Fos转录因子和Csf1r细胞因子在破骨细胞分化中起关节作用,并且在经典单核细胞(CD34low)中高表达。作者据此推测经典单核细胞是大鼠体内主要的破骨细胞前体。针对经典单核细胞向破骨细胞分化过程中差异表达的基因,帮助研究者们了解破骨细胞的起源问题,为骨代谢相关疾病(骨质疏松症)的早期诊断提供生物标志物。 破骨细胞多核化是破骨细胞成熟的标志,也是破骨细胞分化的最后一步[75]。已经明确的是巨噬细胞基于其来源具有异质性,并且细胞分化非同步进行,破骨前体细胞融合并发育成多核巨噬细胞,但仍有很多细胞保持单核细胞状态。探究非同步分化细胞之间的转录组差异具有重要意义。有研究利用scRNA-seq技术对单个有核(single nucleated cells,SNCs)和多核细胞(multinucleated cells,MNCs)进行RNA测序发现,单核细胞比多核细胞更活跃、更有运动性、它们附着于骨质上进行骨吸收[76];并结合RT-PCR,TRAP染色和免疫荧光染色明确了单核细胞是破骨前体细胞即多核细胞。进一步进行骨髓源巨噬细胞与单核细胞、多核细胞之间基因表达模式比较发现:7个基因(Aifl,Calcrl,Gsta3,Ifit2,Marco,Tmem26及Ube3a)表达呈现显著差异,并采用CRISPR-Cas9基因组编辑工具生成缺乏每个基因的RAW264.7细胞联合蛋白检测评估它们的作用,最终表明在Calcr1缺陷的RAW264.7细胞中,细胞融合受阻,是细胞融合所必需的。文章对破骨细胞融合成熟过程中,提供了潜在的新的决定因素,突出了骨质疏疏松治疗的潜在靶点。对通过抑制破骨成熟融合治疗骨质疏松方案提供靶点。破骨细胞分化模式见图5。"

2.6 免疫细胞与骨髓血管内皮细胞的异质性 ARRON等[77]在2000年提出“骨免疫学”的概念将骨代谢和免疫系统两个学科联系起来,自此,两者通过各种生理和病理状态紧密联系在一起。自然杀伤细胞、调节性T细胞、γδ-T细胞和CD8+ T细胞也可以通过分泌细胞因子和趋化因子调节骨内免疫微环境,参与骨质疏松的发病[78]。总体来说,随着骨免疫学研究的深入,将炎症状态引入骨质疏松发病机制将为骨质疏松防治提供新的理论依据,扩大靶点筛查范围和有效药物研发。利用scRNA-seq 定义细胞类型并表征其功能,具有重要的推动作用。 ZHOU等[79]表征骨髓微环境特征时发现,只有静息或活化状态的肥大细胞存在差异分布,说明肥大细胞的潜在重要性,以肥大细胞为研究对象筛选出关键基因,其中细胞周期蛋白依赖性激酶抑制剂1A (cyclin-dependent kinase inhibitor 1A,CDKN1A)、细胞内黏附分子1(intracellular adhesion molecule 1,ICAM1)和核苷酸结合寡聚结构域样受体(nucleotide-binding oligomerization domain-like receptor family pyrin-contaiing 3,NLRP3)已被报道参与炎症反应影响骨质疏松症的发生[80-82],具有进一步挖掘其潜力。 骨免疫学从单细胞层面可以理解为免疫细胞与骨髓内各种细胞间的细胞通讯,既往缺乏准确和高分辨率表征骨组织细胞和免疫细胞间的关系和相互作用。近些年单细胞组学测序技术快速进步,XU等[83]揭示了人椎骨组织中破骨细胞的分化轨迹及T细胞和B细胞积极参与调节骨稳态,通过识别配体-受体发现,免疫细胞和破骨细胞具有双向调节的特性,细胞通讯发现破骨细胞作为配体或受体与CD8- TEM细胞、初始B细胞存在配对关系,证实骨细胞和免疫细胞间的双向通讯的分子靶标和潜在功能。破骨细胞可能通过CD160-TNFRSF14配体-受体相互作用进行细胞间通讯,这是一种正反馈调节机制。根据细胞间通讯的结果,通过免疫细胞介导抑制破骨细胞生成将是重要课题研究方向,同时更有助于确定新的治疗靶点,也为药物发展铺平道路。 另有学者探索成骨细胞谱系细胞与骨微环境免疫细胞的体内骨免疫通讯,表明在骨微环境中,中性粒细胞/单核细胞及浆样树突状细胞广泛参与成骨过程[84]。例如:RETN-CAP1 (RESISTIN通路)是中性粒/单核细胞与成骨细胞系相互作用表达最高的通路;AREG-EGFR (EGF通路)是浆样树突状细胞与成骨细胞系之间表达量最高的通路。进一步研究提示核心基因之间的强相互作用表明它们在维持老年男性高骨密度方面具有独特的功能。在绝经后女性受试者中,核心基因可以预测低/高骨密度组,进一步强调了它们在该人群中的潜在功能。 骨髓血管内皮细胞在成血管-成骨偶联的时空关系中起核心作用[85]。虽然已经利用单细胞RNA测序揭示了骨髓细胞分类学,增加了研究者们对骨代谢和造血细胞间相互作用的理解[56]。但是,关于骨髓血管内皮细胞的时空定位及其功能了解是有限的。有研究从小鼠股骨中分离的CD31+CD45-骨髓细胞进行了分析,定义了11个亚群[86],H型和L型内皮细胞在组织学上分别表现为主要存在干骺端和骨干的特化毛细血管群。作者同时发现了一种未被识别的毛细血管亚群,称为S型内皮细胞,它完全起源于骨骺次级骨化中心,甚至比H型血管更具有可塑性,对骨骺成骨具有强大的贡献,此发现提高了对骨和血管偶联的认识,尤其是在长骨的软骨内成骨方面。 破骨细胞系亚群鉴定研究进展见表3。 目前的研究表明骨髓血管内皮细胞是骨骼中骨形成和骨吸收的活跃参与者,骨髓内皮细胞与骨髓其他细胞群相互的串扰可能为骨代谢提供新的切入点[87]。例如,骨髓血管内皮细胞与破骨细胞间的细胞通讯显著,成纤维细胞生长因子2对骨骼的修复作用有望成为新的靶点[83]。 中医药防治骨质疏松症有其特色之处,中药复方成分复杂、作用靶点众多,识别具体作用机制困难,单细胞转录组能够识别特定的细胞亚群、鉴定疾病相关核心靶点,两者结合可以从细胞层面理解中药复方的作用机制。有研究利用液质联用技术和单细胞转录组分析研究参苓白术散的作用机制,发现参苓白术散通过调控免疫细胞与骨细胞的相互作用防治骨质疏松症[88]。将单细胞组学技术体系纳入中医药研究的框架,建立相应基于单细胞组学技术的中医药现代性研究策略,将研究尺度从宏观投射到微观细胞层面,可以将复杂中药成分作用于单细胞亚群,探究其对亚群转录组的影响,进而发掘潜在的中医药价值。 2.7 scRNA-seq在骨质疏松症应用的挑战 鉴于细胞RNA作为信使,形成调节及管理细胞功能的主要执行者——蛋白,转录组是维持细胞身份和任何生物体生存的重要组成部 分[89]。因此,全基因转录组分析解决了几十年尚未解答的问题。但是每种技术都不是完美的,具有其自身的局限性。所有单细胞测序方法主要限制之一可能均是基于DNA。首先,目前的技术水平无法直接从单细胞中测序RNA,意味着必须将RNA反转录成cDNA并进行扩增,这就带来3个挑战:一方面如何减少反转录过程中RNA的损失;另一方面,提供足够的DNA用于测序的同时排除基因组DNA对cDNA的影响。对于单细胞分离方面,单一的阳性选择方案并不理想,因为分离的细胞由多种类型的细胞组成,如果主要目的是分离纯度较高的细胞则应考虑使用正选择和负选择相结合的方式,但是细胞数量上不足问题也凸显。由于高度矿化的组织中含有低数量的细胞,所以从骨组织获得高质量细胞的技术难度很高。最后,对于样品选择的问题也影响实验的结果,通过分析检索所得文献,单纯的骨质疏症实验样品很难获得,多合并骨骼其他疾病(关节炎),势必会影响实验分析结果。另外,模式生物的应用可以提供更为广泛深入的尝试。 在未来需要不断完善scRNA-seq,并需要与其他技术结合,例如空间转录组、单细胞多组学,谱系追踪及活体成像技术等。近年来,空间转录组学迅速发展,可以弥补单细胞转录组缺失空间信息这一劣势,二者结合更加还原组织原貌,且空间转录组不需要分离细胞,直接使用完整的组织切片进行分析,更有利于探究矿化的骨组织;还可以与单细胞组学(基因组、表观组和蛋白组)进行整合,并得到一个关于单细胞的综合理解;上述技术的结合均可以促使骨质疏症领域重新审视骨组织细胞分类学,围绕骨组织细胞的形成、寿命和归宿问题展开更大范围的研究,为进一步开发潜在诊断生物标志物和治疗措施提供新机遇和活力。"

| [1] 中华医学会骨质疏松和骨矿盐疾病分会.原发性骨质疏松症诊疗指南(2022)[J].中华骨质疏松和骨矿盐疾病杂志,2022,15(6):573-611. [2] ARTHUR A, GRONTHOS S. Clinical application of bone marrow mesenchymal stem/stromal cells to repair skeletal tissue. Int J Mol Sci. 2020;21(24):9759. [3] OLSEN TK, BARYAWNO N. Introduction to single-cell RNA sequencing. Curr Protoc Mol Biol. 2018;122(1):e57. [4] SLOVIN S, CARISSIMO A, PANARIELLO F, et al. Single-cell RNA sequencing analysis: a step-by-step overview. Methods Mol Biol. 2021;2284:343-365. [5] ANAPARTHY N, HO YJ, MARTELOTTO L. Single-cell applications of next-generation sequencing. Cold Spring Harb Perspect Med. 2019;9:a026898. [6] NEFTEL C, LAFFY J, FILBIN MG, et al. An integrative model of cellular states, plasticity, and genetics for glioblastoma. Cell. 2019;178(4):835-849. [7] BONORA G, RAMANI V, SINGH R, et al. Single-cell landscape of nuclear configuration and gene expression during stem cell differentiation and X inactivation. Genome Biol. 2021;22(1):279. [8] TUCKER NR, CHAFFIN M, FLEMING SJ, et al. Transcriptional and cellular diversity of the human heart. Circulation. 2020;142(5):466-482. [9] SAULER M, MCDONOUGH JE, ADAMS TS, et al. Characterization of the COPD alveolar niche using single-cell RNA sequencing. Nat Commun. 2022;13(1):494. [10] YEO AT, RAWAL S, DELCUZE B, et al. Single-cell RNA sequencing reveals evolution of immune landscape during glioblastoma progression. Nat Immunol. 2022;23:971-984. [11] TANG F, BARBACIORU C, WANG Y, et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nat Methods. 2009;6(5):377-382. [12] PICELLI S, BJÖRKLUND ÅK, FARIDANI OR, et al. Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nat Methods. 2013;10(11): 1096-1098. [13] SEIRUP M, CHU LF, SENGUPTA S, et al. Reproducibility across single-cell RNA-seq protocols for spatial ordering analysis. PLoS One. 2020;15(9):e0239711. [14] JAITIN DA, KENIGSBERG E, KEREN-SHAUL H, et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science. 2014;343(6172):776-779. [15] MACOSKO EZ, BASU A, SATIJA R, et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 2015; 161(5):1202-1214. [16] HASHIMSHONY T, SENDEROVICH N, AVITAL G, et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 2016;17:77. [17] ZHENG GX, TERRY JM, BELGRADER P, et al. Massively parallel digital transcriptional profiling of single cells. Nat Commun. 2017;8:14049. [18] GIERAHN TM, WADSWORTH MH 2ND, HUGHES TK, et al. Seq-Well: portable, low-cost RNA sequencing of single cells at high throughput. Nat Methods. 2017;14(4):395-398. [19] STOECKIUS M, HAFEMEISTER C, STEPHENSON W, et al. Simultaneous epitope and transcriptome measurement in single cells. Nat Methods. 2017;14(9):865-868. [20] RODRIGUEZ-MEIRA A, BUCK G, CLARK SA, et al. Unravelling intratumoral heterogeneity through high-sensitivity single-cell mutational analysis and parallel RNA sequencing. Mol Cell. 2019;73(6):1292-1305.e8. [21] XING QR, FARRAN CAE, ZENG YY, et al. Parallel bimodal single-cell sequencing of transcriptome and chromatin accessibility. Genome Res. 2020;30(7):1027-1039. [22] ZHU C, ZHANG Y, LI YE, et al. Joint profiling of histone modifications and transcriptome in single cells from mouse brain. Nat Methods. 2021;18(3): 283-292. [23] ZHANG B, SRIVASTAVA A, MIMITOU E, et al. Characterizing cellular heterogeneity in chromatin state with scCUT&Tag-pro. Nat Biotechnol. 2022;40(8):1220-1230. [24] LIU Y, DISTASIO M, SU G, et al. High-plex protein and whole transcriptome co-mapping at cellular resolution with spatial CITE-seq. Nat Biotechnol. 2023; 41(10):1405-1409. [25] KUROKI Y, AGATA K. Isolation of planarian viable cells using fluorescence-activated cell sorting for advancing single-cell transcriptome analysis. Genes Cells. 2023;28(11):800-810. [26] ZHOU WM, YAN YY, GUO QR, et al. Microfluidics applications for high-throughput single cell sequencing. J Nanobiotechnology. 2021;19(1):312. [27] DATTA S, MALHOTRA L, DICKERSON R, et al. Laser capture microdissection: big data from small samples. Histol Histopathol. 2015;30(11):1255-1269. [28] WELZEL G, SEITZ D, SCHUSTER S. Magnetic-activated cell sorting (MACS) can be used as a large-scale method for establishing zebrafish neuronal cell cultures. Sci Rep. 2015;5:7959. [29] AMANN RI, KRUMHOLZ L, STAHL DA. Fluorescent-oligonucleotide probing of whole cells for determinative, phylogenetic, and environmental studies in microbiology. J Bacteriol. 1990;172(2):762-770. [30] DEMONTIERO O, VIDAL C, DUQUE G. Aging and bone loss: new insights for the clinician. Ther Adv Musculoskelet Dis. 2012;4(2):61-76. [31] WANG Z, LI X, YANG J, et al. Single-cell RNA sequencing deconvolutes the in vivo heterogeneity of human bone marrow-derived mesenchymal stem cells. Int J Biol Sci. 2021;17(15):4192-4206. [32] SACCHETTI B, FUNARI A, MICHIENZI S, et al. Self-renewing osteoprogenitors in bone marrow sinusoids can organize a hematopoietic microenvironment. Cell. 2007;131:324-336. [33] LI H, GHAZANFARI R, ZACHARAKI D, et al. Low/negative expression of PDGFR-α identifies the candidate primary mesenchymal stromal cells in adult human bone marrow. Stem Cell Reports. 2014;3(6):965-974. [34] MATSUSHITA Y, NAGATA M, KOZLOFF KM, et al. A Wnt-mediated transformation of the bone marrow stromal cell identity orchestrates skeletal regeneration. Nat Commun. 2020;11(1):332. [35] TIKHONOVA AN, DOLGALEV I, HU H, et al. The bone marrow microenvironment at single-cell resolution. Nature. 2019;569:222-228. [36] JOSEPHSON AM, BRADASCHIA-CORREA V, LEE S, et al. Age-related inflammation triggers skeletal stem/progenitor cell dysfunction. Proc Natl Acad Sci USA. 2019;116(14):6995-7004. [37] AMBROSI TH, SCIALDONE A, GRAJA A, et al. Adipocyte accumulation in the bone marrow during obesity and aging impairs stem cell-based hematopoietic and bone regeneration. Cell Stem Cell. 2017;20(6):771-784.e6. [38] REMARK LH, LECLERC K, RAMSUKH M, et al. Loss of Notch signaling in skeletal stem cells enhances bone formation with aging. Bone Res. 2023; 11(1):50. [39] ZHU D, GAO J, TANG C, et al. Single-cell RNA sequencing of bone marrow mesenchymal stem cells from the elderly people. Int J Stem Cells. 2022; 15(2):173-182. [40] ZHANG Z, ZHANG T, ZHOU L, et al. Identification of diagnostic genes and effective drugs associated with osteoporosis treatment by single-cell RNA-seq analysis and network pharmacology. Mediators Inflamm. 2022; 2022:6830635. [41] XIE Z, YU W, YE G, et al. Single-cell RNA sequencing analysis of human bone-marrow-derived mesenchymal stem cells and functional subpopulation identification. Exp Mol Med. 2022;54(4):483-492. [42] HONG SH, LEE MH, KOO MA, et al. Stem cell passage affects directional migration of stem cells in electrotaxis. Stem Cell Res. 2019;38:101475. [43] YANG YK, OGANDO CR, WANG SEE C, et al. Changes in phenotype and differentiation potential of human mesenchymal stem cells aging in vitro. Stem Cell Res Ther. 2018;9(1):131. [44] FIÉVET L, ESPAGNOLLE N, GEROVSKA D, et al. Single-cell RNA sequencing of human non-hematopoietic bone marrow cells reveals a unique set of inter-species conserved biomarkers for native mesenchymal stromal cells. Stem Cell Res Ther. 2023;14(1):229. [45] WANG Z, LI X, YANG J, et al. Single-cell RNA sequencing deconvolutes the in vivo heterogeneity of human bone marrow-derived mesenchymal stem cells. Int J Biol Sci. 2021;17(15):4192-4206. [46] 李莉莉,魏琦岩,王艳芳,等.FGF/FGFR信号调控成骨细胞分化的研究进展[J].中国生物工程杂志,2017,37(6):107-113. [47] HU Y, TIAN H, CHEN W, et al. The critical role of the Piezo1/β-catenin/ATF4 axis on the stemness of Gli1+ BMSCs during simulated microgravity-induced bone loss. Adv Sci (Weinh). 2023;10(32):e2303375. [48] 黄浩然,卫杨文祥,章家皓,等.Piezo1介导的机械应力刺激在抗骨质疏松中的作用[J].中国组织工程研究,2023,27(17):2716-2722 [49] ZHU X, BAI W, ZHENG H. Twelve years of GWAS discoveries for osteoporosis and related traits:advances, challenges and applications. Bone Res. 2021; 9(1):23. [50] WU D, LI L, WEN Z, et al. Romosozumab in osteoporosis: yesterday, today and tomorrow. J Transl Med. 2023;21(1):668. [51] REID IR, BILLINGTON EO. Drug therapy for osteoporosis in older adults. Lancet. 2022;399(10329):1080-1092. [52] DOOLITTLE ML, KHOSLA S, SAUL D. Single-cell integration of BMD GWAS results prioritize candidate genes influencing age-related bone loss. JBMR Plus. 2023;7(10):e10795. [53] CAPULLI M, PAONE R, RUCCI N. Osteoblast and osteocyte:games without frontiers. Arch Biochem Biophys. 2014;561:3-12. [54] LONG F. Building strong bones: molecular regulation of the osteoblast lineage. Nat Rev Mol Cell Biol. 2011;13(1):27-38. [55] JENSEN ED, GOPALAKRISHNAN R, WESTENDORF JJ. Regulation of gene expression in osteoblasts. Biofactors. 2010;36(1):25-32. [56] BARYAWNO N, PRZYBYLSKI D, KOWALCZYK MS, et al. A cellular taxonomy of the bone marrow stroma in homeostasis and leukemia. Cell. 2019;177(7): 1915-1932.e16. [57] ZHANG HX, CAO C, LI XH, et al. Imputation of human primary osteoblast single cell RNA-seq data identified three novel osteoblastic subtypes. Front Biosci (Landmark Ed). 2022;27(10):295. [58] KONTOGEORGOS G, KRANTZ E, TRIMPOU P, et al. Teriparatide treatment in severe osteoporosis-a controlled 10-year follow-up study. BMC Musculoskelet Disord. 2022;23(1):1011. [59] SHI Y, LIAO X, LONG JY, et al. Gli1+ progenitors mediate bone anabolic function of teriparatide via Hh and Igf signaling. Cell Rep. 2021;36(7):109542. [60] 高远,张长青,陶诗聪.骨细胞与骨衰老[J].国际骨科学杂志,2023, 44(1):1-4. [61] ROBLING AG. BONEWALD LF. The osteocyte:new insights. Annu Rev Physiol. 2020;82:485-506. [62] HANAI A, KAWABATA A, NAKAJIMA K, et al. Single-cell RNA sequencing identifies Fgf23-expressing osteocytes in response to 1, 25-dihydroxyvitamin D3 treatment. Front Physiol. 2023;14:1102751. [63] WANG JS, WEIN MN. Pathways controlling formation and maintenance of the osteocyte dendrite network. Curr Osteoporos Rep. 2022;20(6):493-504. [64] BONEWALD LF. The amazing osteocyte. J Bone Miner Res. 2011;26(2): 229-238. [65] TIEDE-LEWIS LM, DALLAS SL. Changes in the osteocyte lacunocanalicular network with aging. Bone. 2019;122:101-113. [66] FABRE S, FUNCK-BRENTANO T, COHEN-SOLAL M. Anti-sclerostin antibodies in osteoporosis and other bone diseases. J Clin Med. 2020;9(11):3439. [67] TSUKASAKI M, TAKAYANAGI H. Osteoimmunology:evolving concepts in bone-immune interactions in health and disease. Nat Rev Immunol. 2019;19(10):626-642. [68] OKAMOTO K, NAKASHIMA T, SHINOHARA M, et al. Osteoimmunology: the conceptual framework unifying the immune and skeletal systems. Physiol Rev. 2017;97(4):1295-1349. [69] TSUKASAKI M, HUYNH NC, OKAMOTO K, et al. Stepwise cell fate decision pathways during osteoclastogenesis at single-cell resolution. Nat Metab. 2020;2(12):1382-1390. [70] OMATA Y, OKADA H, UEBE S, et al. Interspecies single-cell RNA-Seq analysis reveals the novel trajectory of osteoclast differentiation and therapeutic Targets. JBMR Plus. 2022;6(7):e10631. [71] YAHARA Y, BARRIENTOS T, TANG YJ, et al. Erythromyeloid progenitors give rise to a population of osteoclasts that contribute to bone homeostasis and repair. Nat Cell Biol. 2020;22(1):49-59. [72] GINHOUX F, JUNG S. Monocytes and macrophages: developmental pathways and tissue homeostasis. Nat Rev Immunol. 2014;14(6):392-404. [73] UDAGAWA N, KOIDE M, NAKAMURA M, et al. Osteoclast differentiation by RANKL and OPG signaling pathways. J Bone Miner Metab. 2021;39(1):19-26. [74] JIRUI W, WENCHAO W, MIN T, et al. Single-cell RNA sequencing reveals classical monocytes are the major precursors of rat osteoclasts. Biocell. 2022;46(3):655-665. [75] KODAMA J, KAITO T. Osteoclast multinucleation: review of current literature. Int J Mol Sci. 2020;21(16):5685. [76] CHO E, CHEON S, DING M, et al. Identification of novel genes for cell fusion during osteoclast formation. Int J Mol Sci. 2022;23(12):6421. [77] ARRON JR, CHOI Y. Osteoimmunology-bone versus immune system. Nature. 2000;408(6812):535-536. [78] SRIVASTAVA RK, DAR HY, MISHRA PK. Immunoporosis: immunology of osteoporosis-role of T cells. Front Immunol. 2018;9:657. [79] ZHOU Z, HUANG Z, KHAN HM, et al. Identification of 12 hub genes associated to the pathogenesis of osteoporosis based on microarray and single-cell RNA sequencing data. Funct Integr Genomics. 2023;23(2):186. [80] GUO W, JIN P, LI R, et al. Dynamic network biomarker identifies cdkn1a-mediated bone mineralization in the triggering phase of osteoporosis. Exp Mol Med. 2023;55(1):81-94. [81] SUN Y, WANG X, CHEN G, et al. miRNA-187-5p Regulates osteoblastic differentiation of bone marrow mesenchymal stem cells in mice by targeting ICAM1. Biomed Res Int. 2020;2020:6139469. [82] GUARANÁ WL, LIMA CAD, BARBOSA AD, et al. Can polymorphisms in NLRP3 inflammasome complex be associated with postmenopausal osteoporosis severity? Genes (Basel). 2022;13(12):2271. [83] XU Y, HUANG S, LI Z, et al. Single-cell RNA landscape of osteoimmune microenvironment in osteoporotic vertebral compression fracture and Kümmell’s disease. Front Cell Dev Biol. 2023;11:1276098. [84] WANG S, GREENBAUM J, QIU C, et al. Single-cell RNA sequencing reveals in vivo osteoimmunology interactions between the immune and skeletal systems. Front Endocrinol (Lausanne). 2023;14:1107511. [85] ONO N, ONO W, MIZOGUCHI T, et al. Vasculature-associated cells expressing nestin in developing bones encompass early cells in the osteoblast and endothelial lineage. Dev Cell. 2014;29(3):330-339. [86] IGA T, KOBAYASHI H, KUSUMOTO D, et al. Spatial heterogeneity of bone marrow endothelial cells unveils a distinct subtype in the epiphysis. Nat Cell Biol. 2023;25(10):1415-1425. [87] TUCKERMANN J, ADAMS RH. The endothelium-bone axis in development, homeostasis and bone and joint disease. Nat Rev Rheumatol. 2021;17(10): 608-620. [88] 陈伟坚,姜涛,周宜,等.LC-MS联合单细胞测序分析探讨参苓白术散治疗原发性骨质疏松症的作用机制[J].中国骨质疏松杂志,2023,29(10): 1458-1465. [89] SALIBA AE, WESTERMANN AJ, GORSKI SA, et al. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 2014;42(14):8845-8860. |
| [1] | Han Haihui, Ran Lei, Meng Xiaohui, Xin Pengfei, Xiang Zheng, Bian Yanqin, Shi Qi, Xiao Lianbo. Targeting fibroblast growth factor receptor 1 signaling to improve bone destruction in rheumatoid arthritis [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(9): 1905-1912. |
| [2] | Zhao Jiyu, Wang Shaowei. Forkhead box transcription factor O1 signaling pathway in bone metabolism [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(9): 1923-1930. |
| [3] | Zhou Jinhai, Li Jiangwei, Wang Xuquan, Zhuang Ying, Zhao Ying, Yang Yuyong, Wang Jiajia, Yang Yang, Zhou Shilian. Three-dimensional finite element analysis of anterior femoral notching during total knee arthroplasty at different bone strengths [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(9): 1775-1782. |
| [4] | Wang Wentao, Hou Zhenyang, Wang Yijun, Xu Yaozeng. Apelin-13 alleviates systemic inflammatory bone loss by inhibiting macrophage M1 polarization [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(8): 1548-1555. |
| [5] | Chen Shuai, Jin Jie, Han Huawei, Tian Ningsheng, Li Zhiwei . Causal relationship between circulating inflammatory cytokines and bone mineral density based on two-sample Mendelian randomization [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(8): 1556-1564. |
| [6] | Zhu Hanmin, Wang Song, Xiao Wenlin, Zhang Wenjing, Zhou Xi, He Ye, Li Wei, . Mitophagy regulates bone metabolism [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(8): 1676-1683. |
| [7] | Zhao Jiacheng, Ren Shiqi, Zhu Qin, Liu Jiajia, Zhu Xiang, Yang Yang. Bioinformatics analysis of potential biomarkers for primary osteoporosis [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(8): 1741-1750. |
| [8] | Liu Qi, Li Linzhen, Li Yusheng, Jiao Hongzhuo, Yang Cheng, Zhang Juntao. Icariin-containing serum promotes chondrocyte proliferation and chondrogenic differentiation of stem cells in the co-culture system of three kinds of cells [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(7): 1371-1379. |
| [9] | Zhang Zhenyu, Liang Qiujian, Yang Jun, Wei Xiangyu, Jiang Jie, Huang Linke, Tan Zhen. Target of neohesperidin in treatment of osteoporosis and its effect on osteogenic differentiation of bone marrow mesenchymal stem cells [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(7): 1437-1447. |
| [10] | Yang Zhihang, Sun Zuyan, Huang Wenliang, Wan Yu, Chen Shida, Deng Jiang. Nerve growth factor promotes chondrogenic differentiation and inhibits hypertrophic differentiation of rabbit bone marrow mesenchymal stem cells [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(7): 1336-1342. |
| [11] | Li Yueyao, Zhang Min, Yang Jiaju. Cistanoside A mediates p38/MAPK pathway to inhibit osteoclast activity [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(6): 1144-1151. |
| [12] | Zheng Lin, Jin Wenjun, Luo Shanshan, Huang Rui, Wang Jie, Cheng Yuting, An Zheqing, Xiong Yue, Gong Zipeng, Liao Jian. Eucommia ulmoides promotes alveolar bone formation in ovariectomized rats [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(6): 1159-1167. |
| [13] |
Huang Xiaobin, Ge Jirong, Li Shengqiang, Xie Lihua, Huang Jingwen, He Yanyan, Xue Lipeng.
Mechanisms of different yin nourishing and kidney tonifying methods on osteoclastysis pathway in ovariectomized rats #br#
#br#
[J]. Chinese Journal of Tissue Engineering Research, 2025, 29(6): 1214-1219.
|
| [14] | Qian Kun, Li Ziqing, Sun Shui . Endoplasmic reticulum stress in the occurrence and development of common degenerative bone diseases [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(6): 1285-1295. |
| [15] | Lan Shuangli, Xiang Feifan, Deng Guanghui, Xiao Yukun, Yang Yunkang, Liang Jie. Naringin inhibits iron deposition and cell apoptosis in bone tissue of osteoporotic rats [J]. Chinese Journal of Tissue Engineering Research, 2025, 29(5): 888-898. |
| Viewed | ||||||
|
Full text |
|
|||||
|
Abstract |
|
|||||